技术分享:新型无DNA切割的dCas9-SSAP基因编辑系统
Tips:在“大片段DNA高效插入的基因编辑工具FiCAT”技术分享中,我们介绍了将Cas9与PB转座酶相结合开发的基因编辑工具FiCAT,可实现大片段DNA在基因组上的高效定点插入。本期将介绍dCas9与SSAP相结合开发的新型基因编辑工具dCas9-SSAP,通过无DNA切割的同源重组来介导精准基因编辑。
CRISPR-Cas9介导的精准基因编辑需要三个关键步骤:精准识别、精准切割和精准修复。在第二步精准切割中,由于CRISPR-Cas9的切割会形成DNA双链断裂(DSB),造成基因组上一定程度的不稳定,因此在临床应用中存有潜在风险。CRISPR-Cas9的基因编辑效率主要取决于第三步精准修复,是由细胞自行选择DNA损伤修复机制来完成的,主要包括同源修复(HDR)与非同源末端连接(NHEJ)修复等。细胞启用不同修复机制会产生不同编辑产物,HDR修复机制最为精准,在接头处两端不会引入任何突变,但是修复效率低[1]。
为进一步优化基因编辑系统,科学家们致力于寻找无需DNA切割就能精准修复的系统,即简化为精准识别、精准修复两步就能完成精准基因编辑,从而在提高基因编辑效率的同时,又能保证安全性。2022年2月,在Nature cell biology杂志上发表了一种新型无DNA切割的基因编辑工具dCas9-SSAP,将dCas9系统的精准识别能力与单链退火蛋白(SSAP)的同源重组能力相结合,实现DNA片段在基因组上的高效定点插入,具有广阔应用前景[2]。研究者首先将目标锁定到噬菌体同源重组系统中的多个SSAP,它们介导的同源重组不依赖于DNA断裂[3, 4]。dCas9系统具有与Cas9系统一样的精准识别能力,能高效定位到基因组的靶序列上,解开DNA双链形成R-茎环,且不对DNA进行切割[5-7]。因此,将SSAP与结合dCas9-sgRNA-MS2茎环结构的MCP蛋白相融合,即可利用dCas9系统招募SSAP到靶序列上,进而介导靶序列发生同源重组[8](图一a和c)。研究者选取了三个管家基因DYNLT1,HSP90AA1和ACTB来进行测试,在对应基因组中的终止密码前定点插入P2A-mKate元件,利用红色荧光蛋白mKate的表达来评价基因编辑效率(图一b和c)。通过在细胞水平比较LBet、RecT、gp2.5这三种噬菌体SSAP,发现RecT系统的mKate阳性细胞比例最高,即基因编辑效率最高(图一d和e),因此选择RecT的SSAP系统用于后续研究。
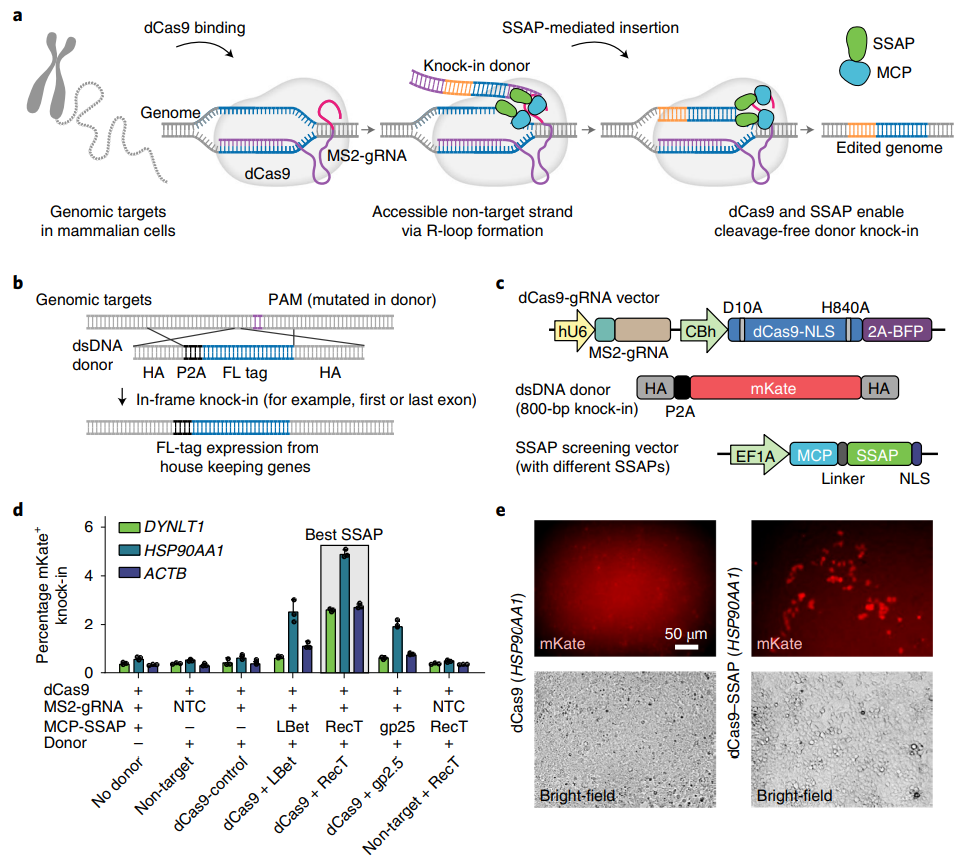
图一 dcas9-SSAP系统的开发[2]
接着,研究者将dCas9-SSAP系统与Cas9系统进行平行性比较。在获得的最终编辑产物中,由于Cas9系统涉及多个DNA损伤修复方式会产生多样化的编辑产物,而dCas9-SSAP系统的编辑产物主要为精准修复产物,准确率高达90-99%(图二a)。两个系统的精准编辑效率相当,dCas9-SSAP系统效率高一些(图二a和b);都能高效实现1.7 kb DNA的精准插入,但效率低于800 bp DNA的插入(图二b)。研究者在多个基因靶点上进行测试,dCas9-SSAP系统的最高精准编辑效率可接近20%(图二c)。值得注意的是,研究者并没有进一步研究dCas9-SSAP系统可插入的最大DNA长度。
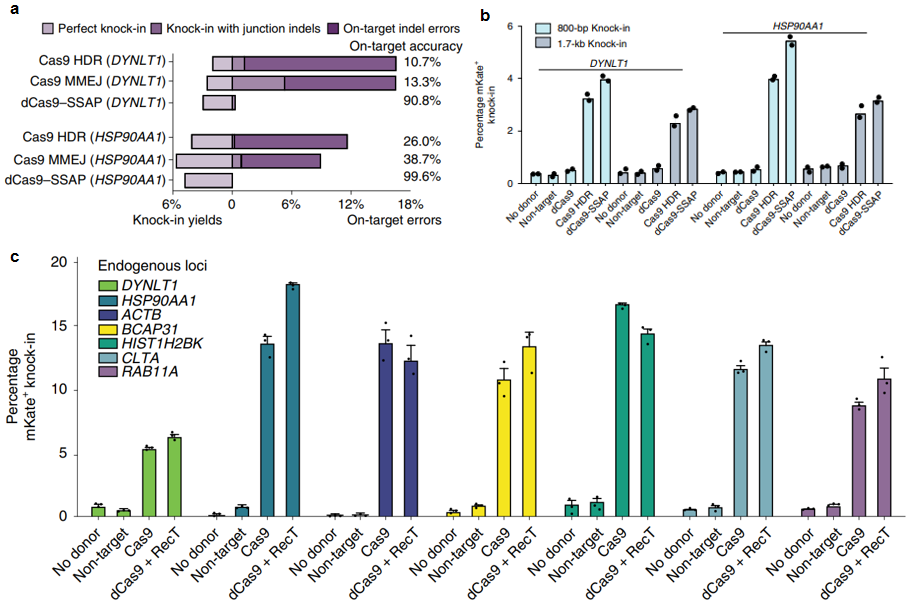
图二 dCas9-SSAP系统与Cas9系统的精准编辑效率比较[2]
研究者进一步证明dCas9-SSAP系统是否确实不依赖于DNA断裂后的HDR修复模式,通过添加HDR修复所需的Mre11-Rad50-Nbs1(MRN)复合体的抑制剂Mirin,或者RAD51的抑制剂B02和RI-1来抑制HDR修复通路(图三a)。结果表明,三种抑制剂的添加均不影响dCas9-SSAP系统的精准编辑效率,对照组Cas9系统的效率被显著降低(图三b),表明dCas9-SSAP系统区别于Cas9系统,并不依赖于DNA损伤修复机制。研究者采用胸腺嘧啶核苷双阻断法(DTB)将细胞同步化到G1-S期(图三c),发现同Cas9系统一致,DTB处理显著降低了dCas9-SSAP系统的精准编辑效率(图三d),表明dCas9-SSAP系统为细胞周期依赖,S期更有利于dCas9解开DNA双链。
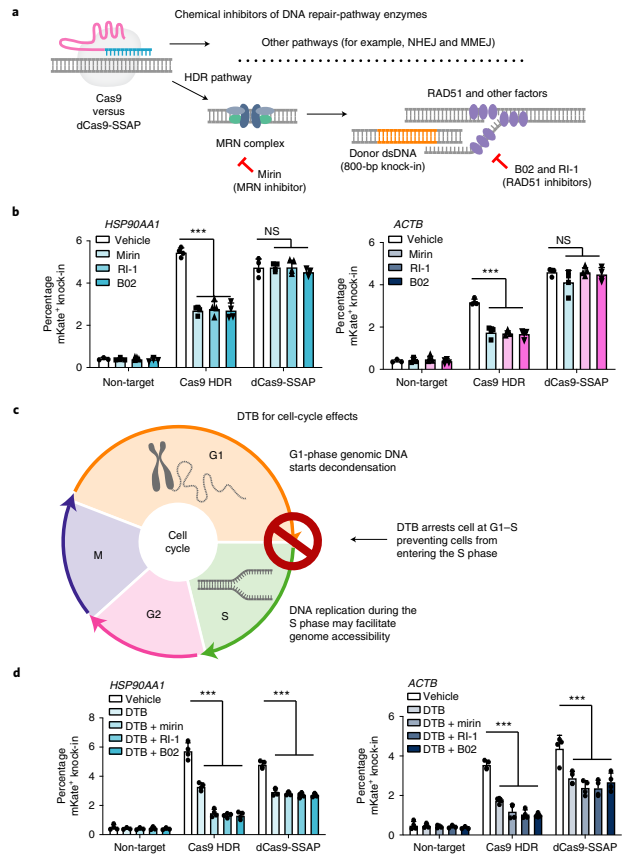
图三 dCas9-SSAP系统的机制研究[2]
最后,为了扩大dCas9-SSAP系统的应用前景,研究者筛选出截短型mSSAP系统,可将整个系统元件设计在两个载体中,利用AAV病毒系统来进行基因递送(图四c)。根据RecT蛋白的二级结构,共设计了14个不同长度的RecT截短体,并筛选出精准编辑效率与全长相近的最短截短体mRecT(45-251 AA)(图四a和b)。研究者进一步将dCas9系统换成体积更小的dSaCas9系统,使得dSaCas9-mSSAP系统可包装进两种AAV病毒。细胞水平的基因递送结果表明dSaCas9-mSSAP系统的精准编辑效率与全长dSaCas9-SSAP系统基本一致(图四d)。
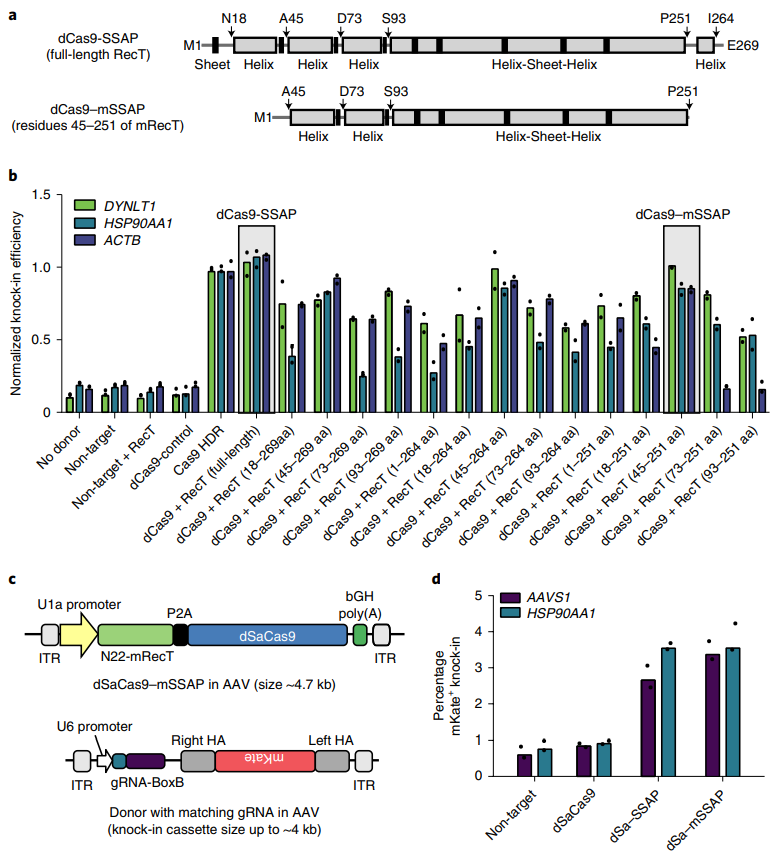
图四 截短型dSaCas9-mSSAP系统[2]
综上所述,dCas9-SSAP基因编辑工具不依赖于DNA损伤修复机制,极少造成DNA损伤,保证了基因组DNA的稳定性。其编辑产物主要为精准编辑产物,不会产生其他各种错误修复的基因型,显著提高了在体基因治疗的安全性。dCas9-SSAP系统的编辑效率与Cas9系统的精准编辑效率相当,甚至可能更高,为科学研究和基因治疗等转化应用提供了一种更为安全的选择。

GTP研发中心拥有自主知识产权的“人造精子细胞”介导的半克隆技术,可在体外实现“人造精子细胞”的多位点改造并进行功能元件测试,最后将“人造精子细胞”通过卵母细胞注射可以一步法获得基因改造小鼠。基于“人造精子细胞”单倍体打靶高效率的优势,可快速构建定制化小鼠,如基因组安全位点大片段敲入、条件性基因敲除/敲入和人源化基因改造等。多个复杂调控元件可以通过多次体外细胞打靶构建在同一株“人造精子细胞”中,经细胞水平质控后,一步法获得基因改造小鼠。

如有需要欢迎联系我们,GTP研发中心将竭诚为您设计最佳方案,制定专属于您的基因改造小鼠。
网址:http://www.sibcb.ac.cn/gtp/
邮箱:gtp_order@sibcb.ac.cn
地址:上海市岳阳路320号
责任编辑:张一鸣
新媒体运营:suway
参考文献
1. Maizels N, Davis L: Initiation of homologous recombination at DNA nicks. Nucleic Acids Research 2018, 46(14):6962-6973.
2. Wang C, Qu Y, Cheng JKW, Hughes NW, Zhang Q, Wang M, Cong L: dCas9-based gene editing for cleavage-free genomic knock-in of long sequences. Nat Cell Biol 2022, 24(2):268-278.
3. Noirot P, Kolodner RD: DNA strand invasion promoted by Escherichia coli RecT protein. J Biol Chem 1998, 273(20):12274-12280.
4. Thomason LC, Costantino N, Court DL: Examining a DNA Replication Requirement for Bacteriophage lambda Red- and Rac Prophage RecET-Promoted Recombination in Escherichia coli. mBio 2016, 7(5):eD1443-16.
5. Jones DL, Leroy P, Unoson C, Fange D, Curic V, Lawson MJ, Elf J: Kinetics of dCas9 target search in Escherichia coli. Science 2017, 357(6358):1420-1423.
6. Sternberg SH, Redding S, Jinek M, Greene EC, Doudna JA: DNA interrogation by the CRISPR RNA-guided endonuclease Cas9. Nature 2014, 507(7490):62-67.
7. Knight SC, Xie L, Deng W, Guglielmi B, Witkowsky LB, Bosanac L, Zhang ET, El Beheiry M, Masson JB, Dahan M et al: Dynamics of CRISPR-Cas9 genome interrogation in living cells. Science 2015, 350(6262):823-826.
8. Wang CK, Cheng JKW, Zhang QH, Hughes NW, Xia Q, Winslow MM, Cong L: Microbial single-strand annealing proteins enable CRISPR gene-editing tools with improved knock-in efficiencies and reduced off-target effects. Nucleic Acids Res 2021, 49(6):e36.

