技术分享:细胞选择性基因组编辑工具eiCRISPR系统的开发
CRISPR/Cas9系统是一种功能强大的基因组编辑工具,有助于阐明生物学中的基因功能,以及开发新的遗传疾病治疗方法[1]。该系统是由人工设计的sgRNA引导Cas9核酸酶对靶DNA进行特异性切割,从而实现对基因组DNA的定向改造。与以前报道的工具相比,该系统大大简化了基因组的编辑操作[2]。在一些特定的应用中需要严格时空调控CRISPR/Cas9活性,为此诱导型Cas9蛋白和调控sgRNA活性的方法被开发出来[3]。但这些方法通常会导致Cas9核酸酶活性受损或者需要对sgRNA进行复杂的化学修饰[4],此外,它们缺乏对疾病细胞的选择性,限制了其在化学生物学领域的应用[5]。
2022年12月,Journal of the American Chemical Society(JACS)杂志报道了一种由疾病相关酶诱导的CRISPR系统(enzyme-inducible CRISPR,eiCRISPR),它由Cas9蛋白、自封闭失活的向导RNA(bsgRNA)和化学笼修饰的脱氧核酶(DNAzyme)三部分组成,通过正交化学激活的方式实现肿瘤细胞选择性的基因组编辑。进一步地,研究者利用一种可生物降解的脂质纳米颗粒(LNP)递送系统,进行细胞及活体层面eiCRISPR系统的高效递送,实现肿瘤细胞特异性的基因组编辑与治疗[6]。
首先,研究者设计bsgRNA来调控eiCRISPR系统的活性。在bsgRNA的5' 端增加了与sgRNA间隔区互补的封闭序列,后者可以阻断bsgRNA和靶基因之间的相互作用,从而关闭eiCRISPR的基因组编辑活性(图一a)。分别测试携带6、13、17nt长度封闭序列的bsgRNA,发现均可抑制CRISPR/Cas9系统的切割活性,其中17nt封闭序列的抑制效果最佳(图一b和c),表明bsgRNA的设计可关闭eiCRISPR系统。
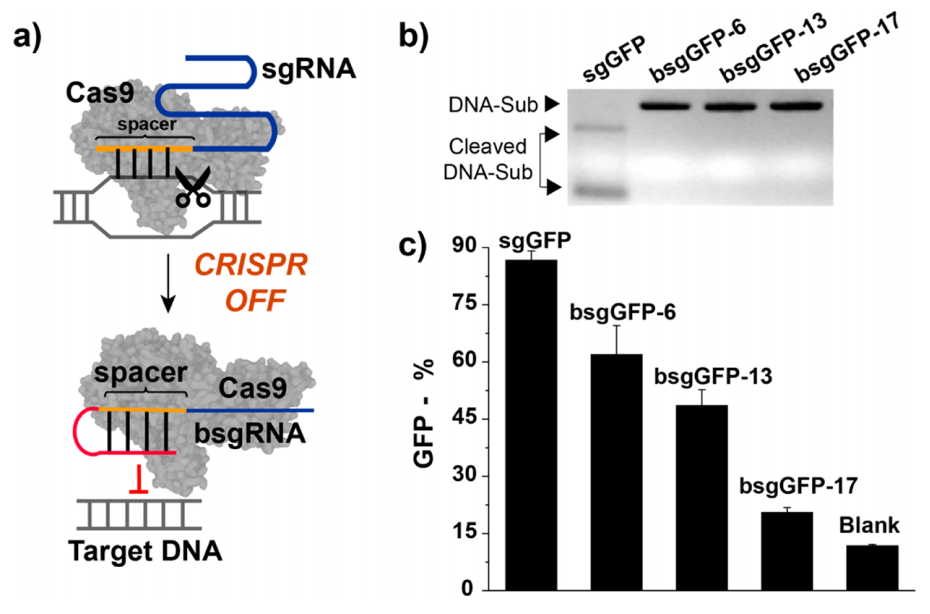
图一 bsgRNA的封闭序列设计与优化[6]
接着,研究者测试bsgRNA的封闭序列能否被DNAzyme特异性识别并切割,从而激活eiCRISPR系统。选择17nt封闭序列的bsgRNA(bsgGFP-17)和8-17E DNAzyme进行体外测试(图二a),发现8-17E DNAzyme处理后,bsgRNA产生了较短的RNA(图二b);并且在Cas9蛋白存在的情况下,恢复了对靶基因的DNA切割活性(图二c)。然而,催化活性突变的mt17E DNAzyme对照组则不能切割bsgRNA以及激活系统(图二b和c),表明DNAzyme序列特异性地介导bsgRNA切割成sgRNA,恢复对靶DNA的靶向性。研究者进一步将8-17E DNAzyme进行光笼修饰,获得无活性的17E-NB DNAzyme;在紫外光的照射下,17E-NB DNAzyme脱去光笼,恢复成有活性的8-17E DNAzyme(图二d)。电喷雾电离质谱(ESI-MS)表征了紫外光下DNAzyme的光笼修饰变化(图二d)。体外细胞实验结果显示,无活性的17E-NB DNAzyme对bsgRNA的激活效率较低;用紫外光照射15分钟后,可观察到有效的基因组编辑以及靶基因GFP敲除(图二e)。上述结果表明,8-17E DNAzyme可特异性切割bsgRNA,激活eiCRISPR系统;还能进一步将8-17E DNAzyme进行光笼修饰,通过光控激活eiCRISPR系统,实现条件性基因组编辑。
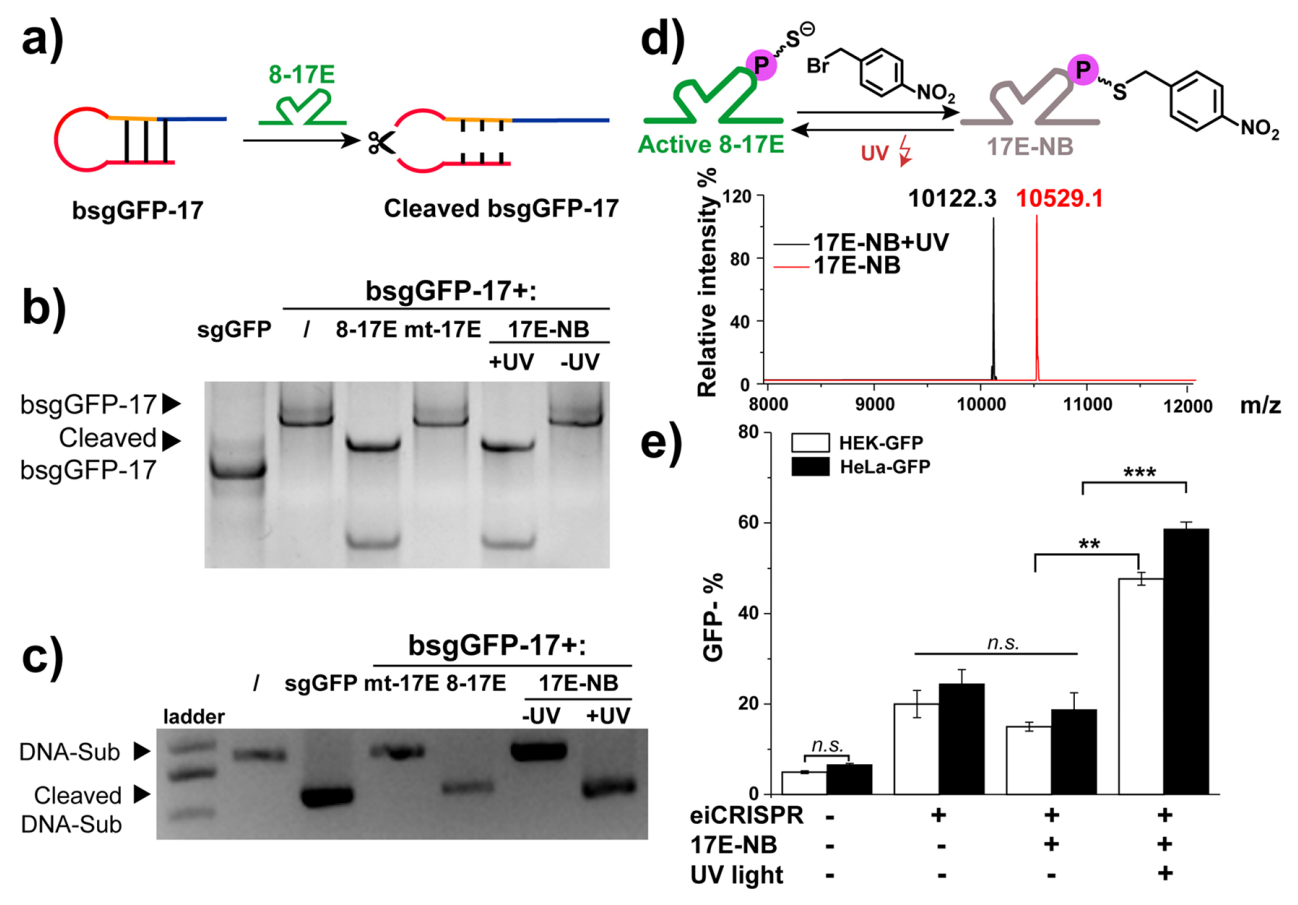
图二 17E-NB DNAzyme可实现光控激活eiCRISPR系统的基因组编辑[6]
研究者前期已证明肿瘤细胞内源性信号(如活性氧ROS和酶)的失调可以触发化学笼修饰蛋白质的脱笼反应[7]。另外,肿瘤细胞高表达的NAD(P)H:醌氧化还原酶(NQO1)可以重新激活被苯醌(QP)偶联的蛋白质[8]。于是,研究者测试苯基硼酸(PBA)和QP修饰的8-17E DNAzyme能否分别响应内源性 ROS 和 NQO1的激活(图三a)。ESI-MS分析证实,修饰后的17E-PBA和17E-QP分别在ROS和NQO1存在的情况下恢复成8-17E DNAzyme(图三b和c)。在Cas9存在时,17E-PBA和17E-QP可进一步响应激活信号实现靶基因的DNA切割(图三d)。体外细胞实验结果显示,17E-PBA和17E-QP均可响应HeLa细胞的内源性信号并降低60% GFP的表达,这与天然8-17E DNAzyme的基因编辑效率相当;然而它们在非肿瘤的HEK细胞中的编辑效率较低;阴性对照组其他修饰的17E-BA不响应肿瘤内源性信号(图三e)。以上数据表明,经化学笼修饰的DNAzyme可响应肿瘤细胞内源性信号,实现eiCRISPR系统的肿瘤细胞选择性基因组编辑。
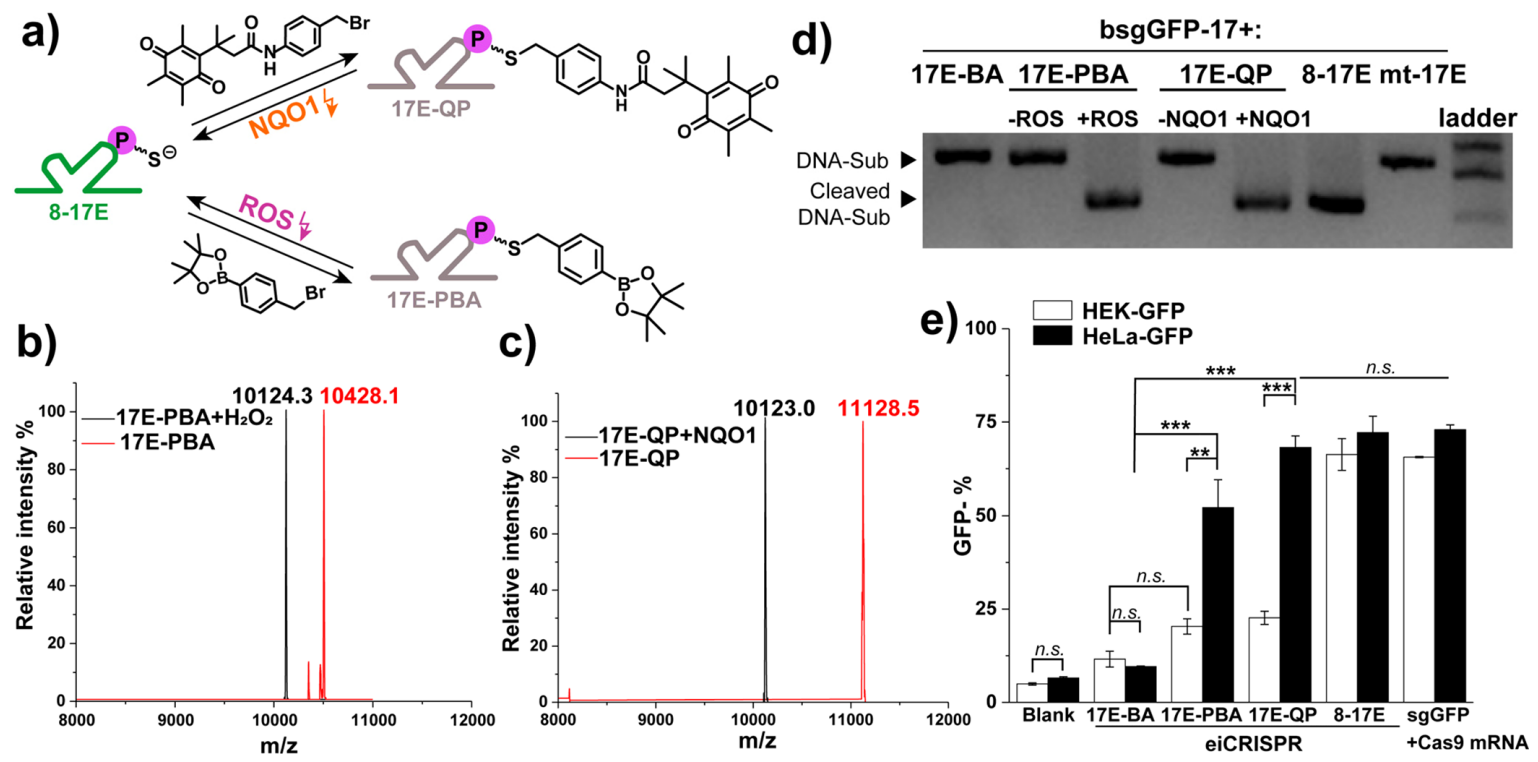
图三 eiCRISPR系统可实现肿瘤细胞选择性基因组编辑[6]
接下来,研究者测试eiCRISPR系统在肿瘤治疗中的应用,设计了靶向宫颈癌致癌性的人乳头瘤病毒18型(HPV18)E6基因,并在HPV18阳性的HeLa细胞中测试eiCRISPR系统的有效性。采用T7EI检测HeLa细胞基因组DNA的indel效率,发现阴性对照组17E-BA DNAzyme的indel效率低至1.4%,而17E-QP和17E-PBA DNAzyme的效率提高了14倍以上(图四a),展现出肿瘤细胞特异性的基因编辑能力。并且呈现出浓度依赖性的肿瘤细胞杀伤作用(图四b);在添加NQO1抑制剂双香豆素后,17E-QP DNAzyme的肿瘤细胞杀伤作用被显着抑制(图四c),表明eiCRISPR系统可选择性依赖肿瘤细胞内源性信号如NQO1,有效地激活肿瘤细胞的HPV18 E6基因编辑。
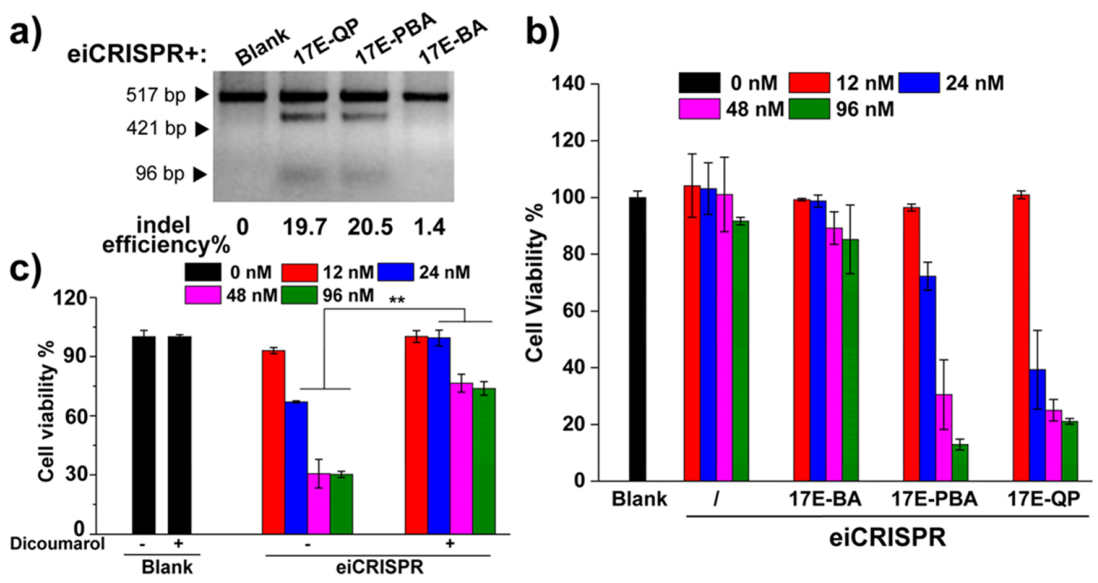
图四 正交激活eiCRISPR系统的HPV18 E6基因编辑和抗肿瘤研究[6]
最后,研究者测试了eiCRISPR系统的在体编辑能力,评估其对荷瘤小鼠异种移植物的肿瘤治疗有效性。将靶向HPV18 E6基因的eiCRISPR系统(17E-QP DNAzyme、bsgHPV-17和Cas9 mRNA)利用一种可生物降解的脂质纳米颗粒(LNP)进行递送,静脉注射到HeLa荷瘤小鼠体内。肿瘤生长监测显示,与接受PBS注射的小鼠相比,注射17E-QP DNAzyme实验组可显著抑制肿瘤生长,实验终点时肿瘤体积下降到对照组的30%(图五a和b)。肿瘤基因组DNA的T7EI检测显示,注射17E-QP DNAzyme实验组的HPV18 E6基因被切割,indel效率约为20.1%(图五c)。采用末端脱氧核苷酸转移酶介导的dUTP缺口末端标记(TUNEL)法检测细胞凋亡,发现17E-QP DNAzyme实验组的小鼠肿瘤发生了明显的细胞凋亡(图五d)。肿瘤切片的组织学H&E染色分析也发现经17E-QP DNAzyme实验组治疗后肿瘤细胞密度下降(图五d),对小鼠其他组织器官并无明显毒副作用。以上数据表明,eiCRISPR系统的在体编辑能力可有效抑制荷瘤小鼠的肿瘤生长。
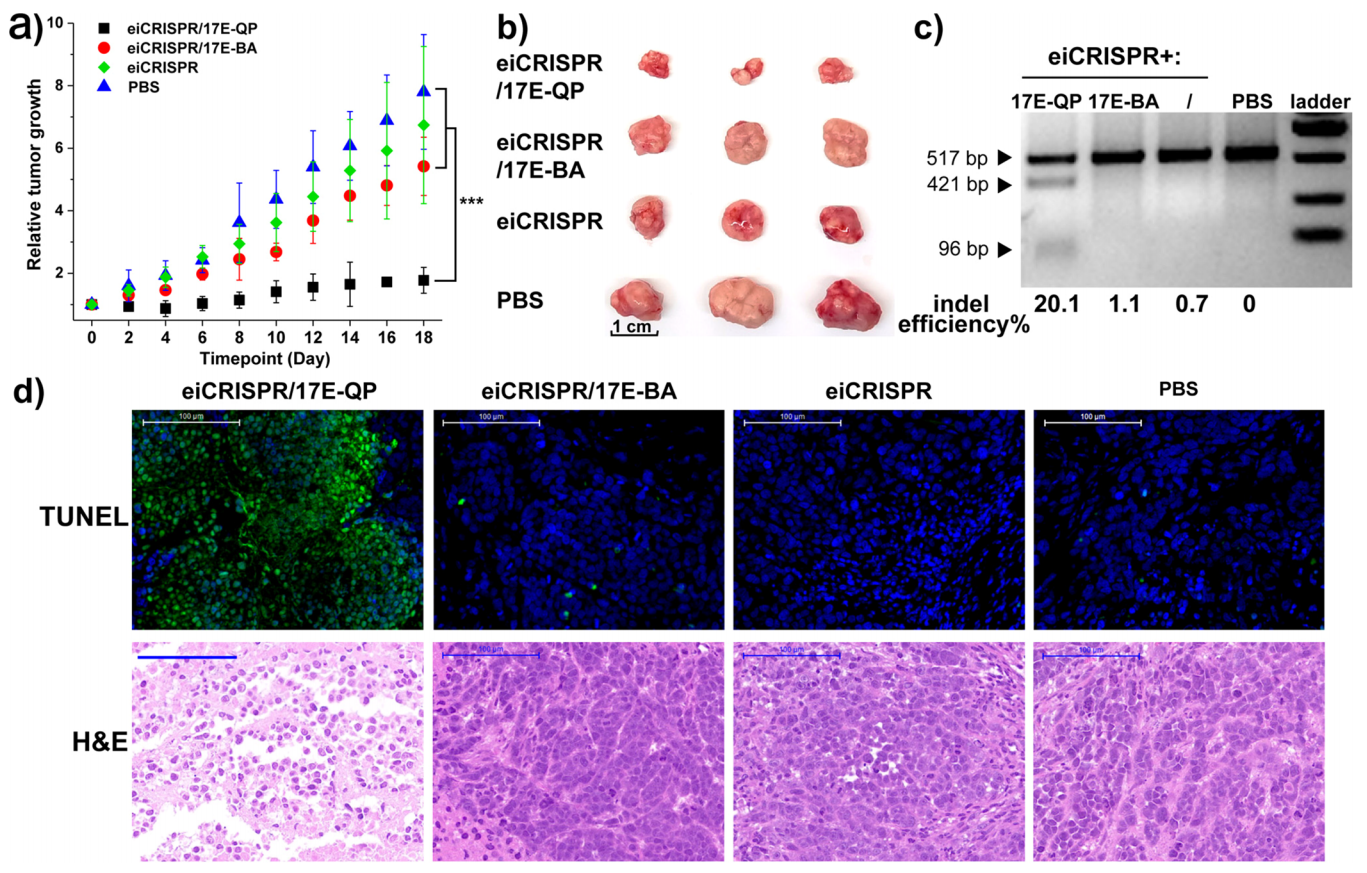
图五 在体递送eiCRISPR系统可抑制小鼠肿瘤生长[6]
综上所述,研究者开发的eiCRISPR系统,利用bsgRNA来关闭基因组编辑活性;当化学笼修饰的DNAzyme响应激活信号恢复成有活性的DNAzyme时,可特异性切割bsgRNA的封闭序列,进而激活eiCRISPR系统(图六a)。研究者设计的eiCRISPR系统可以被肿瘤细胞中失调的内源性信号正交激活,使细胞选择性的基因组编辑成为可能。后续进一步将DNAzyme的位点特异性化学笼修饰策略与细胞内源性化学反应相兼容,可升级开发新的eiCRISPR系统,用于细胞选择性基因组编辑和靶向基因治疗(图六b和c)。
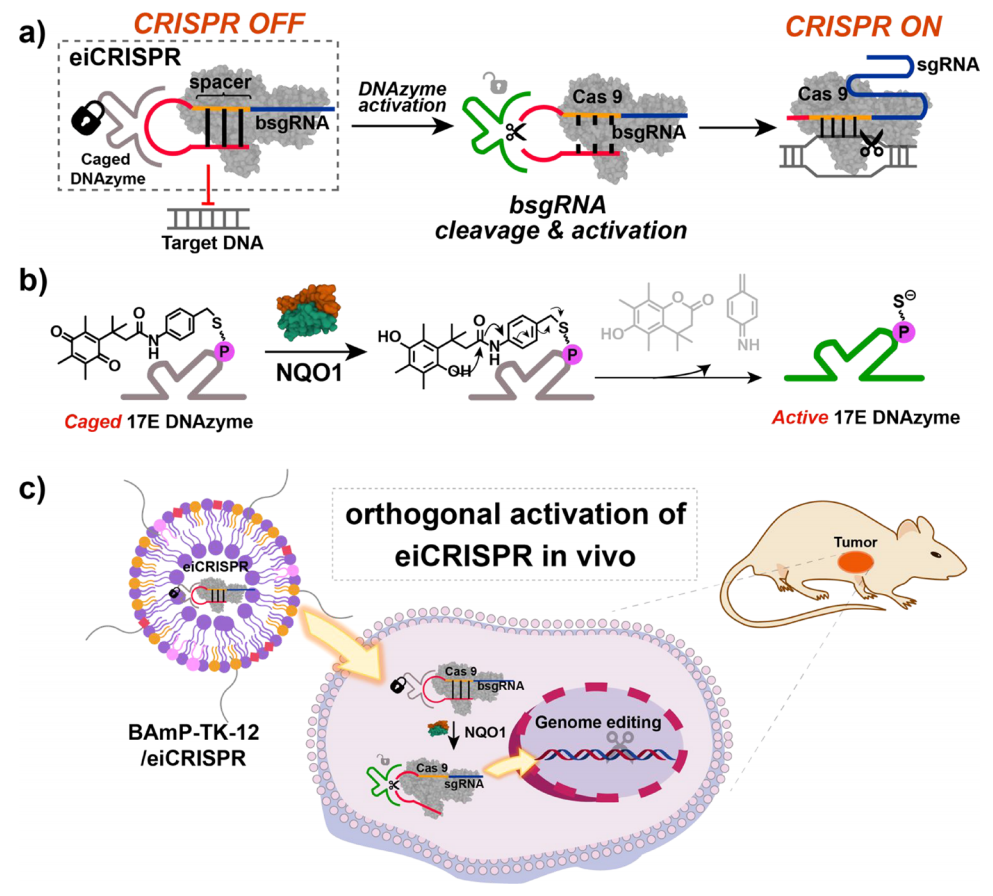
图六 细胞选择性eiCRISPR基因编辑系统[6]

GTP研发中心拥有自主知识产权的“人造精子细胞”介导的半克隆技术,可在体外实现“人造精子细胞”的多位点改造并进行功能元件测试,最后将“人造精子细胞”通过卵母细胞注射可以一步法获得基因改造小鼠。基于“人造精子细胞”单倍体打靶高效率的优势,可快速构建定制化小鼠,如基因组安全位点大片段敲入、条件性基因敲除/敲入和人源化基因改造等。多个复杂调控元件可以通过多次体外细胞打靶构建在同一株“人造精子细胞”中,经细胞水平质控后,一步法获得基因改造小鼠。

如有需要欢迎联系我们,GTP研发中心将竭诚为您设计最佳方案,制定专属于您的基因改造小鼠。
网址:http://www.sibcb.ac.cn/gtp/
地址:上海市岳阳路320号
责任编辑:多一百
新媒体运营:suway
参考文献
1. Clow PA, Du MH, Jillette N, Taghbalout A, Zhu JJ, Cheng AW: CRISPR-mediated multiplexed live cell imaging of nonrepetitive genomic loci with one guide RNA per locus. Nature communications 2022, 13(1).
2. Cai WQ, Wang M: Engineering nucleic acid chemistry for precise and controllable CRISPR/Cas9 genome editing. Sci Bull 2019, 64(24):1841-1849.
3. Iwasaki RS, Ozdilek BA, Garst AD, Choudhury A, Batey RT: Small molecule regulated sgRNAs enable control of genome editing in E. coli by Cas9. Nature Communications 2020, 11(1).
4. Zhang DY, Liu LP, Jin SJ, Tota E, Li ZJ, Piao XJ, Zhang X, Fu XD, Devaraj NK: Site-Specific and Enzymatic Cross-Linking of sgRNA Enables Wavelength-Selectable Photoactivated Control of CRISPR Gene Editing. J Am Chem Soc 2022, 144(10):4487-4495.
5. Chen G, Abdeen AA, Wang Y, Shahi PK, Robertson S, Xie R, Suzuki M, Pattnaik BR, Saha K, Gong S: A biodegradable nanocapsule delivers a Cas9 ribonucleoprotein complex for in vivo genome editing. Nature nanotechnology 2019, 14(10):974-980.
6. Cai W, Liu J, Chen X, Mao L, Wang M: Orthogonal Chemical Activation of Enzyme-Inducible CRISPR/Cas9 for Cell-Selective Genome Editing. J Am Chem Soc 2022, 144(48):22272-22280.
7. Wang M, Sun S, Neufeld CI, Perez-Ramirez B, Xu Q: Reactive oxygen species-responsive protein modification and its intracellular delivery for targeted cancer therapy. Angewandte Chemie 2014, 53(49):13444-13448.
8. Chang J, Cai WQ, Liang CJ, Tang Q, Chen XH, Jiang Y, Mao LQ, Wang M: Enzyme-Instructed Activation of Pro-protein Therapeutics In Vivo. J Am Chem Soc 2019, 141(45):18136-18141.



