技术分享:新型丝氨酸重组酶的系统性发现与开发
大丝氨酸重组酶(LSRs)是噬菌体携带的一种DNA整合酶,可以促进噬菌体基因组等移动遗传元件(MGEs)位点特异性整合到细菌基因组中。通过识别附着位点即DNA供体和受体分子上的识别序列,LSRs与靶序列形成联会复合体并催化靶位点切割、链交换和DNA重新连接。这种重组机制使得LSRs能够在不需要任何辅助因子以及不产生DNA双链断裂(DSBs)的情况下,进行位点特异性DNA插入。LSRs对供体DNA的大小没有明显的上限限制,使其成为一种极具吸引力的基因组编辑工具。然而,迄今为止,只有少数LSRs如Bxb1和PhiC31,可以催化DNA单向整合到同源附着位点[1, 2],现有LSRs的实际应用也受到整合效率低等因素的限制[3]。
2022年10月,Nature Biotechnology杂志在线报道了一种算法可识别出数千个全新的LSRs及其DNA附着位点,将现有的LSRs多样性扩大了100倍以上。这些LSRs可分为三种类型:着陆垫型、基因组靶向型和多靶向型。研究者在人源细胞系中测试这些LSRs的重组活性,发现新型LSRs的重组效率高达40-75%,比现有的Bxb1整合酶效率高7倍,而且可以重组超过7 kb的大片段DNA[4]。
两个不同的附着位点天然存在于噬菌体MGE(attP)和靶细菌基因组(attB)上,LSR可介导这两个附着位点之间的DNA重组,使得噬菌体MGE整合入细菌基因组中。当MGE插入后,附着位点保留在整合序列的边界,形成attL和attR。研究者通过比较基因组学的方法,从194585个临床和环境细菌的基因组中分析鉴定潜在的LSRs及其附着位点,最终筛选出了12638个候选LSRs及其attP和attB附着位点(图一a),并通过生物信息学预测这些LSRs的位点特异性(图一b)。研究者认为如果许多个远距离的LSRs均可靶向相同的靶基因簇,那么这些LSRs很可能是位点特异性的(图一c);反之,如果LSRs可靶向许多个不同靶基因簇,那意味着它们具有多靶向性(图一d)。由此,研究者发现了多个靶向相同基因簇的LSRs(图一e和f),其中一组共有33个特异性LSRs都靶向脯氨酰异构酶编码序列中的单个保守位点(图一f)。另外,研究者还发现了多靶向性的LSRs,它们具有大量相似的attB靶位点(图一g)。其中一个多靶向型LSR,可以靶向21个不同的attB靶位点。这些靶位点的比对结果显示都具有保守的TT二核苷酸核心,且在5′和3′末端富含T和A核苷酸(图一h),表明这些多靶向性的LSR直系同源物可能具有宽松的序列特异性。以上分析表明,研究者开发的计算方法可以大规模系统性发现潜在的LSRs及其附着位点。
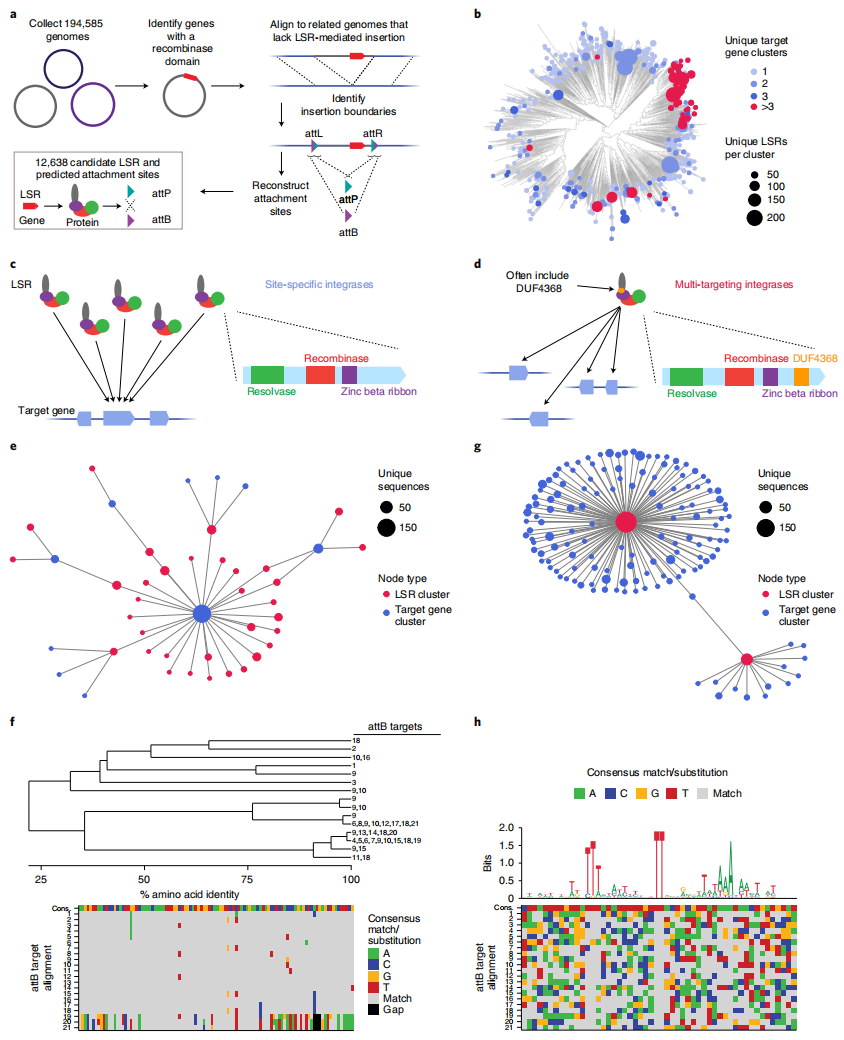
图一 潜在的LSRs及其特异性靶位点的系统性发现与分类[4]
接着,研究者首先选择了一批预测为单靶向特异性的LSRs来验证其应用效果。LSRs的应用之一是将遗传元件整合到已预先安装的基因组着陆垫处。为此,研究者合成了人源密码子优化的LSRs基因及其预测的attP和attB序列,并通过质粒重组试验来测试这些LSRs在HEK293FT人源细胞中的重组效率(图二a)。其中,attP质粒包含了一个无启动子的mCherry基因,该基因在与attB质粒重组后获得启动子,产生包含两个质粒序列的重组产物,从而表达mCherry荧光(图二a)。结果显示,在17个候选LSRs中,与对照组相比,有15个具有更强的mCherry荧光强度和更多的mCherry阳性细胞,其中有13个LSRs重组效率优于PhiC31,更有3个LSRs优于Bxb1(图二b和c)。研究者进一步选择其中具有多样性和良好重组效率的5个LSRs测试它们之间的正交性,结果发现这些LSRs高度正交,只能有效地整合到它们的同源附着位点上(图二d)。
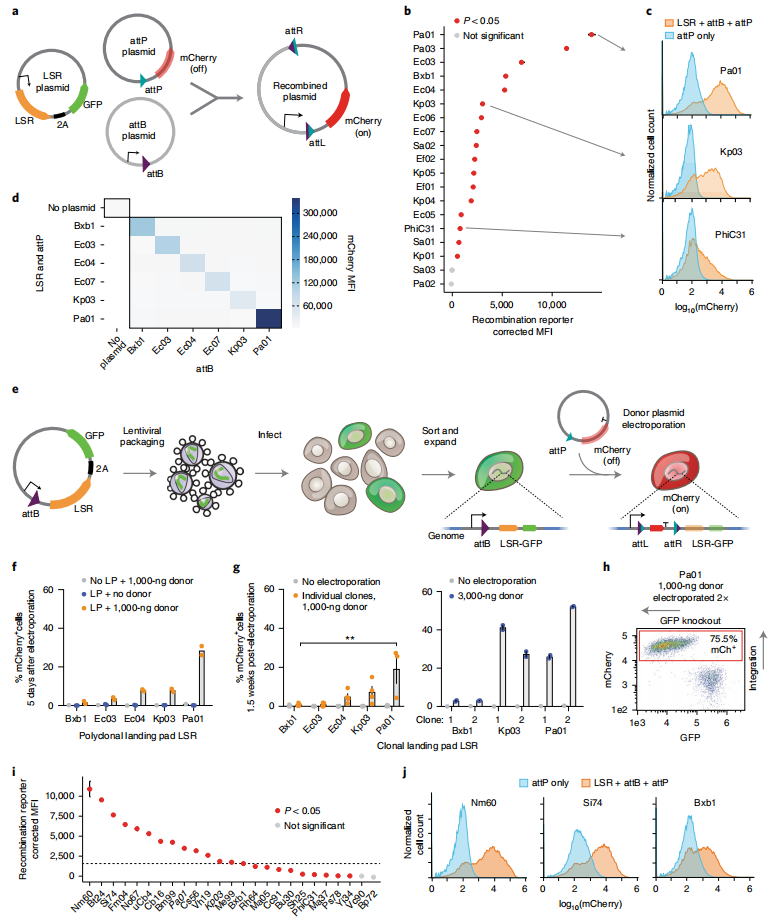
图二 着陆垫型LSRs的开发[4]
随后,研究者使用慢病毒将pEF1α-attB-LSR-T2A-GFP整合到K562细胞系的基因组中,生成表达LSR且含有attB着陆垫的多克隆K562细胞系,来测试LSR在人源基因组中的整合效率(图二e)。当3 kb的attP供体质粒实现成功重组后,将驱动mCherry表达,而LSR和GFP的表达终止(图二e)。结果显示,测试的4个新型LSRs重组效率均优于Bxb1(图二f)。用同样的方法测试LSRs在低拷贝的单克隆K562细胞系中的整合效率,发现Pa01在多个单克隆中的整合效率明显优于Bxb1(图二g左),且增加供体DNA剂量可随之提高Pa01的整合效率(图二g右),连续两次的供体质粒电转可将整合效率提高到75%以上(图二h)。第二轮的测试中,研究者从预测数据库中选择attB/P位点和人类基因组相似性低的LSRs,发现这轮21个新的LSRs中有17个在质粒重组试验中具有整合功能,其中16个的效率显著优于PhiC31,11个显著优于Bxb1(图二i和j)。以上数据表明,这些新发现的LSRs可以高效地将供体质粒整合到含有着陆垫的人源染色体DNA上,且整合效率高于已知的Bxb1或PhiC31。
近年来,平行报告基因分析(PRA)已成为研究各种分子元件文库的有效手段,包括增强子、启动子和非翻译区[5]。然而,PRA可能受到基因组位置效应或递送异质性造成的不利影响[6],因此,需要将PRA报告系统高效且稳定地整合到基因组单个位置[7, 8]。为了探索新的着陆垫型LSRs在功能基因组学中的运用,研究者建立了一个小型PRA,以测试整合在基因组中的合成增强子激活转录报告基因的能力(图三a)。研究者将包含不同数量TetO的报告基因整合到含有着陆垫的K562细胞克隆中,添加强力霉素Dox诱导,rTetR-VP48可结合到TetO上激活报告基因的表达(图三a)。报告基因包含有黄色荧光蛋白和IgG1-Fc表面标记,可实现高表达和低表达细胞的定量鉴定与快速分选(图三a)。结果显示,实验组和对照组的报告基因激活程度都与转录因子结合位点TetO的数量相对应(图三b),但只有实验组中这两者之间具有显著相关性(图三c)。以上结果表明,着陆垫型LSRs可以很好地用于报告基因文库的构建及其平行定量测量,实现各种功能基因组学研究。
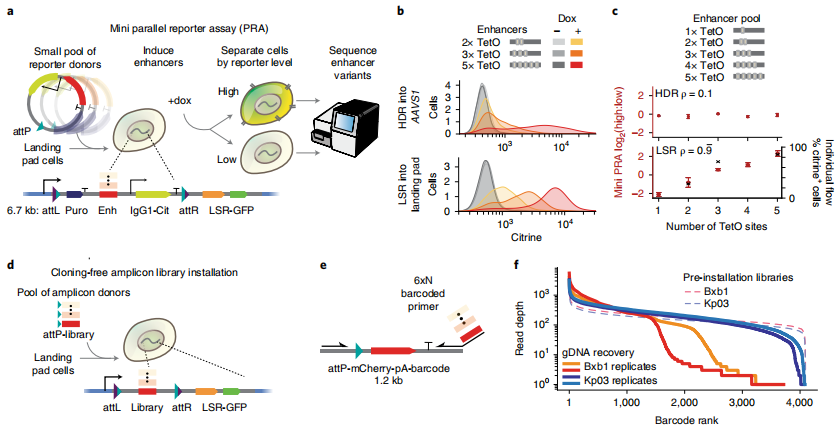
图三 着陆垫型LSRs用于平行报告基因分析和扩增子文库整合[4]
基因筛选涉及大规模质粒文库克隆构建[9],为了克服这一繁杂的构建步骤,研究者利用着陆垫型LSRs的整合方法,将PCR扩增子文库直接转染到含有Kp03和Bxb1着陆垫的细胞中(图三d和e)。结果显示,Kp03在整合线性供体方面的效率同样高于Bxb1(图三f)。以上数据表明,着陆垫型LSRs使得线性供体快速整合入基因组成为可能。
对于基因靶向型LSRs,可以在一个或多个安全位置直接将目标序列整合到人类基因组中。研究者使用BLAST搜索GRCh38人类基因组中的所有attB/P序列(图四a),筛选出与人类基因组至少有一个位点高度匹配的LSRs共856个(图四b和c)。接下来,研究者在人类基因组中定位了这些LSRs的整合位点,发现Sp56和Pf80在预测的靶位点有很高的特异性(图四d和e)。对于Dn29,61.9%的整合仅发生在前5个靶位点(图四f-h)。这种效率和特异性的结合,使得Dn29成为一个候选基因组靶向型LSR(图四h)。以上数据表明,研究者使用的计算方法发现了直接靶向人类基因组特定位点的LSRs,并预测了它们的靶向附着位点的偏好性。
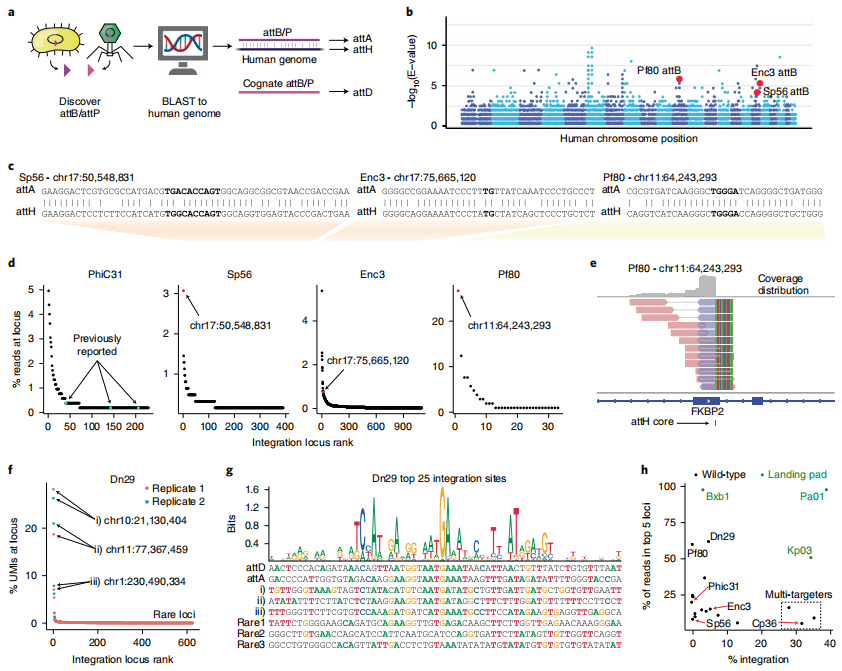
图四 直接靶向人类基因组特定位点的LSRs开发[4]
一些LSRs已进化出多靶向能力,使其能够靶向基因组中许多不同的attB位点[10]。相对于半随机整合酶,如慢病毒、猪痘或睡美人转座酶,有效插入一系列假位点对转基因整合非常有用。为了探索新的多靶向型LSRs,研究者测试来自产气荚膜梭菌的LSR(Cp36)的整合效率,结果显示Cp36成功地将DNA整合到K562和HEK293FT细胞的基因组中,效率高达40%(图五a)。利用整合位点定位分析,研究者确定了2000多个Cp36的独特整合位点(图五b),分析了Cp36整合位点的序列基序,发现该基序由富含A的5’区、AA二核苷酸核心区和富含T的3’区组成(图五c)。研究者进而比较了Cp36与半随机PiggyBac转座酶的整合效率,发现两者的效率相似(图五d),Cp36可在完成第一次整合反应后再次实现第二次整合且不丢失第一个整合序列(图五e),说明Cp36的整合具有单向性,这与双向整合酶PiggyBac不同。此外,用Cp36同时递送mCherry和BFP荧光报告供体可实现细胞同时表达这两种标记物(图五f),表明Cp36可在单次转染后产生具有多种遗传元件的细胞。以上数据表明,多靶向型LSRs的整合效率高且具有单向性。
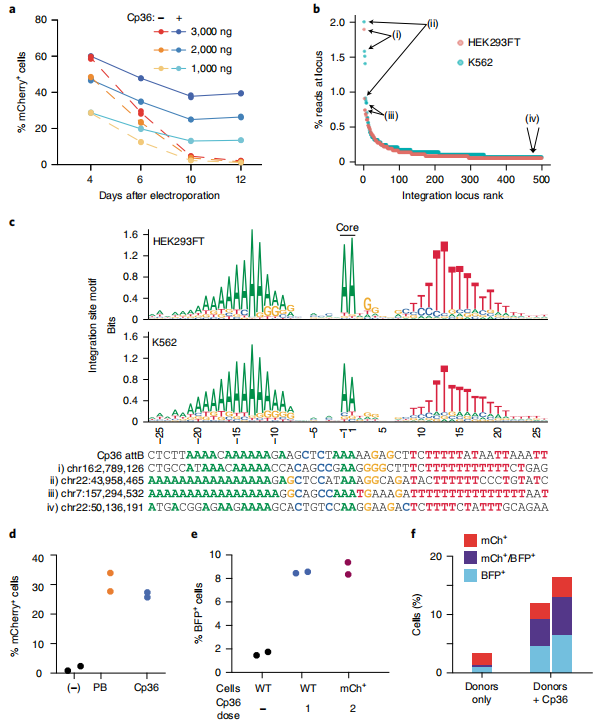
图五 多靶向型LSRs的开发[4]
综上所述,研究者发现了数千个新型LSRs,其中实验测试了60个以上新型LSRs的功能,分别展示了三个关键应用:(1)利用着陆垫型LSRs在基因组的着陆垫上高效整合质粒和扩增子文库;(2)直接靶向人类基因组中的特定位点,且比现有整合酶的重组效率更高;(3)在基因组上高效整合大片段DNA,以及在同一细胞中单次操作同时整合多个DNA片段。这些新型LSRs的重组效率大大优于现有的LSRs重组酶,可以将大片段DNA精准、高效地插入人类基因组中,为功能基因组学的研究以及人类遗传病的治疗提供了强有力的新工具。

GTP研发中心拥有自主知识产权的“人造精子细胞”介导的半克隆技术,可在体外实现“人造精子细胞”的多位点改造并进行功能元件测试,最后将“人造精子细胞”通过卵母细胞注射可以一步法获得基因改造小鼠。基于“人造精子细胞”单倍体打靶高效率的优势,可快速构建定制化小鼠,如基因组安全位点大片段敲入、条件性基因敲除/敲入和人源化基因改造等。多个复杂调控元件可以通过多次体外细胞打靶构建在同一株“人造精子细胞”中,经细胞水平质控后,一步法获得基因改造小鼠。

如有需要欢迎联系我们,GTP研发中心将竭诚为您设计最佳方案,制定专属于您的基因改造小鼠。
网址:http://www.sibcb.ac.cn/gtp/
邮箱:gtp_order@sibcb.ac.cn
地址:上海市岳阳路320号
责任编辑:甜点
新媒体运营:suway
参考文献
1. Smith MCM. Phage-encoded Serine Integrases and Other Large Serine Recombinases. Microbiology spectrum 2015, 3(4).
2. Jusiak B, Jagtap K, Gaidukov L, Duportet X, Bandara K, Chu JL, Zhang L, Weiss R, Lu TK. Comparison of Integrases Identifies Bxb1-GA Mutant as the Most Efficient Site-Specific Integrase System in Mammalian Cells. ACS synthetic biology 2019, 8(1):16-24.
3. Sivalingam J, Krishnan S, Ng WH, Lee SS, Phan TT, Kon OL. Biosafety Assessment of Site-directed Transgene Integration in Human Umbilical Cord-lining Cells. Mol Ther 2010, 18(7):1346-1356.
4. Durrant MG, Fanton A, Tycko J, Hinks M, Chandrasekaran SS, Perry NT, Schaepe J, Du PP, Lotfy P, Bassik MC et al. Systematic discovery of recombinases for efficient integration of large DNA sequences into the human genome. Nature biotechnology 2022.
5. King DM, Hong CKY, Shepherdson JL, Granas DM, Maricque BB, Cohen BA. Synthetic and genomic regulatory elements reveal aspects of cis-regulatory grammar in mouse embryonic stem cells. eLife 2020, 9.
6. Klein JC, Agarwal V, Inoue F, Keith A, Martin B, Kircher M, Ahituv N, Shendure J. A systematic evaluation of the design and context dependencies of massively parallel reporter assays. Nature methods 2020, 17(11):1083-1091.
7. Cao J, Novoa EM, Zhang Z, Chen WCW, Liu D, Choi GCG, Wong ASL, Wehrspaun C, Kellis M, Lu TK. High-throughput 5' UTR engineering for enhanced protein production in non-viral gene therapies. Nature communications 2021, 12(1):4138.
8. Matreyek KA, Stephany JJ, Chiasson MA, Hasle N, Fowler DM. An improved platform for functional assessment of large protein libraries in mammalian cells. Nucleic acids research 2020, 48(1):e1.
9. Hsu PD, Lander ES, Zhang F. Development and applications of CRISPR-Cas9 for genome engineering. Cell 2014, 157(6):1262-1278.
10. Smith MC, Thorpe HM. Diversity in the serine recombinases. Molecular microbiology 2002, 44(2):299-307.



