技术分享:外泌体介导Cas9 RNP复合物的有效递送,实现肝脏疾病的组织特异性基因治疗
CRISPR-Cas9系统介导的在体细胞基因组编辑,已在多种遗传疾病治疗中展示出巨大的潜力[1],但仍存在在体递送效率低和缺乏组织特异性等问题[2],限制了其广泛的临床应用。对于治疗性基因组编辑,Cas9核酸酶和单导向RNA(sgRNA)必须被有效地递送到细胞内并进入细胞核。其递送通常有三种选择,即编码Cas9和sgRNA的质粒DNA、Cas9 mRNA和sgRNA以及Cas9核糖核蛋白(RNP)。其中,RNP递送可避免前两种选择涉及到的DNA和mRNA在转录和翻译过程中遇到的问题,并在低免疫反应和弱脱靶活性的情况下高效完成基因组编辑[3]。然而,RNP的治疗性递送因其超过载体的承载能力而受到限制,并且容易在制剂和血流循环过程中发生降解或变性,阻碍了基于RNP的基因组编辑系统的治疗应用[4]。
外泌体是由细胞天然释放的纳米级细胞外囊泡,它们固有的生物相容性、运输能力和血流稳定性使其成为治疗性递送的潜在工具[5]。从转染了CRISPR-Cas9质粒的特定细胞中获得的内源性外泌体,已成功将Cas9 RNP递送到靶细胞[6],但该方法比较繁琐耗时。通过电穿孔将基于DNA或mRNA的CRISPR-Cas9系统装载到纯化的外泌体中,已被安全有效地用于治疗[7]。但是,通过电穿孔将RNP等大蛋白装载到外泌体中的方式,迄今尚未有成功报道。
2022年9月,Science Advances上报道了一种称为exosomeRNP的基因组编辑递送系统,该系统通过电穿孔将Cas9 RNP装载到从肝星状细胞(HSC;LX-2)分离纯化的外泌体中。exosomeRNP具有强大的肝脏特异性在体基因组编辑活性,可用于治疗不同的肝脏疾病,包括急性肝损伤、慢性肝纤维化和肝细胞癌(HCC)[8]。
首先,研究者对exosomeRNP进行表征分析。研究者成功从LX-2细胞中分离纯化出外泌体,蛋白印迹法检测结果显示其裂解产物表达了特异性标记物CD63和TSG101,但不表达高尔基体标记物GM130(图一A和B);动态光散射(DLS)和透射电镜(TEM)检测显示,外泌体大小在50-200 nm之间,呈碟状纳米囊泡结构(图一C和D)。通过电穿孔可成功将Cas9 RNP装载入纯化的外泌体中,形成exosomeRNP纳米复合物,其大小和形态没有发生明显变化(图一E-G)。
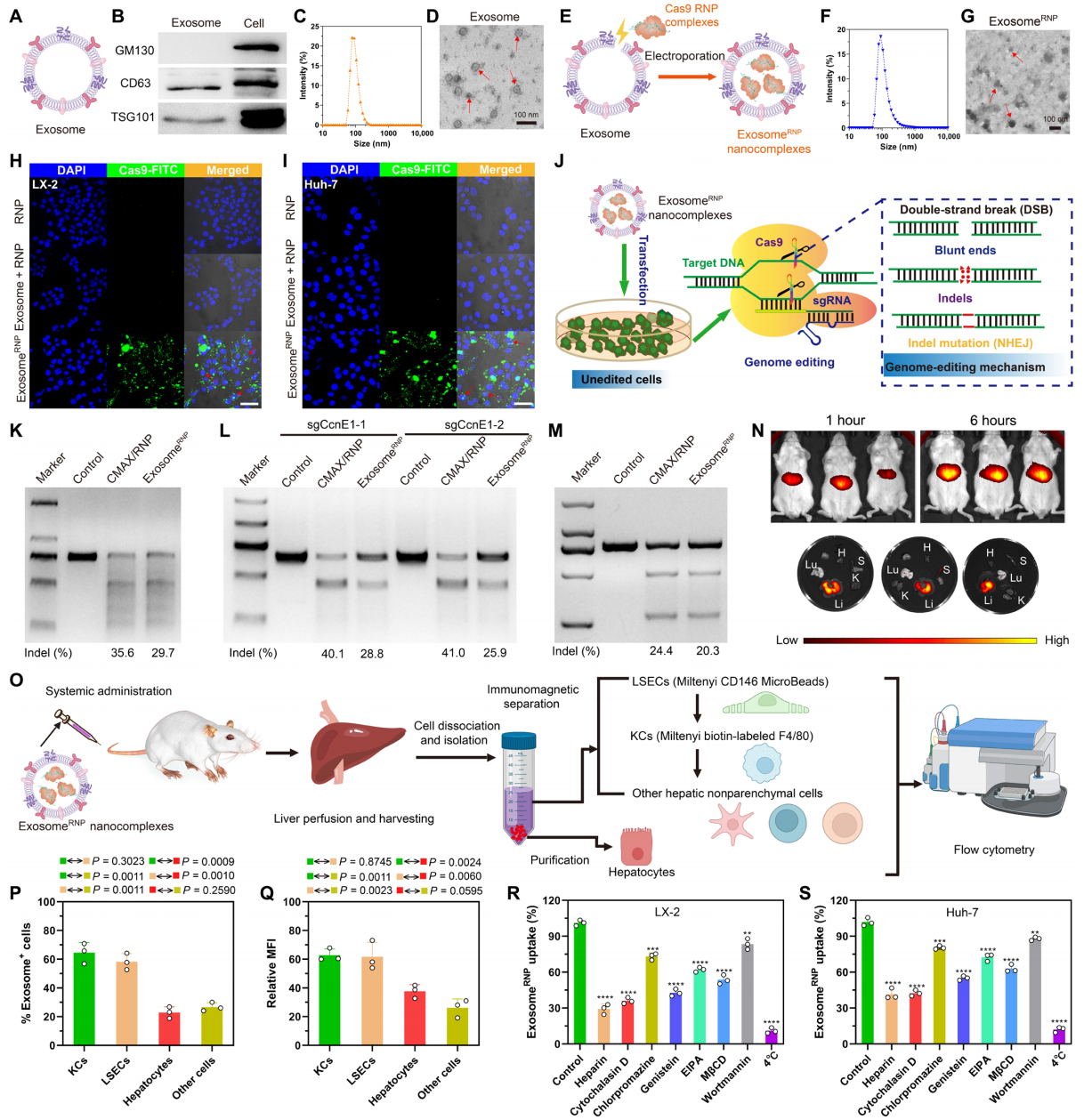
图一 exosomeRNP的特性、基因组编辑活性、生物分布以及细胞摄取机制[8]
为了检测体外递送效果,研究者利用exosomeRNP将Cas9-FITC递送至LX-2和Huh-7细胞中,结果显示仅在exosomeRNP组观察到有效的细胞质和细胞核递送(图一H和I)。研究者进一步在小鼠肝细胞中检测体外基因组编辑效果(图一J),T7E1结果显示exosomeRNP组在Bbc3(PUMA)、Ccne1和Kat5位点均实现了明显的序列插入/缺失,其效率与商品化RNP递送试剂CRISPRMAX(CMAX)相当(图一K-M)。在体内递送效果的评价中,exosomeRNP经小鼠尾静脉注射后,发现DiR标记的外泌体主要分布在小鼠的肝组织中(图一N)。进一步检测DiI标记的exosomeRNP在小鼠肝组织不同细胞类群中的生物分布,采用密度梯度法和磁珠法分离肝实质细胞、非实质库普弗细胞(KCs)、肝窦内皮细胞(LSECs)和其他非实质细胞(图一O)。结果显示,成功摄取外泌体的阳性细胞比例在不同细胞类群中有显著差别,其中KCs和LSECs显著高于肝实质细胞(图一P),且两者的相对平均荧光强度(MFI)也显著高于肝实质细胞(图一Q)。接下来,研究者具体研究exosomeRNP在细胞中的摄取机制。当细胞在4°C条件下孵育时,exosomeRNP的摄取显著减少(图一R和S),表明其细胞摄取具有能量依赖性。用不同抑制机制的一组内吞抑制剂分别处理细胞,结果显示所有组均抑制了exosomeRNP的摄取,其中肝素和细胞松弛素D的抑制作用最为显著(图一R和S),表明exosomeRNP可以通过多种途径被细胞摄取,包括网格蛋白介导的内吞作用、小窝蛋白介导的内吞、巨胞饮作用和吞噬作用。以上数据表明,研究者成功地从LX-2细胞中分离纯化出外泌体,通过电穿孔将Cas9 RNP装载入exosomeRNP中。exosomeRNP递送具有肝组织特异性,可以在体外以及体内有效地实现肝细胞内Cas9 RNP递送以及基因组编辑,提示其在治疗肝脏相关疾病方面具有潜在益处。
进而,研究者利用exosomeRNP分别测试三种肝脏疾病模型的治疗效果。其一是急性肝损伤模型。过量服用对乙酰氨基酚(APAP)会导致急性肝功能衰竭,而p53上调的凋亡调控因子PUMA在APAP诱导的肝损伤中起着关键作用[9]。因此,研究者设计了靶向PUMA的sgRNA来研究exosomeRNP介导的基因组编辑对急性肝损伤的治疗疗效(图二A)。结果显示,APAP处理后的小鼠肝脏中PUMA蛋白表达以及血清中AST和ALT水平增加,但在exosomeRNP治疗后被显著降低(图二B,E和F)。T7E1结果显示exosomeRNP递送导致靶位点序列插入/缺失率为26.1%,且仅在肝脏中观察到(图二C和D)。肝组织的HE染色结果显示,exosomeRNP治疗后的小鼠肝组织小叶中心细胞坏死和充血显著减少(图二G和J),TUNEL阳性细胞显著减少(图二H和K),与染色质结合的核蛋白HMGB1的核定位比例显著增加(图二I和L)。此外,exosomeRNP治疗还提高了小鼠的72小时生存率(图二M)。以上数据表明,exosomeRNP介导的基因组编辑可改善APAP诱导的急性肝损伤。
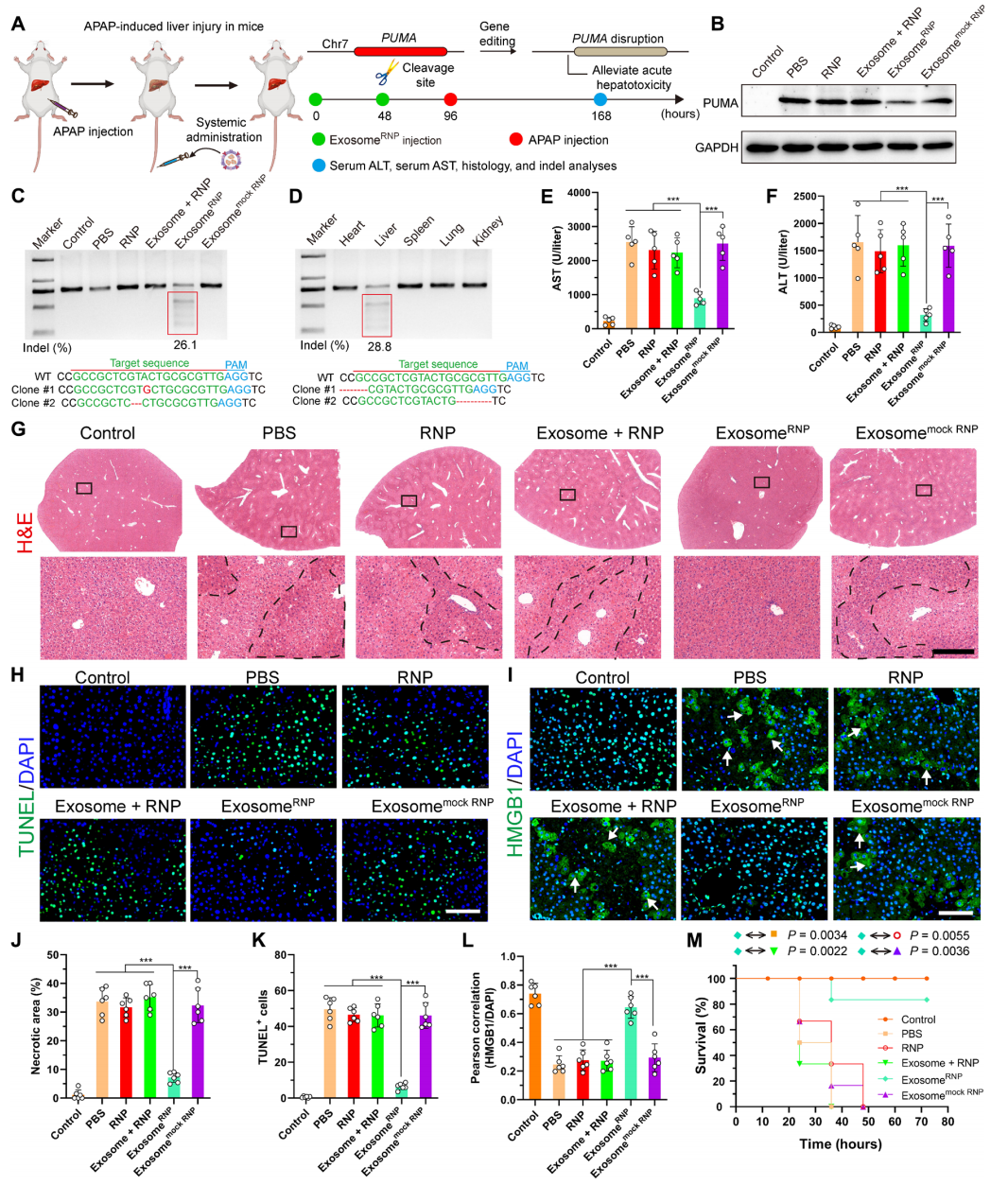
图二 exosomeRNP介导的基因组编辑可改善急性肝损伤[8]
其二是慢性肝纤维化模型。细胞周期蛋白E1(Ccne1)促进HSC的增殖,并在肝纤维化中发挥重要作用[10]。因此,研究者设计了针对Ccne1的sgRNA来研究exosomeRNP介导的基因组编辑在CCL4诱导的小鼠肝纤维化模型中的治疗效果(图三A)。结果显示,exosomeRNP治疗后,靶位点序列的插入/缺失率约为9.7%,且仅在肝组织中观察到(图三B和C);exosomeRNP治疗显著降低了CCNE1和a-SMA的表达(图三D)。小鼠肝切片的HE染色(图三E)、肝纤维化Ishak评分(图三F)、胶原蛋白表达(图三G)和羟脯氨酸含量测定(图三H)结果显示,靶向Ccne1的exosomeRNP治疗显著改善了肝纤维化疾病指标。以上数据表明,exosomeRNP介导的基因组编辑能有效改善CCL4诱导的肝纤维化。
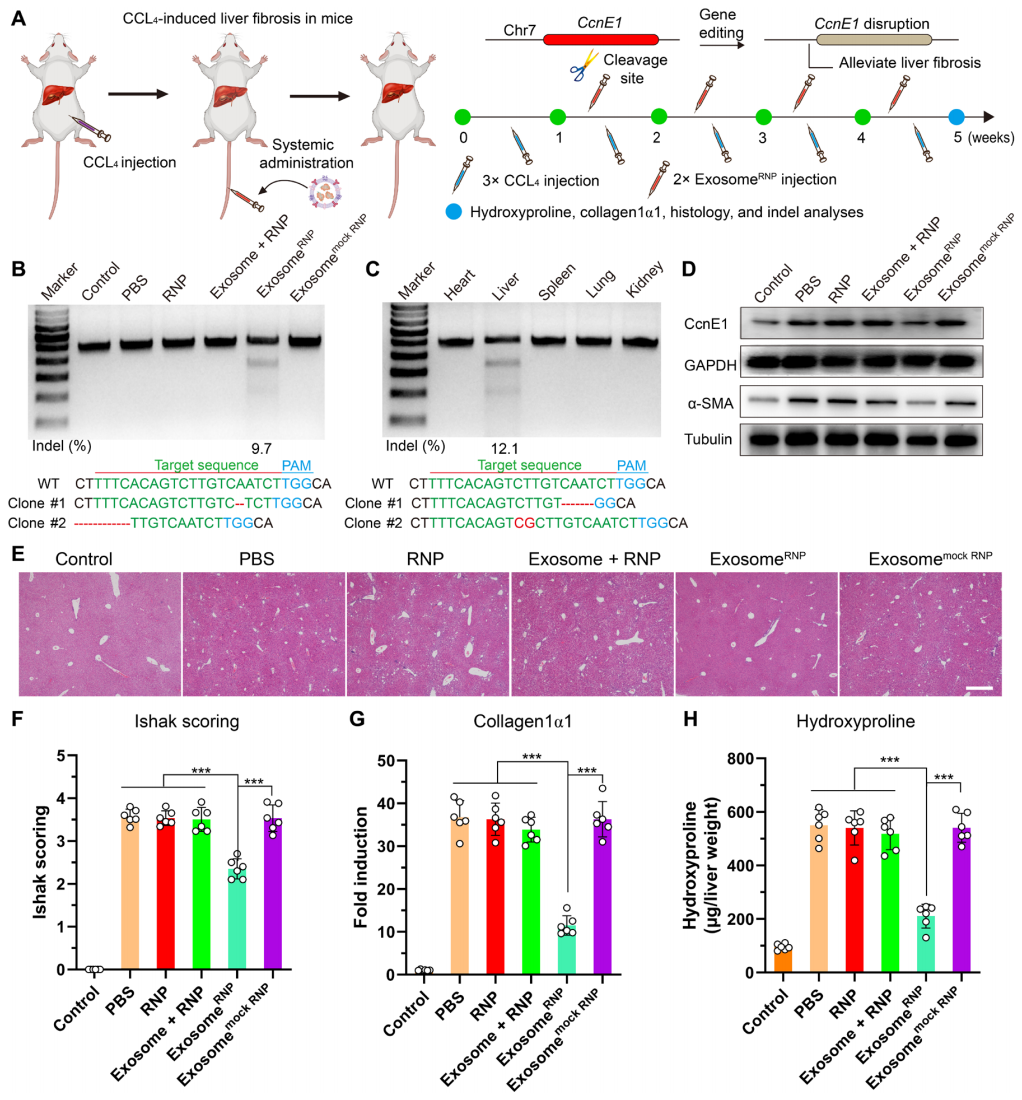
图三 exosomeRNP介导的基因组编辑可改善CCL4诱导的肝纤维化[8]
其三是肝细胞癌(HCC)模型。KAT5蛋白是HCC生长所必需的,破坏KAT5可在体外以及体内抑制HCC的增殖[11]。最后,研究者设计了靶向Kat5的sgRNA研究exosomeRNP介导的基因组编辑对原位HCC模型的治疗效果(图四A)。exosomeRNP治疗后,KAT5蛋白表达水平被显著降低(图四B);小鼠表现为最小的肿瘤体积以及最弱的生物发光强度负荷(图四C);小鼠生存期被显著延长(图四D);靶位点序列的插入/缺失率约为21.3%(图四E);肝脏切片的肿瘤细胞最少(图四F)。以上数据表明,exosomeRNP介导的靶向Kat5基因组编辑能有效改善原位HCC发展进程。
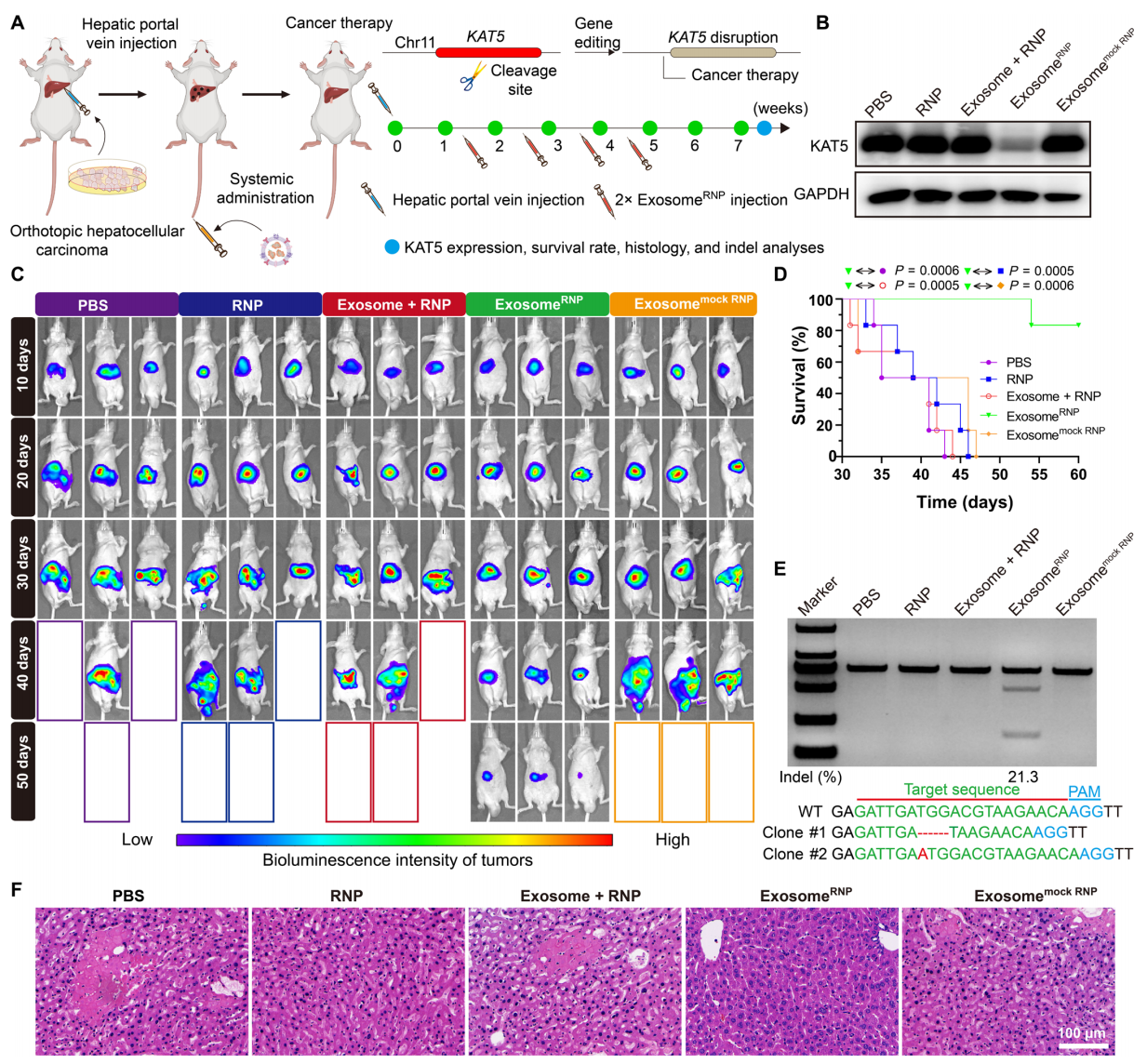
图四 exosomeRNP介导的基因组编辑用于治疗原位HCC[8]
综上所述,研究者开发了一种基于外泌体的递送系统exosomeRNP,通过电穿孔将Cas9 RNP有效地装载到来源于肝星状细胞的外泌体中,实现肝脏特异性递送和基因组编辑。exosomeRNP在急性肝损伤、慢性肝纤维化和HCC小鼠模型中均展现出强大的治疗潜力。exosomeRNP不仅克服了RNP的递送障碍,还为肝病的组织特异性精准基因治疗开辟了新的途径。此外,该技术可拓展成为有效的通用型非病毒载体,用于各种蛋白质及RNP的组织特异性细胞递送。

GTP研发中心拥有自主知识产权的“人造精子细胞”介导的半克隆技术,可在体外实现“人造精子细胞”的多位点改造并进行功能元件测试,最后将“人造精子细胞”通过卵母细胞注射可以一步法获得基因改造小鼠。基于“人造精子细胞”单倍体打靶高效率的优势,可快速构建定制化小鼠,如基因组安全位点大片段敲入、条件性基因敲除/敲入和人源化基因改造等。多个复杂调控元件可以通过多次体外细胞打靶构建在同一株“人造精子细胞”中,经细胞水平质控后,一步法获得基因改造小鼠。

如有需要欢迎联系我们,GTP研发中心将竭诚为您设计最佳方案,制定专属于您的基因改造小鼠。
网址:http://www.sibcb.ac.cn/gtp/
地址:上海市岳阳路320号
责任编辑:甜点
新媒体运营:suway
参考文献
1. Wan T, Ping Y. Delivery of genome-editing biomacromolecules for treatment of lung genetic disorders. Adv Drug Deliv Rev 2021, 168:196-216.
2. Behr M, Zhou J, Xu B, Zhang H. In vivo delivery of CRISPR-Cas9 therapeutics: Progress and challenges. Acta Pharm Sin B 2021, 11(8):2150-2171.
3. Xu X, Wan T, Xin H, Li D, Pan H, Wu J, Ping Y. Delivery of CRISPR/Cas9 for therapeutic genome editing. The journal of gene medicine 2019, 21(7):e3107.
4. Duan L, Ouyang K, Xu X, Xu L, Wen C, Zhou X, Qin Z, Xu Z, Sun W, Liang Y. Nanoparticle Delivery of CRISPR/Cas9 for Genome Editing. Frontiers in genetics 2021, 12:673286.
5. Kalluri R, LeBleu VS. The biology, function, and biomedical applications of exosomes. Science 2020, 367(6478).
6. Ye Y, Zhang X, Xie F, Xu B, Xie P, Yang T, Shi Q, Zhang CY, Zhang Y, Chen J et al. An engineered exosome for delivering sgRNA:Cas9 ribonucleoprotein complex and genome editing in recipient cells. Biomater Sci 2020, 8(10):2966-2976.
7. Gulei D, Berindan-Neagoe I. Activation of Necroptosis by Engineered Self Tumor-Derived Exosomes Loaded with CRISPR/Cas9. Mol Ther Nucleic Acids 2019, 17:448-451.
8. Wan T, Zhong J, Pan Q, Zhou T, Ping Y, Liu X. Exosome-mediated delivery of Cas9 ribonucleoprotein complexes for tissue-specific gene therapy of liver diseases. Sci Adv 2022, 8(37):eabp9435.
9. Chen D, Ni HM, Wang L, Ma X, Yu J, Ding WX, Zhang L. p53 Up-regulated Modulator of Apoptosis Induction Mediates Acetaminophen-Induced Necrosis and Liver Injury in Mice. Hepatology 2019, 69(5):2164-2179.
10. Nevzorova YA, Bangen JM, Hu W, Haas U, Weiskirchen R, Gassler N, Huss S, Tacke F, Sicinski P, Trautwein C et al. Cyclin E1 controls proliferation of hepatic stellate cells and is essential for liver fibrogenesis in mice. Hepatology 2012, 56(3):1140-1149.
11. Kwan SY, Sheel A, Song CQ, Zhang XO, Jiang T, Dang H, Cao Y, Ozata DM, Mou H, Yin H et al. Depletion of TRRAP Induces p53-Independent Senescence in Liver Cancer by Down-Regulating Mitotic Genes. Hepatology 2020, 71(1):275-290.



