技术分享:Cas9通过卷曲螺旋二聚体招募核酸外切酶协同增强基因敲除效率
CRISPR/Cas系统是一种功能强大且用途广泛的基因组编辑工具,可以通过纠正诱发疾病的突变或破坏致病基因来治疗各种遗传性疾病[1]。因此,提高CRISPR/Cas系统的编辑效率是生物技术和转化医学的热门研究。目前提高编辑效率的方法包括优化gRNA设计[2]、突变Cas及其同源物[3]、以及将Cas与其他蛋白融合使用[4]等。虽然不同的蛋白融合方式可拓宽CRISPR/Cas系统的应用潜力,但也存在一些缺点,比如融合蛋白的折叠问题和蛋白大小超过病毒载体包装上限等。一种利用卷曲螺旋二聚体肽链(Coiled-coil dimerization peptides,CC) 的蛋白结合方法可规避上述融合蛋白的缺点,这些短肽可基于静电和疏水相互作用实现紧密且特异的同源或异源二聚体配对[5],已被用于dCas9与转录调控结构域的结合[6]。
2022年6月,Nature Communications上报道了一种基于Cas9和核酸外切酶协同增强基因敲除效率的CCExo(CRISPR-Coilled coil-Exonuclease)系统。该系统利用基因编码的卷曲螺旋二聚体肽链将核酸外切酶招募到Cas9/gRNA复合物上,进一步降解切割位点的DNA链,在不同细胞系的多个靶点和癌症动物模型中均显示出比传统CRISPR/Cas系统更高的基因敲除效率[7]。
由于CRISPR/Cas系统诱导的DNA双链断裂(DSB)会触发招募非同源末端连接(NHEJ)机制相关蛋白,包括感知DNA断裂的蛋白质和DNA修饰酶[8]。因此研究者推断核酸外切酶可通过降解DSB区域的DNA,阻碍目标DNA的正确修复连接,从而提高基因敲除效率(图一a)。首先,研究者将Cas9分别与3种不同的核酸外切酶ExoI、mTREX2和ExoIII共价融合,分别靶向HEK293细胞系的MYD88、EMX1和VEGFα基因组进行测试。结果发现3种核酸外切酶实验组的插入/缺失比率虽都有所增加,但仅有效率最高的ExoIII实验组结果较为显著(图一b),表明Cas9与核酸外切酶的共价融合方式并没有显著提高基因敲除效率。
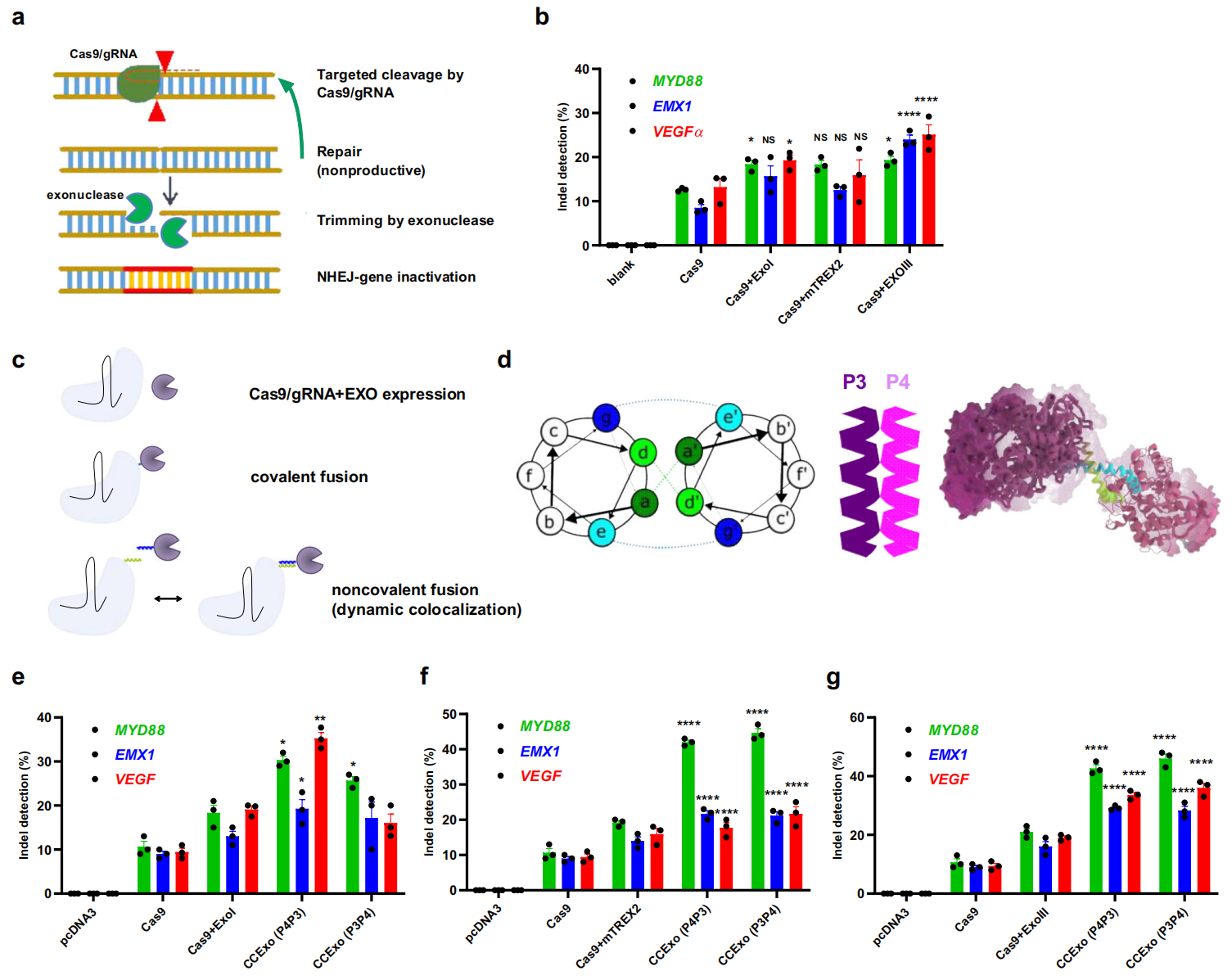
图一 Cas9与核酸外切酶通过卷曲螺旋二聚体结合后可显著提高基因敲除效率[7]
因此,研究者选择卷曲螺旋二聚体肽链迫使Cas9和ExoIII相接近的非共价方式来进行测试,分别将Cas9和ExoIII与先前设计的由四种七肽重复单元组成的卷曲螺旋二聚体P3或P4相融合,使得它们在P3和P4的相互作用下形成异二聚体[9],将ExoIII招募到Cas/gRNA复合物上(图一c-d)。研究者测试不同的非共价组合方式,发现卷曲螺旋二聚体驱动的Cas9-核酸外切酶二聚化(CCExo)相比传统CRISPR/Cas系统都显著增加了插入/缺失比率,其中ExoIII在所有靶点的基因敲除效率仍是最高(图一e-g)。
由于卷曲螺旋二聚体的亲和力和方向可能会影响CCExo系统的效率[9],研究者进一步在HEK293细胞上测试另外三对卷曲螺旋二聚体:强亲和力的N5-N6、亲和力较弱的P3S-P4S以及反向平行的P3-AP4(图二a),并设计多个靶向CD47基因第二外显子的gRNAs进行测试(图二b)。结果显示,Cas9和ExoIII的共价融合与传统CRISPR/Cas系统的基因敲除效率相似,而CCExo实验组均能显著增强基因敲除效率,其中N5N6组的基因敲除效率最高(图二c-j);卷曲螺旋二聚体的方向不会影响CCExo的效率(图二j);并且CCExo系统的Cas9蛋白表达水平与传统CRISPR/Cas系统相当(图二k)。以上数据表明,CCExo系统的优越性能源于非共价结合方式,其基因敲除效率受卷曲螺旋二聚体的亲和力影响,但不依赖于二聚体的方向。
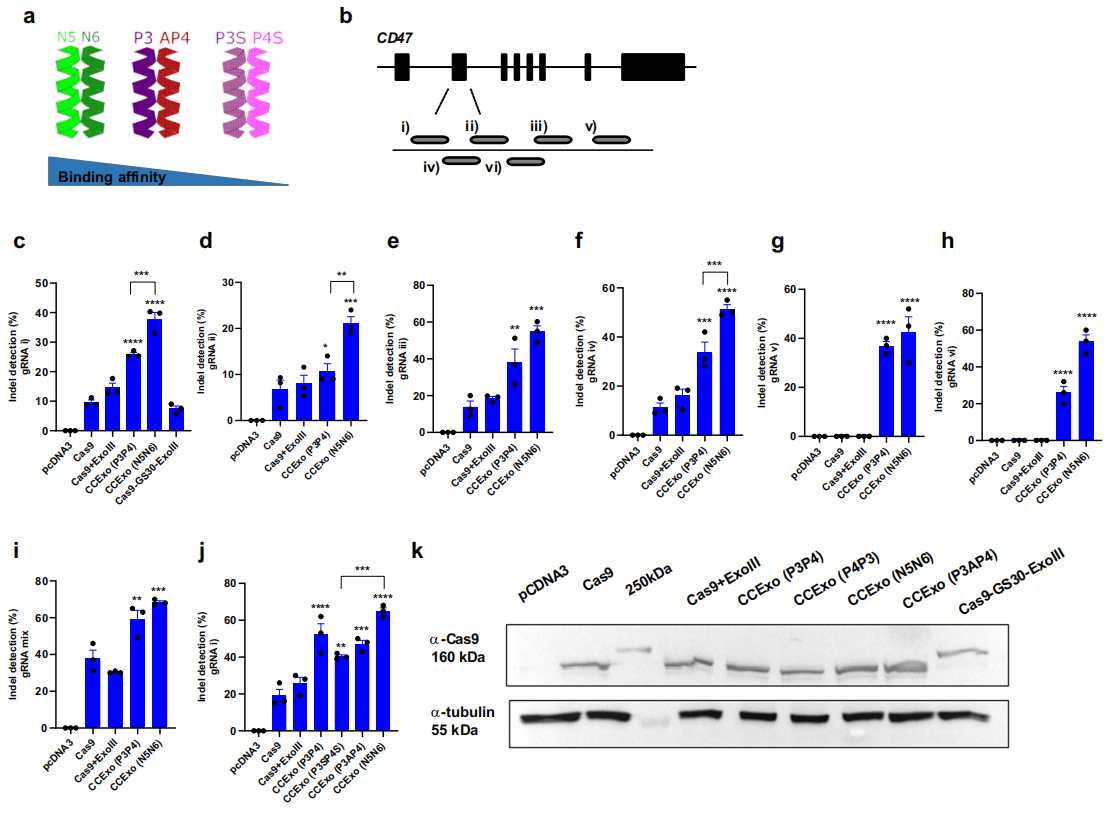
图二 卷曲螺旋二聚体的属性对CCExo系统基因敲除效率的影响[7]
接下来,研究者对CCExo系统的基因编辑产物进行NGS分析。结果显示CCExo系统的基因编辑增加了所有靶位点的片段缺失长度,特别是在亲和力强的N5N6实验组中观察到大于200 bp的片段缺失(图三a-d)。此外,研究者还检测了VEGFα和TRBC1位点的潜在脱靶活性,结果显示N5N6 CCExo系统的脱靶活性与传统CRISPR/Cas系统相比没有差异(图三e)。以上数据进一步表明,CCExo系统可以显著增强Cas9的基因敲除效率,且没有提高脱靶效率。
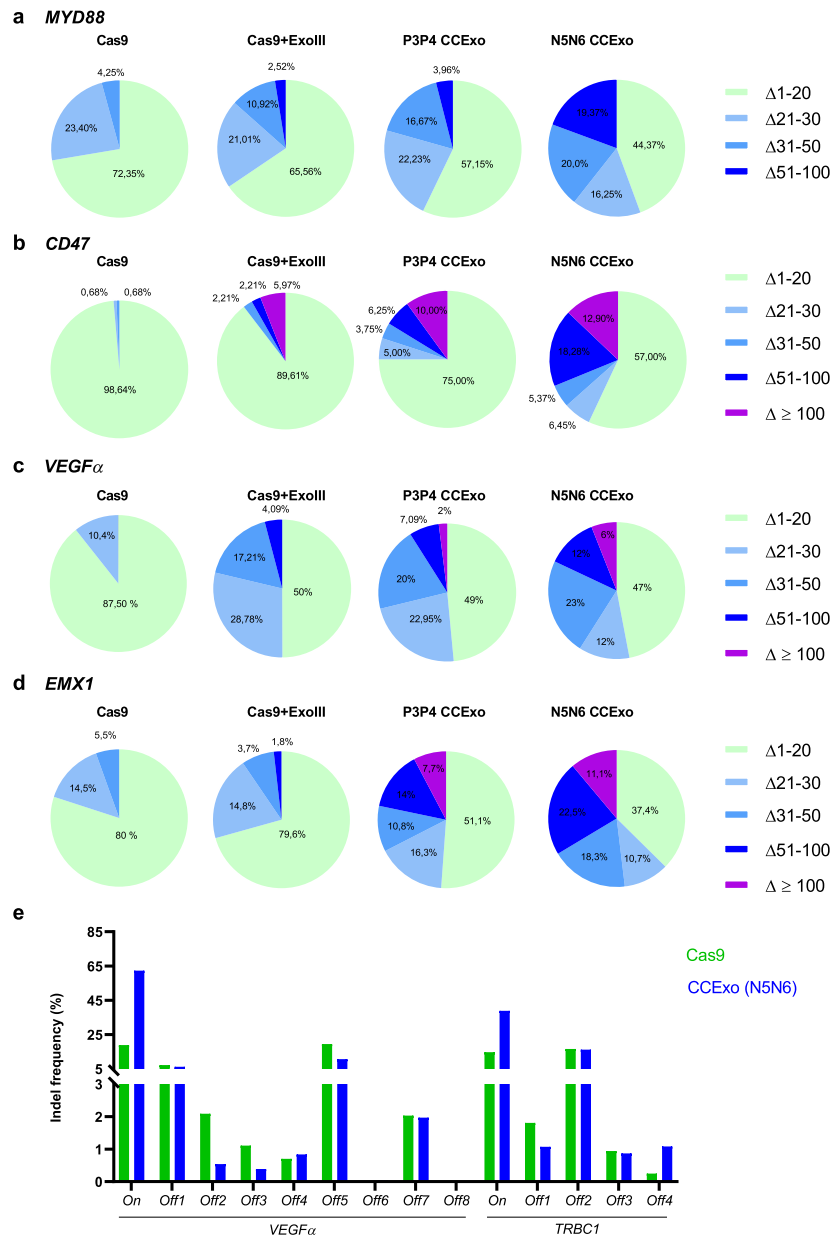
图三 CCExo系统可增加基因敲除片段的长度[7]
最后,研究者将CCExo系统应用于慢性粒细胞白血病(CML)的癌症治疗。CML是一种相对少见的恶性肿瘤,与费城染色体密切相关,是由于BCR和ABL1基因之间的染色体易位引起的(图四a),易位后产生的BCR/ABL1融合基因显著增强了酪氨酸激酶活性,从而驱动癌细胞增殖。研究者将CCExo系统质粒电转导入K562-fLUC-eGFP细胞系,可通过检测fLUC 和eGFP信号强度来测定细胞活力。结果显示,CCExo系统可成功破坏BCR/ABL1融合基因,其中N5N6组可造成约80%的细胞死亡(图四b)。经T7E1分析证实CCExo系统显著增加了插入/缺失比率(图四c)。并且在K562异种移植瘤模型中(图四f),观察到Cas9治疗组的肿瘤尺寸减小,特别是N5N6 CCExo组五分之三的肿瘤被完全根除(图四g和i),小鼠的生存期被显著延长(图四h)。肿瘤组织切片分析显示,N5N6 CCExo组的癌细胞凋亡最为明显(图四j)。此外,研究者将重组蛋白和gRNA组成的RNP复合物电穿孔至CML患者的PBMC中,同样发现N5N6 CCExo组的细胞死亡率以及靶位点的插入/缺失比率最高(图四d和e)。以上数据表明,CCExo系统可用于破坏癌症基因表达,通过显著增强基因敲除效率,来提高癌症治疗效果。
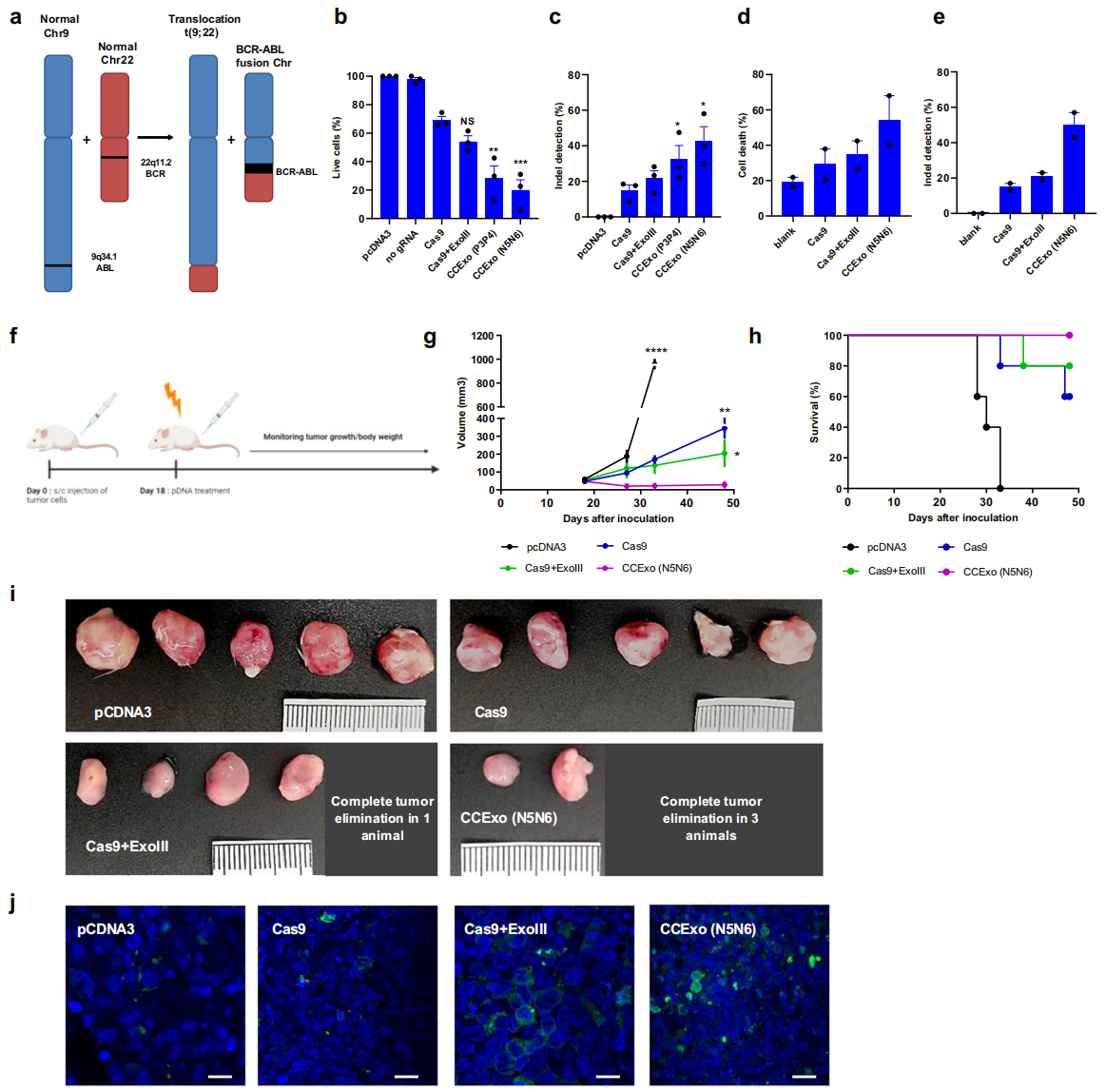
图四 CCExo系统用于破坏癌症基因可增加疗效[7]
综上所述,研究者开发的CCExo系统,利用卷曲螺旋二聚体将核酸外切酶以非共价结合的方式招募至Cas9/gRNA复合物,通过进一步降解切割位点的DNA链,显著增强基因敲除效率。此外,该系统的所有组件可以包装到腺病毒载体中,易于基因递送。后续CCExo系统可以用于治疗CML等其他潜在遗传病因疾病,增强疾病治疗效果,拓宽CRISPR/Cas系统治疗癌症的临床试验范围。

GTP研发中心拥有自主知识产权的“人造精子细胞”介导的半克隆技术,可在体外实现“人造精子细胞”的多位点改造并进行功能元件测试,最后将“人造精子细胞”通过卵母细胞注射可以一步法获得基因改造小鼠。基于“人造精子细胞”单倍体打靶高效率的优势,可快速构建定制化小鼠,如基因组安全位点大片段敲入、条件性基因敲除/敲入和人源化基因改造等。多个复杂调控元件可以通过多次体外细胞打靶构建在同一株“人造精子细胞”中,经细胞水平质控后,一步法获得基因改造小鼠。

如有需要欢迎联系我们,GTP研发中心将竭诚为您设计最佳方案,制定专属于您的基因改造小鼠。
网址:http://www.sibcb.ac.cn/gtp/
地址:上海市岳阳路320号
责任编辑:甜点
新媒体运营:suway
参考文献
1. Pickar-Oliver A, Gersbach CA. The next generation of CRISPR-Cas technologies and applications. Nat Rev Mol Cell Biol 2019, 20(8):490-507.
2. Xie S, Shen B, Zhang C, Huang X, Zhang Y. sgRNAcas9: a software package for designing CRISPR sgRNA and evaluating potential off-target cleavage sites. PLoS One 2014, 9(6):e100448.
3. Kleinstiver BP, Pattanayak V, Prew MS, Tsai SQ, Nguyen NT, Zheng Z, Joung JK. High-fidelity CRISPR-Cas9 nucleases with no detectable genome-wide off-target effects. Nature 2016, 529(7587):490-495.
4. Guilinger JP, Thompson DB, Liu DR. Fusion of catalytically inactive Cas9 to FokI nuclease improves the specificity of genome modification. Nat Biotechnol 2014, 32(6):577-582.
5. Burkhard P, Stetefeld J, Strelkov SV. Coiled coils: a highly versatile protein folding motif. Trends in cell biology 2001, 11(2):82-88.
6. Lebar T, Lainšček D, Merljak E, Aupič J, Jerala R. A tunable orthogonal coiled-coil interaction toolbox for engineering mammalian cells. Nature chemical biology 2020, 16(5):513-519.
7. Lainscek D, Forstneric V, Mikolic V, Malensek S, Pecan P, Bencina M, Sever M, Podgornik H, Jerala R. Coiled-coil heterodimer-based recruitment of an exonuclease to CRISPR/Cas for enhanced gene editing. Nat Commun 2022, 13(1):3604.
8. Hustedt N, Durocher D. The control of DNA repair by the cell cycle. Nat Cell Biol 2016, 19(1):1-9.
9. Gradisar H, Bozic S, Doles T, Vengust D, Hafner-Bratkovic I, Mertelj A, Webb B, Sali A, Klavzar S, Jerala R. Design of a single-chain polypeptide tetrahedron assembled from coiled-coil segments. Nat Chem Biol 2013, 9(6):362-366.



