技术分享:开发识别嘧啶类PAM的Cas9新变体
CRISPR-Cas9技术是一种强大的基因编辑技术,推动了生命科学的发展,并将遗传疾病的新疗法更快地推向临床应用[1]。Cas9对靶点的识别是依赖原间隔序列临近基序(PAM),它通过蛋白质-DNA的相互作用识别PAM,因而特定专属的PAM序列限制了精准基因编辑方法的适用性[2]。为此,科学家们致力于开发筛选不同的Cas蛋白新变体,使其具有更广的或者不同的PAM兼容性。来自化脓性链球菌的SpCas9是目前应用最为广泛的Cas9[3],基于SpCas9的改造变体已有许多报道,但主要适用于嘌呤类PAM,对于嘧啶类PAM仍有不足。与SpCas9相比,大多数Cas同源物在哺乳动物细胞中的活性较低,或对PAM的要求较高,因而针对非SpCas9同源物的筛选进化并不常见[4]。Nme2Cas9是一种来自脑膜炎奈瑟菌的Cas9同源物,它识别N4CC PAM,在哺乳动物细胞中也显示出较高的活性[5]。而且相对于SpCas9的1368个氨基酸,Nme2Cas9只有1082个氨基酸,后续临床应用更为方便。因此,Nme2Cas9是筛选进化嘧啶类PAM的较优候选对象。
2022年9月,Nature Biotechnology杂志在线发表了针对Nme2Cas9的定向进化,借助升级后的SAC-PACE、ePACE和BE-PPA筛选系统,筛选出四种识别嘧啶类PAM的变体,可在含有单个特定嘧啶核苷酸的PAM上实现精确的基因组编辑。其中变体eNme2-T.1和eNme2-T.2可识别N4TN PAM,且编辑活性与现有变体相当;变体eNme2-C和eNme2-C.NR提供限制性更少的PAM要求,可识别N4CN PAM序列,且在多种人源细胞中展现出相当或者更高的编辑活性[6]。
研究者之前已利用噬菌体辅助连续演化系统(PACE)筛选出识别NR PAM的SpCas9变体,但是该系统对于筛选嘧啶类PAM变体的成功率较低[7](图一a左)。因此,研究者将DNA结合选择和下游功能的碱基编辑选择[8]结合起来,开发了升级版的SAC-PACE系统(图一a右)。该系统利用内含肽的自我剪接能力来筛选出正确碱基编辑后,促使噬菌体得以存活增殖从而筛选Cas变体。其中,筛选噬菌体(SP)编码了腺苷脱氨酶(TadA)与Cas变体的融合蛋白。另外,在宿主大肠杆菌的辅助质粒(AP)上,将内含肽Npu插入到glll基因的Leu 10位点的后面,并且Npu被linker序列分隔成N端和C端两个部分,其中linker序列上包含了特定PAM序列以及含有终止密码子的原间隔序列(图一b)。当宿主大肠杆菌被ΔgIII-噬菌体感染后,Cas变体通过识别特定PAM序列,引导TadA完成终止密码子的修复,促使大肠杆菌恢复表达gIII基因,使得ΔgIII-噬菌体恢复增殖能力。研究者将dNme2Cas9与TadA8e融合(Nme2-ABE8e)来测试SAC-PACE系统,过夜培养结果显示,相比对照组,Nme2-ABE8e实验组的噬菌体富集了102-106倍,且富集程度取决于gIII的表达水平(图一c)。这表明SAC-PACE这一灵活高效的筛选系统设计成功,可用于后续筛选拓展PAM范围的Cas变体。此外,研究者开发了ePACE平台,将PACE与连续培养平台eVOLVER相结合,实现高通量连续定向进化筛选,可同时筛选8种PAM的Cas变体(图一d)。还开发了一种基于碱基编辑依赖性PAM分析法BE-PPA,用于快速灵敏地分析进化过程中出现的Nme2Cas9变体的PAM范围。利用上述新型筛选系统和分析方法,研究者对野生型Nme2Cas9识别的PAM进行初步测定,发现Nme2-ABE8e对PAM的偏好性依次为N3NCG<N3NCA<N3NCT<N3NCC,并且对N3NTC也有很好的编辑活性(图一e)。
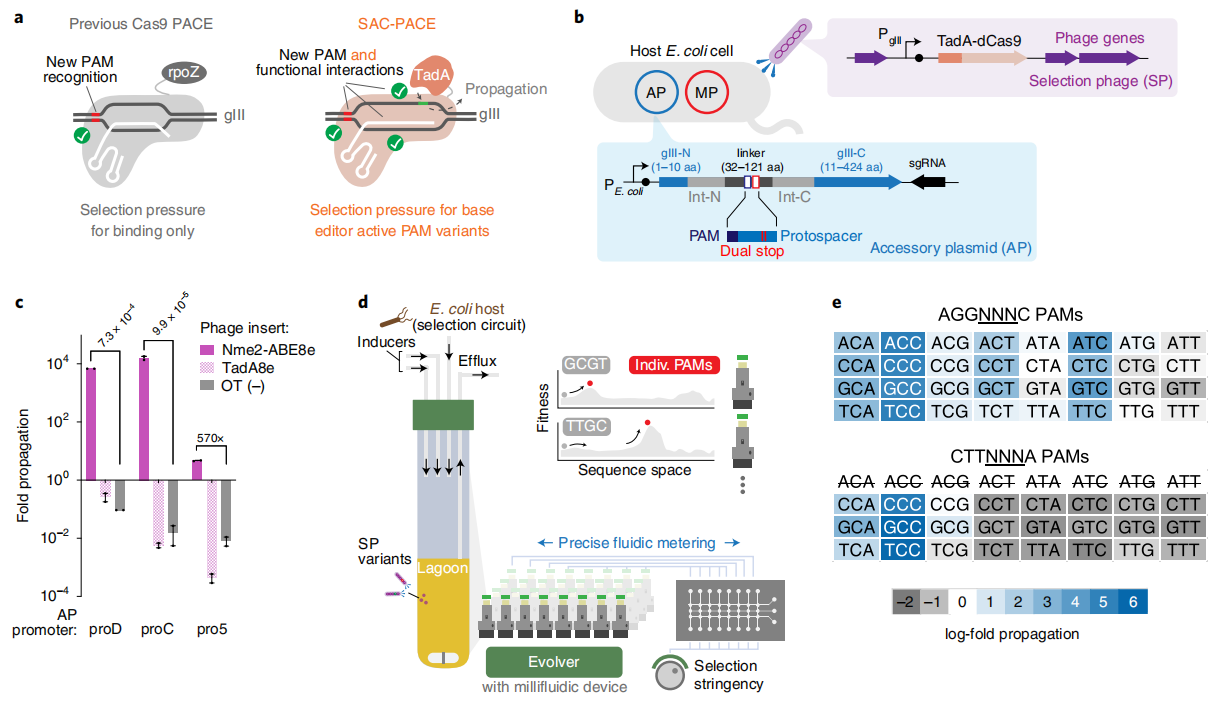
图一 开发Cas9筛选系统和ePACE平台用于高通量连续定向进化筛选[6]
于是,研究者使用野生型Nme2Cas9作为进化起点,利用低严格性的SAC-PACE筛选变体(图二a左),但难以获得高编辑活性的Nme2Cas9变体。随后利用高严格性的分割型SAC-PACE(图二a中)和多重PAM的分割型SAC-PACE系统(图二a右)进化出识别N4CN PAM的变体eNme2-C以及识别N4TN PAM的变体eNme2-T.1和eNme2-T.2(图二b)。eNme2-C-ABE8e在所有N4CN PAM位点上实现了 ≥80% 的A•T-to-G•C编辑,在8个内源性人基因组N4CN位点上显示出强大的编辑活性(图二c-e)。eNme2-T.1-ABE8e和eNme2-T.2-ABE8e在所有N4TN PAM中实现约大于70% 的A•T-to-G•C编辑,相比野生型Nme2-ABE8e,在HEK293T细胞中的编辑活性分别提高了278倍和264倍(图二c,f和g)。以上数据表明,进化筛选出的识别N4TN和N4CN PAM的Nme2Cas9变体扩展了PAM范围,同时具有高效的基因编辑活性。
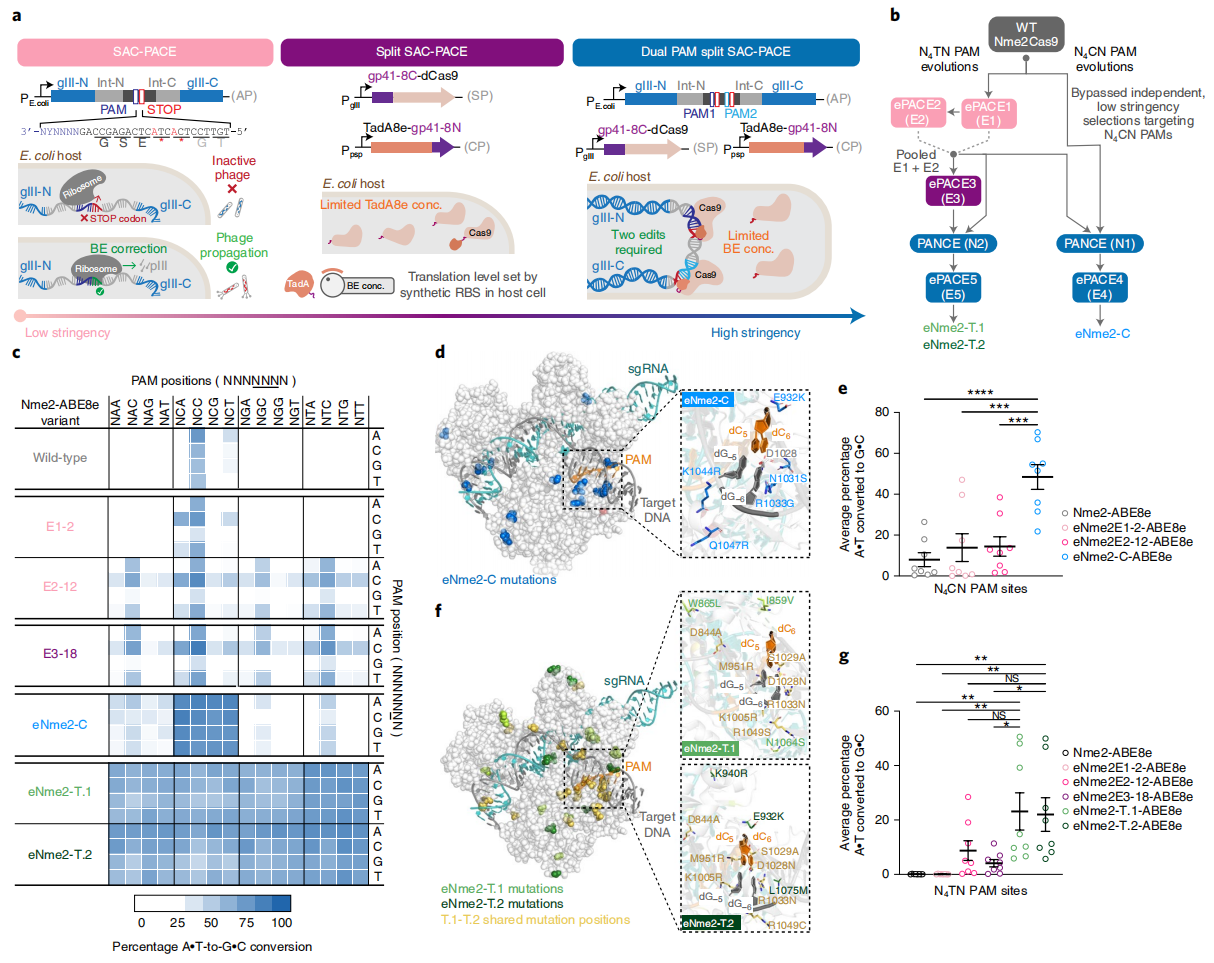
图二 进化获得PAM范围拓展的Nme2Cas9变体[6]
然后,研究者比较了进化的eNme2变体与SpRY的碱基编辑活性(图三a)。在HEK293T细胞中的14个匹配含C的PAM位点,eNme2-C-ABE8e的A•T-to-G•C编辑活性显著高于SpRY-ABE8e和SpRY-HF1-ABE8e(图三b),但在8个匹配含T的PAM位点上,eNme2-T.1-ABE8e和eNme2-T.2-ABE8e的编辑活性低于对照组(图三c)。研究者还证实,eNme2-C衍生的CBE工具eNme2-C-BE4的C•G-to-T•A编辑活性同样优于SpRY-BE4和SpRY-HF1-BE4(图三d)。当RuvC D16A失活突变时,eNme2-C的核酸酶活性会受到抑制,若恢复突变,产生的变体eNme2-C.NR恢复了核酸酶活性,同时保留了识别N4CN PAM位点的能力,其NCN PAM的编辑活性也显著高于SpRY和SpRY-HF1核酸酶(图三e)。以上数据表明,eNme2-C碱基编辑器和eNme2-C.NR核酸酶作为基因编辑的高效变体,为含C PAM位点的编辑提供了最佳选择替换,而eNme2-T.1和eNme2-T.2为含T PAM位点的编辑提供了新的选择。
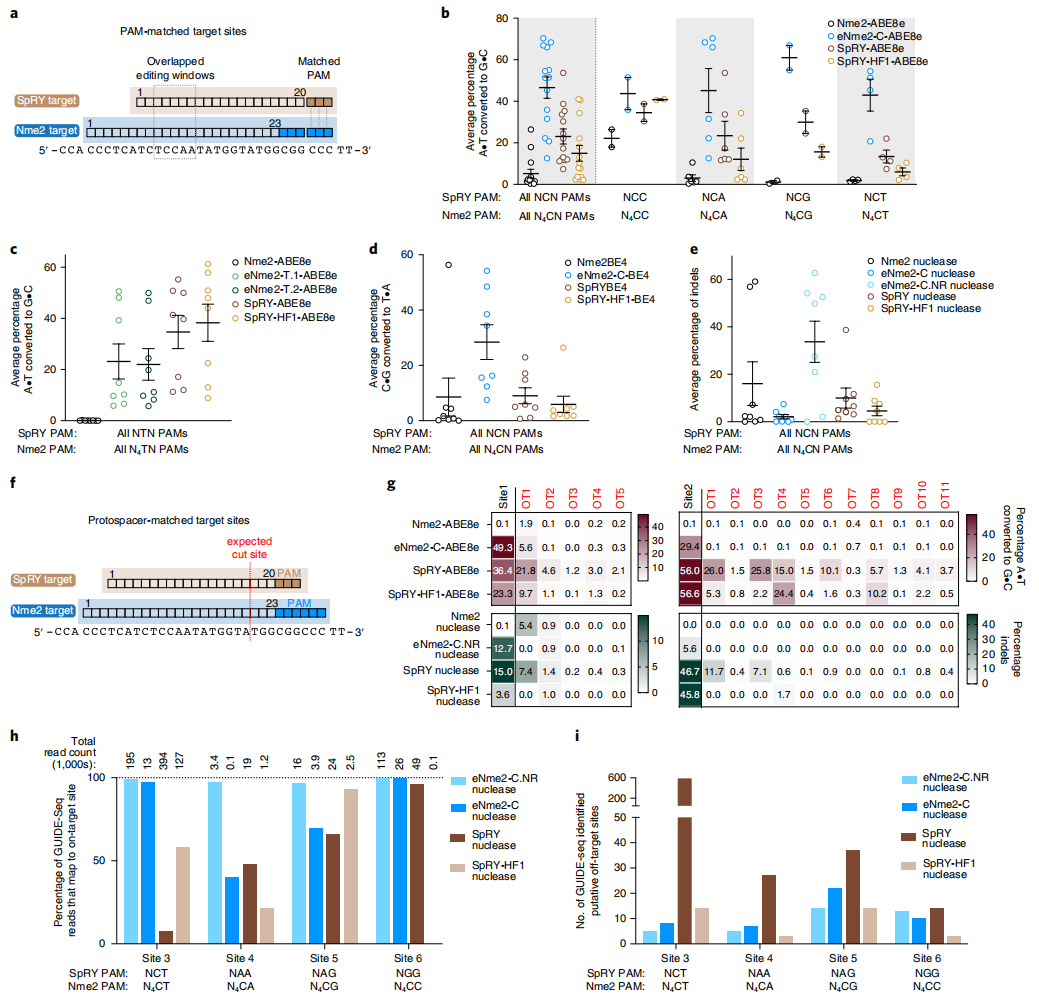
图三 Nme2Cas9变体在哺乳动物细胞中的表征[6]
接下来,研究者选择两个原间隔序列匹配位点来评价脱靶活性(图三f)。对于位点1,eNme2-C.NR没有生成任何大于1%的插入/缺失;位点2也观察到类似结果,eNme2-C-ABE8e或eNme2-C.NR也没有观察到大于1%的插入/缺失(图三g)。全基因组测序结果也显示了eNme2-C.NR具有高靶向(图三h)和低脱靶活性(图三i),整体表现为高特异性。以上数据表明,eNme2-C和eNme2-C.NR保留了Nme2Cas9的高天然特异性。
最后,研究者在多种细胞类型中验证eNme2-C在含有N4CN PAM靶位点上的高效性和特异性。在永生化肝细胞系HUH7中,eNme2-C-ABE8e在含有N4CN PAM的15个测试位点约完成37%的A•T-to-G•C的编辑活性,高于对照组SpRY-ABE8e和SpRY-HF1-ABE8e(图四a)。同样,在U2OS细胞的18个位点也都观察到高于对照的A•T-to-G•C碱基编辑(图四b)。使用eNme2-C-ABE8e mRNA核转染人真皮成纤维细胞,在7个内源位点上实现了64%的A•T-to-G•C碱基编辑(图四c)。
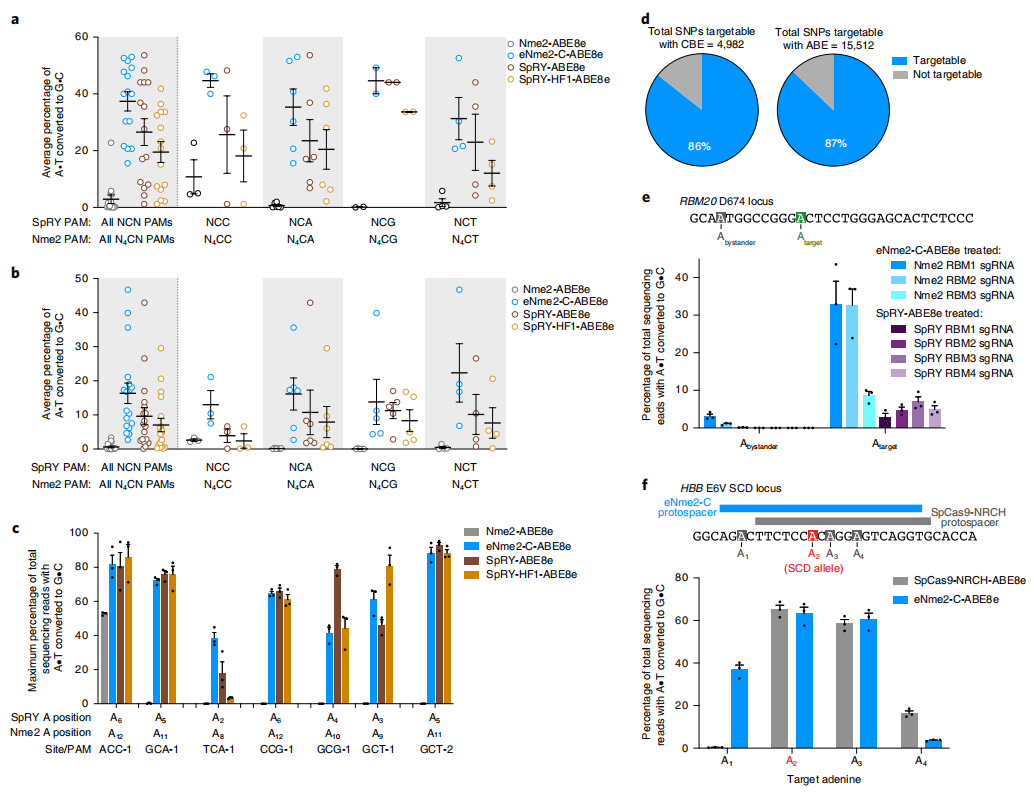
图四 eNme2-C-ABE8e在不同细胞类型和靶基因的普遍高效编辑[6]
作为胞嘧啶或腺嘌呤碱基编辑器,eNme2-C能够分别靶向86%和87%的疾病相关SNPs(图四d)。扩张型心肌病(DCM)是一种原因未明的原发性心肌疾病,2%-3%的家族性DCM病例中均可观察到RBM20基因突变[9]。研究者使用eNme2-C-ABE8e完成了RBM20基因D674G的转变,测试了3个sgRNA都实现特异性靶向编辑腺嘌呤碱基,最高可达33%的A•T-to-G•C的编辑活性(图四e)。此外,eNme2-C-ABE8e还能有效地修复镰状细胞贫血病的点突变,对靶腺嘌呤的编辑效率达63%,与对照组相当(图四f)。以上数据表明,eNme2-C是一种广泛适用的Cas蛋白,能够在多种生物学相关细胞类型中实现精准基因组编辑。eNme2-C不仅扩大了碱基编辑器的靶向范围,还为现有的Cas9变体提供了更多位点特异的替代方案。
综上所述,研究者将SAC-PACE、ePACE和BE-PPA相结合开发出新型蛋白定向进化筛选系统,成功筛选出PAM序列拓展型的Nme2Cas9变体,从野生型的N4CC PAM序列拓展至N4CN和N4TN。高活性的Nme2Cas9变体可特异性识别嘧啶类PAM序列的基因组位点,为精准基因编辑提供了全新的工具,极大地拓展了其应用范围。

GTP研发中心拥有自主知识产权的“人造精子细胞”介导的半克隆技术,可在体外实现“人造精子细胞”的多位点改造并进行功能元件测试,最后将“人造精子细胞”通过卵母细胞注射可以一步法获得基因改造小鼠。基于“人造精子细胞”单倍体打靶高效率的优势,可快速构建定制化小鼠,如基因组安全位点大片段敲入、条件性基因敲除/敲入和人源化基因改造等。多个复杂调控元件可以通过多次体外细胞打靶构建在同一株“人造精子细胞”中,经细胞水平质控后,一步法获得基因改造小鼠。
 如有需要欢迎联系我们,GTP研发中心将竭诚为您设计最佳方案,制定专属于您的基因改造小鼠。
如有需要欢迎联系我们,GTP研发中心将竭诚为您设计最佳方案,制定专属于您的基因改造小鼠。
网址:http://www.sibcb.ac.cn/gtp/
邮箱:gtp_order@sibcb.ac.cn
地址:上海市岳阳路320号
责任编辑:甜点
新媒体运营:suway
参考文献
1. Cong L, Ran FA, Cox D, Lin SL, Barretto R, Habib N, Hsu PD, Wu XB, Jiang WY, Marraffini LA et al. Multiplex Genome Engineering Using CRISPR/Cas Systems. Science 2013, 339(6121):819-823.
2. Huang TP, Newby GA, Liu DR. Precision genome editing using cytosine and adenine base editors in mammalian cells. Nat Protoc 2021, 16(2):1089-1128.
3. Walton RT, Christie KA, Whittaker MN, Kleinstiver BP. Unconstrained genome targeting with near-PAMless engineered CRISPR-Cas9 variants. Science 2020, 368(6488):290-296.
4. Xu XS, Chemparathy A, Zeng LP, Kempton HR, Shang S, Nakamura M, Qi LS. Engineered miniature CRISPR-Cas system for mammalian genome regulation and editing. Molecular cell 2021, 81(20):4333-4345.
5. Edraki A, Mir A, Ibraheim R, Gainetdinov I, Yoon Y, Song CQ, Cao YY, Gallant J, Xue W, Rivera-Perez JA et al. A Compact, High-Accuracy Cas9 with a Dinucleotide PAM for In Vivo Genome Editing. Molecular cell 2019, 73(4):714-726.
6. Huang TP, Heins ZJ, Miller SM, Wong BG, Balivada PA, Wang T, Khalil AS, Liu DR. High-throughput continuous evolution of compact Cas9 variants targeting single-nucleotide-pyrimidine PAMs. Nature biotechnology 2022.
7. Miller SM, Wang T, Randolph PB, Arbab M, Shen MW, Huang TP, Matuszek Z, Newby GA, Rees HA, Liu DR. Continuous evolution of SpCas9 variants compatible with non-G PAMs. Nature biotechnology 2020, 38(4):471-481.
8. Thuronyi BW, Koblan LW, Levy JM, Yeh WH, Zheng C, Newby GA, Wilson C, Bhaumik M, Shubina-Oleinik O, Holt JR et al. Continuous evolution of base editors with expanded target compatibility and improved activity. Nature biotechnology 2019, 37(9):1070-1079.
9. Lennermann D, Backs J, van den Hoogenhof MMG. New Insights in RBM20 Cardiomyopathy. Current heart failure reports 2020, 17(5):234-246.



