技术分享:转录偶联供体DNA可显著提高基因编辑效率
CRISPR/Cas9诱导DNA双链断裂(DSB)进而刺激同源重组(HR)修复实现基因组的精准编辑。然而,这种方法在哺乳动物细胞中精准插入或敲除大片段DNA时是低效的,因为HR修复途径效率远远低于细胞中普遍存在的非同源末端连接(NHEJ)修复途径[1, 2]。提高基因编辑效率的方法有一种是利用转录,该方法可增强HR修复。有研究表明,供体DNA的活跃转录会显著增加基因校正频率[3]。这是因为DSB周围的转录会形成包含DNA-DNA和DNA-RNA混合物的DR-loops[4]。当DSB末端被EXO1核酸酶加工成单链DNA (ssDNA)末端时,其3’突出ssDNA与新转录的RNA可短暂形成一个R-loop[6],而这种R-loop结构会优先招募HR蛋白,促使DSB优先采用HR途径来进行修复[7, 8]。
2022年8月, Nucleic Acids Research上报道了一种转录偶联编辑器TEd,通过转录偶联的供体DNA(TC供体)实现高效的基因编辑,并且将转录的TC供体招募到DSB处可以进一步提高基因编辑效率。在哺乳动物细胞系中,TEd相比经典Cas9介导的HR编辑方法CEd,在实现大片段DNA的敲除、插入、替换以及点突变等基因编辑中效率提高约10倍以上[9]。
首先,研究者测试将HR供体与转录偶联能否提高CRISPR/Cas9介导的基因编辑效率。分别将H1启动子克隆到HR供体质粒同源臂(HA)的左侧或右侧,获得两个转录方向相反的TC供体。其中,一个供体产生与Cas9/gRNA非靶链互补的同源转录偶联RNA (TC RNA),称为TEd/Ph(图一A);另一个TC RNA与靶链互补,称为TEd/Ph-Rev。通过将GFP序列插入到LMNA基因的sgRNA1位点来比较TEd和CEd的编辑效率和准确性(图一B和C)。经FACS分析发现,TEd1/Ph和TEd2/Ph的GFP KI效率显著高于CEd约3-4倍(图一D),定位于核膜的GFP-Lamin融合蛋白也被成功表达(图一E)。然而,TEd1/Ph-Rev或TEd2/Ph-Rev的GFP KI效率均不高于CEd(图一D),表明TC供体的转录方向是提高GFP KI效率的关键。此外,研究者测试了由CMV启动子驱动的TEd效率,称为TEd/Pc,其KI效率高于H1启动子(图一D),表明转录活性更高的CMV启动子提高了TC供体的转录水平,进而提高了KI效率。以上结果表明,链特异性TC供体转录在LMNA基因的GFP KI中提高了至少3倍的效率,并且TC供体转录的方向是TEd进行高效基因编辑的关键。
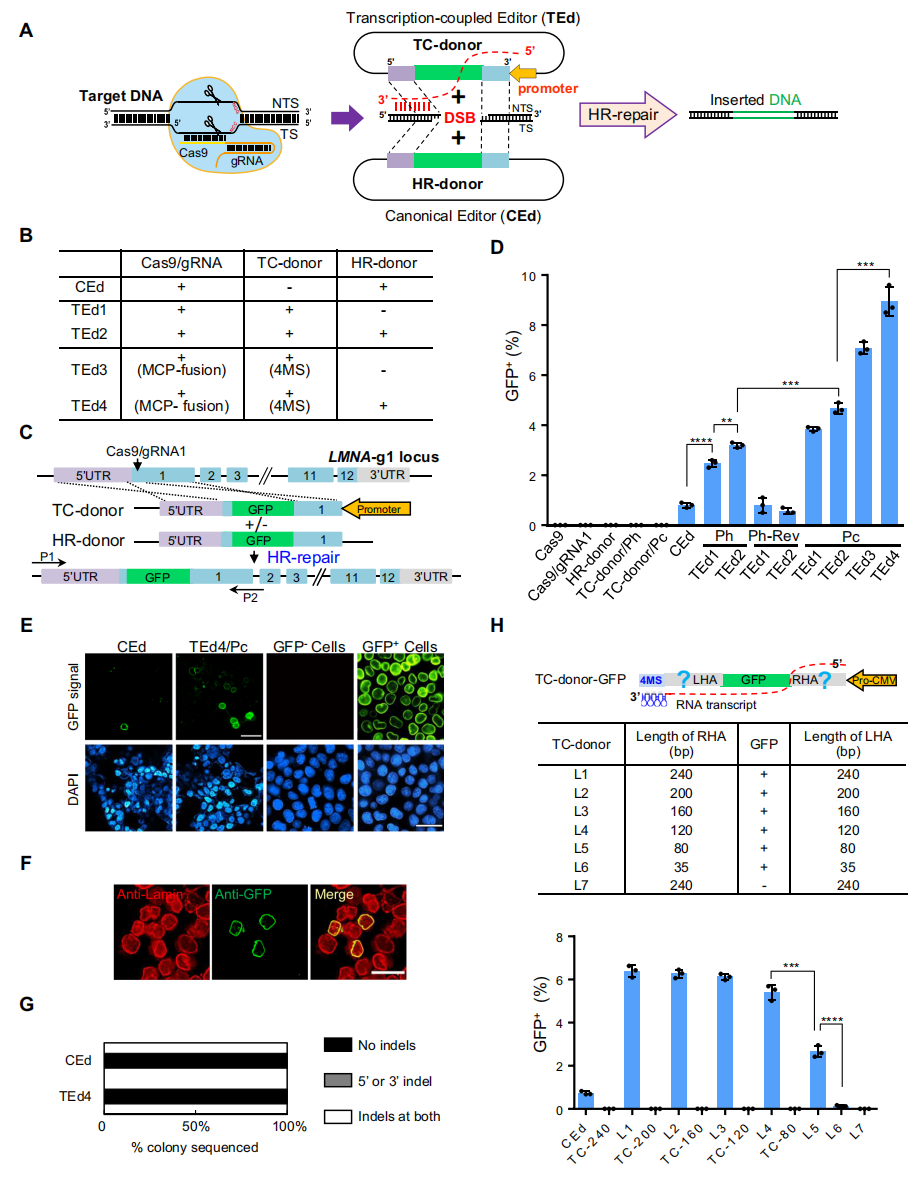
图一 TEd基因编辑方式在哺乳动物细胞中实现高效基因敲入[9]
接下来,研究者测试TEd的基因编辑效率是否受到TC供体和DSB之间距离的限制,利用MCP-MS2系统直接将TC RNA招募到DSB处来检测对KI效率的影响。将4个噬菌体MS2插入TEd1和TEd2的TC RNA的3’端,分别获得TEd3和TEd4(图一B),并将MCP(MS2衣壳蛋白)融合到Cas9的N端或C端,检测这些结构组合在LMNA基因位点的GFP KI效率。结果显示TEd3和TEd4的KI效率显著高于CEd(约10倍)和TEd2(约2倍)(图一D),C端标记的MCP效率略高于N端。通过GFP的核膜定位,以及基因组PCR和TA克隆测序结果证实TEd3、TEd4和CEd均可精准编辑细胞(图一E-G),免疫印迹分析也检测到预期大小的蛋白表达。以上结果表明,将TC RNA招募到DSB处可显著提高TEd编辑效率。此外,研究者还测试了TC供体的同源臂HA长度对HR效率的影响。结果发现,HA长度120bp的KI效率与240bp几乎相同,而小于120bp的则显示出较低的编辑效率(图一H)。以上结果表明,越长的HA可驱动越高的编辑效率,并且120bp足以实现高效的TEd编辑。
目前基因编辑面临的挑战之一是实现高效、精准地插入大片段DNA。研究者分别在HEK293T细胞的GAPDH位点和FBL位点上测试TEd KI不同长度大片段DNA(2.7~10.1kb)的效率(图二A)。结果发现,TEd4在两个位点的大片段DNA KI效率均高于CEd(图二B和C),PCR检测和GFP融合蛋白的细胞核定位确认了KI的准确性(图二D-G)。以上结果表明,对于大片段DNA,TEd相比CEd同样能够实现更加高效、精准的最多10 kb大片段DNA的基因敲入。
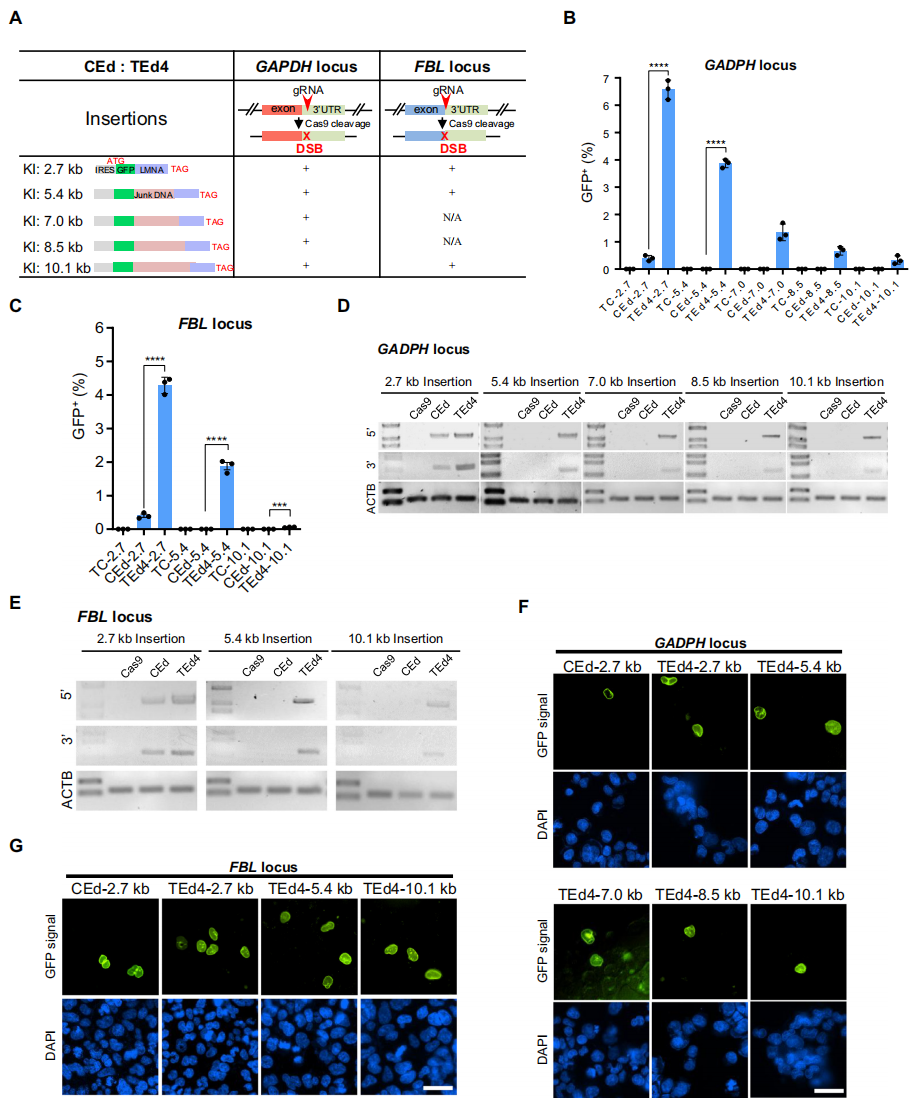
图二 TEd在HEK293T中实现高效的大片段DNA基因敲入[9]
研究者进一步测试TEd4的大片段DNA敲除效率,在HEK293T细胞中比较TEd4和CEd方法敲除10kb CDC42片段的效率(图三A),PCR及测序结果显示该片段被精准敲除,且TEd4的敲除效率高于CEd(图三B)。另外,用18bp的I-SceI位点替换8kb的基因组序列(图三C),同样显示TEd4敲除效率比CEd更高效(图三D),酶切验证I-SceI的准确替换(图三E),并且I-SceI连接处没有发现序列的插入/缺失(图三F)。同样地,用内部核糖体进入位点(IRES)-GFP片段替换8kb的基因组序列(图三G),结果也显示TEd4的基因替换效率显著高于CEd(图三H和I),以及IRES-GFP序列的准确替换(图三J)。以上结果证明,TEd相比CEd在大片段DNA的基因敲除和基因替换上同样更为高效。
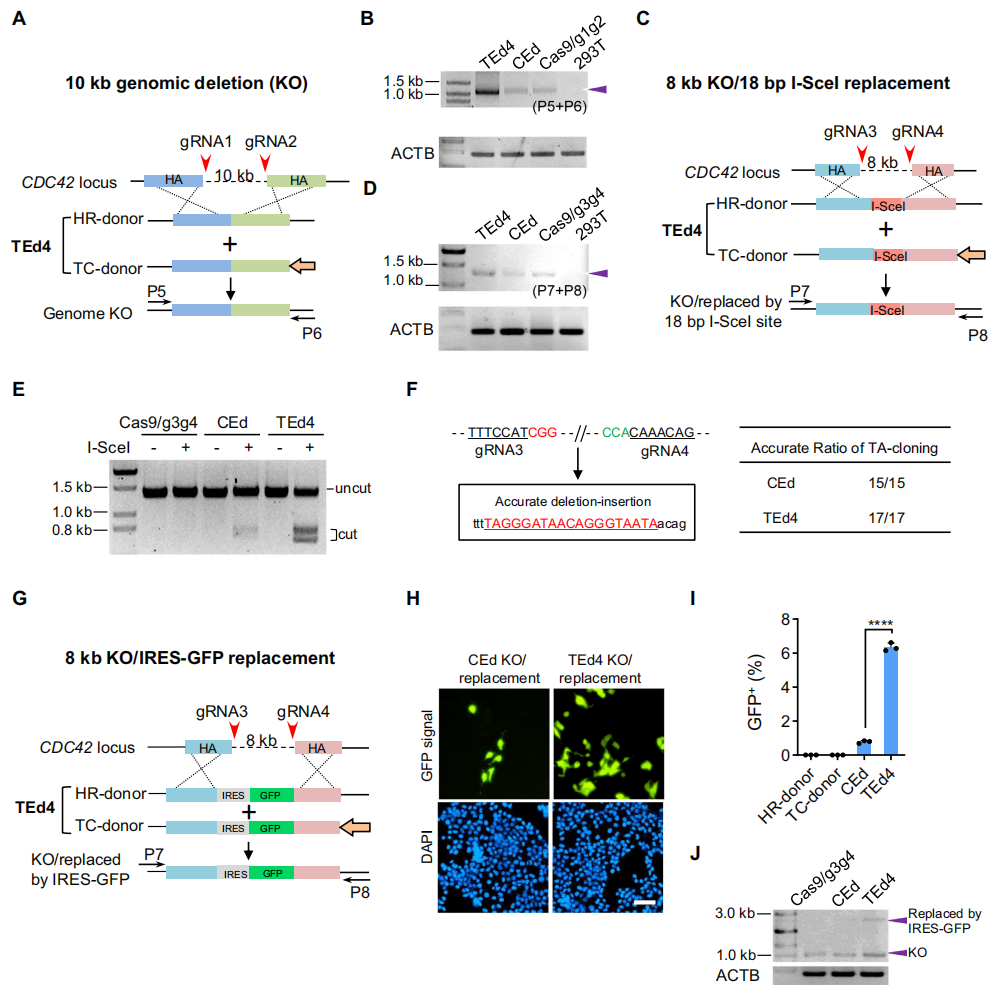
图三 TEd在HEK293T中实现高效大片段DNA的基因敲除和基因替换[9]
最后,研究者比较了TEd和CEd引入点突变的效率。设计了linker序列将mCherry与GFP融合在一起,其中linker序列中包含了一个TAG终止密码子,因而构建的mCherry–linker-GFP报告系统只表达mCherry(图四A)。当利用CRISPR系统将TAG突变为TTG,且AGG的PAM序列突变为AGT后,即可同时表达mCherry和GFP,因此可利用该报告系统评价引入点突变的效率。FACS分析发现TEd4的点突变KI效率是CEd的15倍(图四B),荧光成像显示mCherry和GFP的共表达(图四C),Sanger测序和NGS均验证了点突变KI的准确性(图四D-E)。此外,研究者还在疾病相关位点PRNP引入G127V突变(图四F),结果显示TEd4的点突变KI效率是CEd的10倍(图四G-H)。以上结果表明,TEd能高效、精准地在基因组中引入点突变。
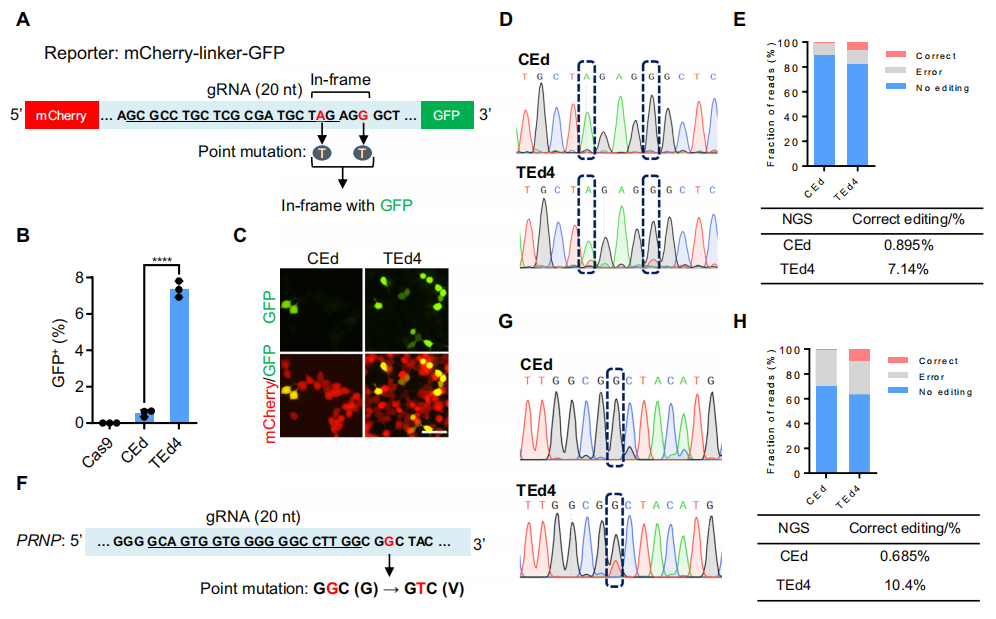
图四 TEd在HEK293T中实现高效的点突变[9]
综上所述,研究者开发的转录偶联基因编辑器TEd,通过对HR供体DNA进行改造,在HR供体DNA上引入活跃的转录并招募到DSB处,可大大提高CRISPR/Cas9介导的同源重组效率,实现哺乳动物细胞内大片段DNA的高效精准敲入、敲除、替换以及点突变,显著提高基因编辑在基础科研和临床转化应用中的编辑效率。

GTP研发中心拥有自主知识产权的“人造精子细胞”介导的半克隆技术,可在体外实现“人造精子细胞”的多位点改造并进行功能元件测试,最后将“人造精子细胞”通过卵母细胞注射可以一步法获得基因改造小鼠。基于“人造精子细胞”单倍体打靶高效率的优势,可快速构建定制化小鼠,如基因组安全位点大片段敲入、条件性基因敲除/敲入和人源化基因改造等。多个复杂调控元件可以通过多次体外细胞打靶构建在同一株“人造精子细胞”中,经细胞水平质控后,一步法获得基因改造小鼠。

如有需要欢迎联系我们,GTP研发中心将竭诚为您设计最佳方案,制定专属于您的基因改造小鼠。
网址:http://www.sibcb.ac.cn/gtp/
地址:上海市岳阳路320号
责任编辑:甜点
新媒体运营:suway
参考文献
1. Chu VT, Weber T, Wefers B, Wurst W, Sander S, Rajewsky K, Kuhn R. Increasing the efficiency of homology-directed repair for CRISPR-Cas9-induced precise gene editing in mammalian cells. Nat Biotechnol 2015, 33(5):543-548.
2. Mao Z, Bozzella M, Seluanov A, Gorbunova V. Comparison of nonhomologous end joining and homologous recombination in human cells. DNA Repair (Amst) 2008, 7(10):1765-1771.
3. Humbert O, Maizels N. Epigenetic modification of the repair donor regulates targeted gene correction. Mol Ther Nucleic Acids 2012, 1:e49.
4. Ouyang J, Yadav T, Zhang JM, Yang H, Rheinbay E, Guo H, Haber DA, Lan L, Zou L. RNA transcripts stimulate homologous recombination by forming DR-loops. Nature 2021, 594(7862):283-288.
5. Ohle C, Tesorero R, Schermann G, Dobrev N, Sinning I, Fischer T. Transient RNA-DNA Hybrids Are Required for Efficient Double-Strand Break Repair. Cell 2016, 167(4):1001-1013.e1007.
6. Liu S, Hua Y, Wang J, Li L, Yuan J, Zhang B, Wang Z, Ji J, Kong D. RNA polymerase III is required for the repair of DNA double-strand breaks by homologous recombination. Cell 2021, 184(5):1314-1329.e1310.
7. Daley JM, Niu H, Miller AS, Sung P. Biochemical mechanism of DSB end resection and its regulation. DNA repair 2015, 32:66-74.
8. Renkawitz J, Lademann CA, Jentsch S. Mechanisms and principles of homology search during recombination. Nature reviews Molecular cell biology 2014, 15(6):369-383.
9. Gao K, Zhang X, Zhang Z, Wu X, Guo Y, Fu P, Sun A, Peng J, Zheng J, Yu P et al. Transcription-coupled donor DNA expression increases homologous recombination for efficient genome editing. Nucleic Acids Res 2022, Aug 5:gkac676.



