技术分享:基于Cas12a的病毒分子诊断新方法sPAMC
近几十年来,许多病毒引起了大规模爆发流行性疾病,如严重急性呼吸系统综合征冠状病毒(SARS-CoV)[1],中东呼吸综合征冠状病毒(MERS-CoV)[2],艾滋病毒(HIV)[3]以及当前全球爆发的新型冠状病毒(SARS-CoV-2,COVID-19)[4]等。及时有效地发现患者并给予治疗是遏制流行性病毒传播的有效手段。
逆转录实时定量PCR(RT-qPCR)是应用最为广泛的病毒检测“金标准”。然而,检测耗时较长且依赖于昂贵的设备限制了其检测速率[5]。由重组酶聚合酶扩增技术(RPA)[6]和环介导等温扩增技术(LAMP)[7]介导的快速恒温DNA扩增反应,由于不需要特殊温控设备,可以真正实现便携式的快速核酸检测。基于RPA或者LAMP技术与CRISPR/Cas系统相结合,加快推进了下一代分子诊断技术的发展。例如新一代核酸检测方法SHERLOCK[8]和DETECTR[9],已被开发用于新冠病毒的临床检测。但是这些方法需要分两步进行,先进行目标序列的RPA或LAMP扩增,再进行CRISPR/Cas的检测,而多个处理步骤增加了检测时间和交叉污染的风险[10, 11]。进而,SHERLOCK被开发为单管法STOPCovid[12]和一步法SHINE[13]来简化步骤,但是灵敏度仍低于RT-qPCR,并且总反应时间约为1小时。因此,探索快速、敏感、便捷的分子诊断方法对于抗击病毒的传播至关重要。
2022年3月,Nature Biomedical Engineering上报道了一种基于Cas12a的病毒分子诊断新方法sPAMC(for suboptimal PAM of Cas12a-based test with enhanced flexibility, speed, sensitivity and reproducibility)[14]。sPAMC单管法利用非典型次优PAM基序来介导Cas12a的激活与荧光探针的切割,通过平衡Cas12a的顺式切割与旁侧切割的活性动力学,使得检测新冠病毒的反应时间缩减为20分钟,敏感性为94.2%,特异性为100%。并且sPAMC单管法相比RT-qPCR,无需提取RNA和特殊温控仪器,灵敏度也相当,为1cp/μl。
研究者在改进基于RPA和Cas12a的单管法检测新冠病毒的过程中,意外地发现针对一些非典型PAM基序设计的Cas12a-crRNA在单管法反应中灵敏度显著高于典型PAM基序(TTTV)。研究者将同个靶序列的PAM基序进行突变,发现虽然Cas12a对典型PAM基序TTTG的旁侧切割活性高于非典型PAM基序GTTG(图一a),但是在单管法反应中非典型的GTTG信号更优(图一b)。另外,在单管法反应的灵敏度测试中,发现非典型的GTTG可检测到最低靶序列浓度为2.34 fM(图一c),而典型的TTTG最低仅能检测到234 fM(图一d)。因此,非典型的PAM基序可将检测效率提高约100倍,并大大缩短反应时间。
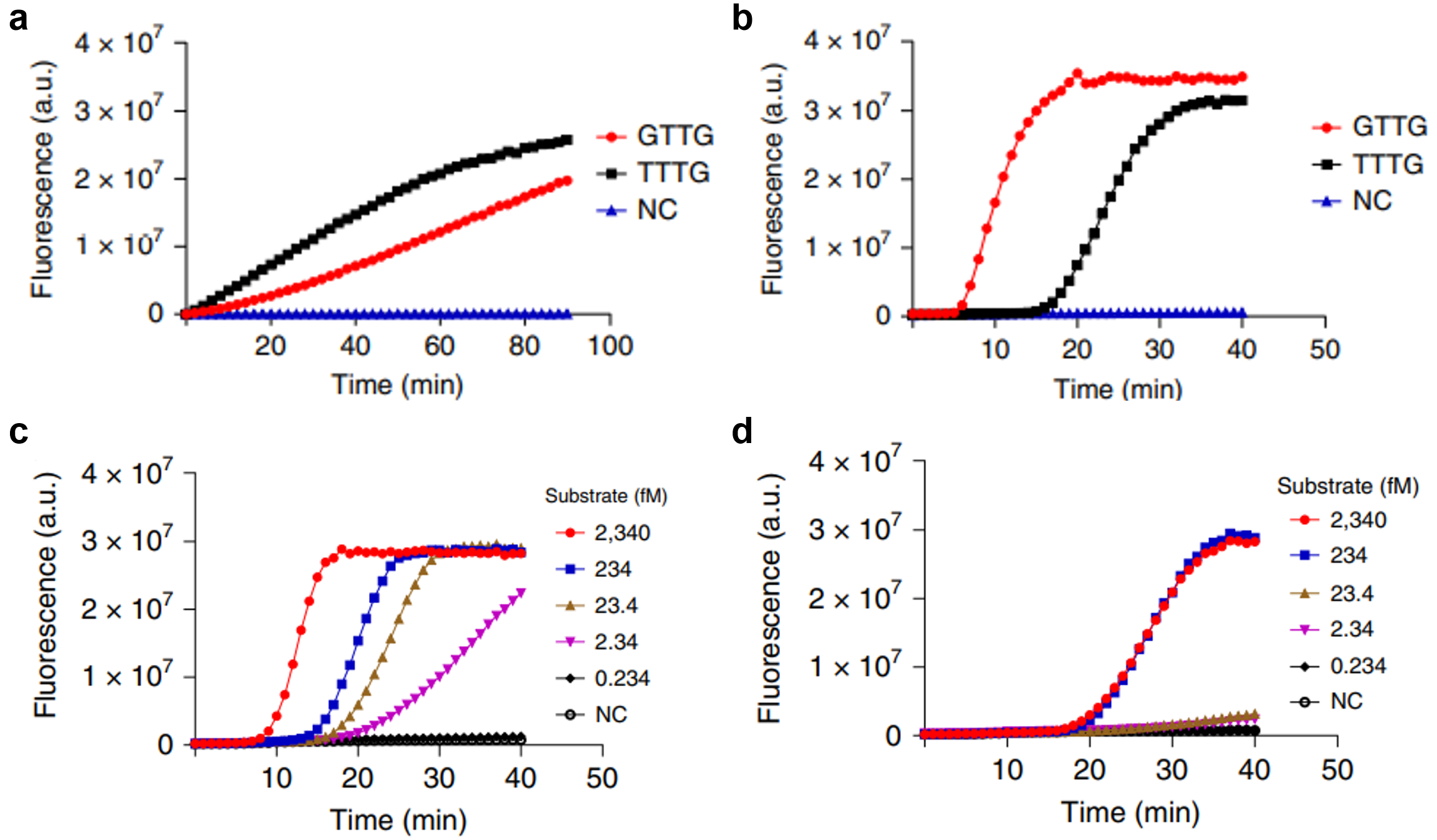
图一 非典型PAM基序在单管法反应中显著优于典型PAM基序[14]
接着,研究者发现采用非典型PAM基序GTTG的单管法反应可获得与单独RPA反应一致的靶序列DNA扩增量,而典型TTTG的靶序列DNA扩增缓慢(图二a)。另外,典型TTTG可介导Cas12a更快速的靶序列顺式切割(图二b),以及更强的靶序列结合能力(图二c)。结果表明,靶序列的RPA扩增与Cas12a的顺式切割存在相互竞争。最终的检测信号可依靠靶序列扩增产生足够的底物,用于Cas12a的激活以及后续旁侧切割活性。
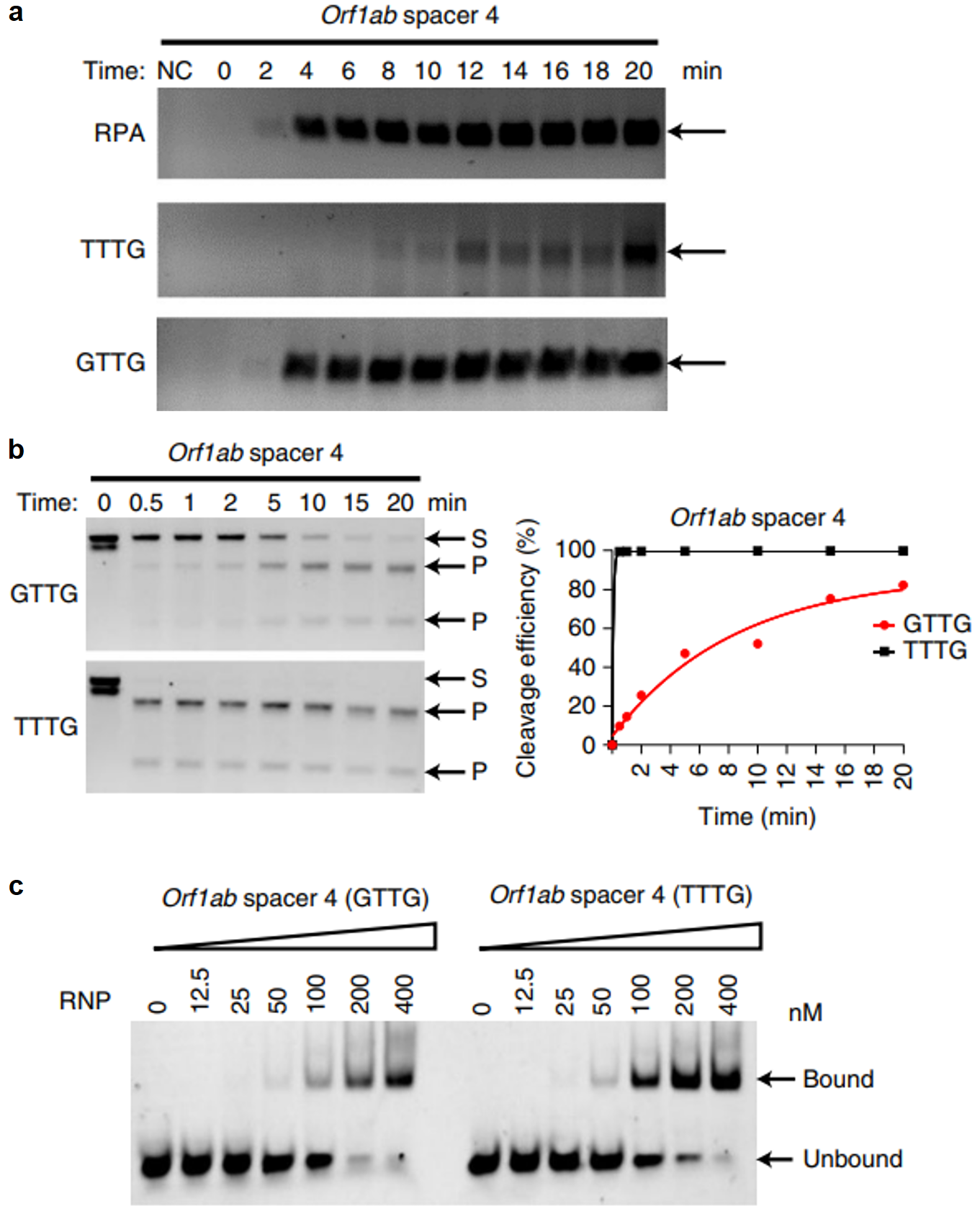
图二 单管法反应中存在靶序列的RPA扩增与Cas12a顺式切割竞争[14]
研究者进一步突变一系列的PAM基序来评价其对Cas12a的顺式切割活性与单管法反应时间的影响。结果显示,典型PAM基序虽然介导Cas12a的顺式切割活性最高,但在单管法反应中耗时较长(图三a,黑点);表现佳的非典型PAM基序虽然顺式切割活性适中,但大大缩短了单管法反应时间(图三a,红点);还有一部分表现差的非典型PAM基序由于顺式切割活性太低,导致单管法反应耗时最长(图三a,蓝点)。结果表明,Cas12a对部分非典型PAM基序的靶序列结合亲和力降低,激活适当的Cas12a顺式切割活性,促进单管法反应的平衡从原先的顺式切割转向为RPA扩增;使得靶序列DNA充分扩增产生足够的激活底物,进而激活Cas12a的旁侧切割活性,使得荧光探针被充分切割释放荧光信号;最终大大提高了单管法核酸检测的灵敏度,并且显著缩短了反应时间(图三b)。经统计发现,sPAMC单管法中非典型PAM基序VTTV的表现最佳,TCTV次之,后续可针对这两种非典型次优PAM基序来设计crRNA。
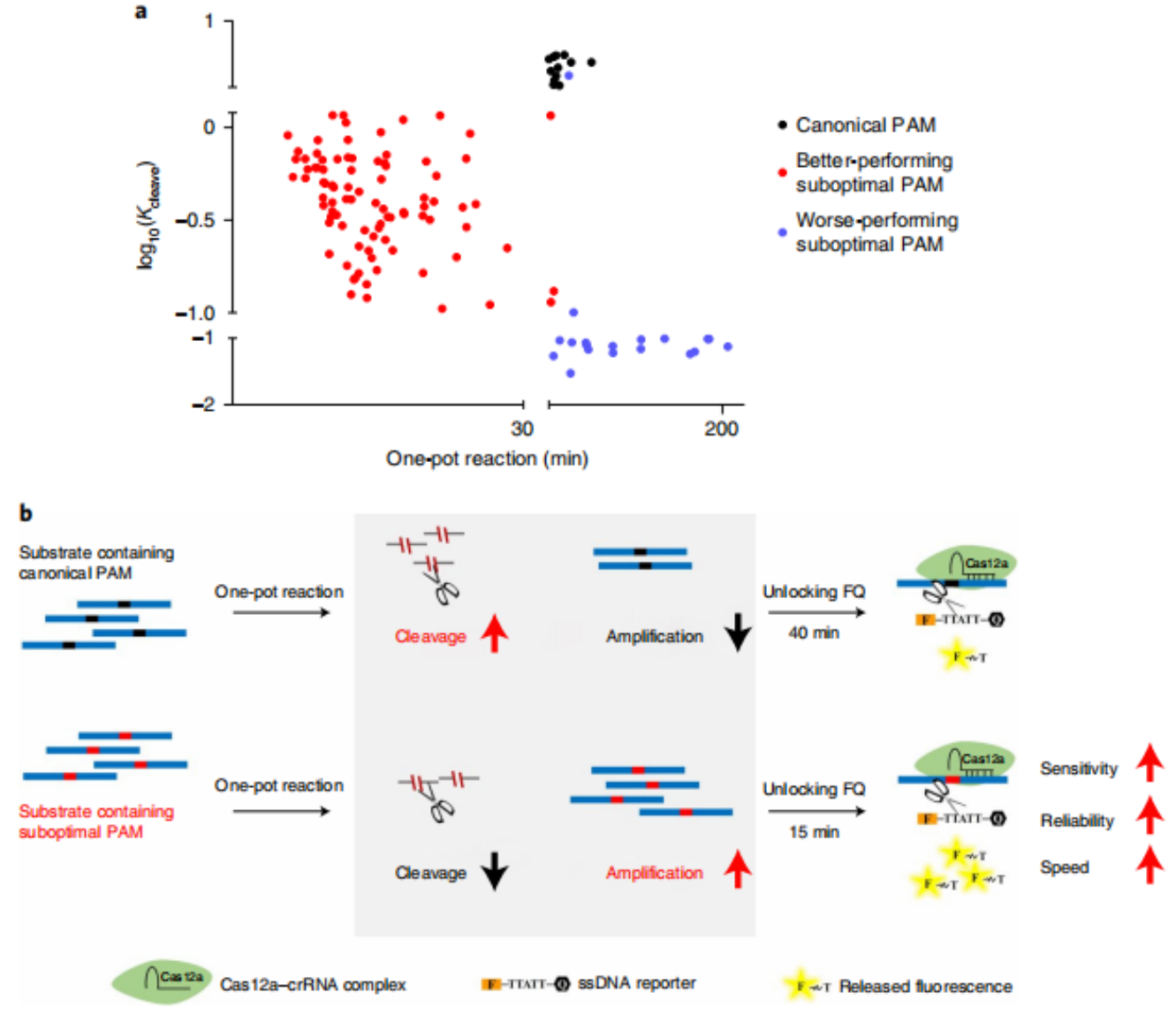
图三 sPAMC单管法提高灵敏度与检测速度的原理图[14]
在开发特定病毒分子诊断的实际应用中,研究者先选择一种DNA病毒的人巨细胞病毒(HCMV)进行sPAMC单管法测试。结果显示,sPAMC单管法检测HCMV的最低病毒拷贝数为24(图四a),灵敏度与RT-qPCR相当(图四b)。sPAMC单管法同样可通过添加不同的荧光探针来实现UV荧光信号检测或者侧向层析可视化检测(图四c)。反应15分钟后,拷贝数24的最低可检测病毒实验组可经紫外照射检测到明显阳性信号(图四d)。侧向层析可视化检测的灵敏度会低些,最低仅能检测到Ct值为33-34的病毒样本(图四e)。因此,研究者后续仅利用sPAMC单管法的UV检测来开发特定病毒的分子诊断方法。
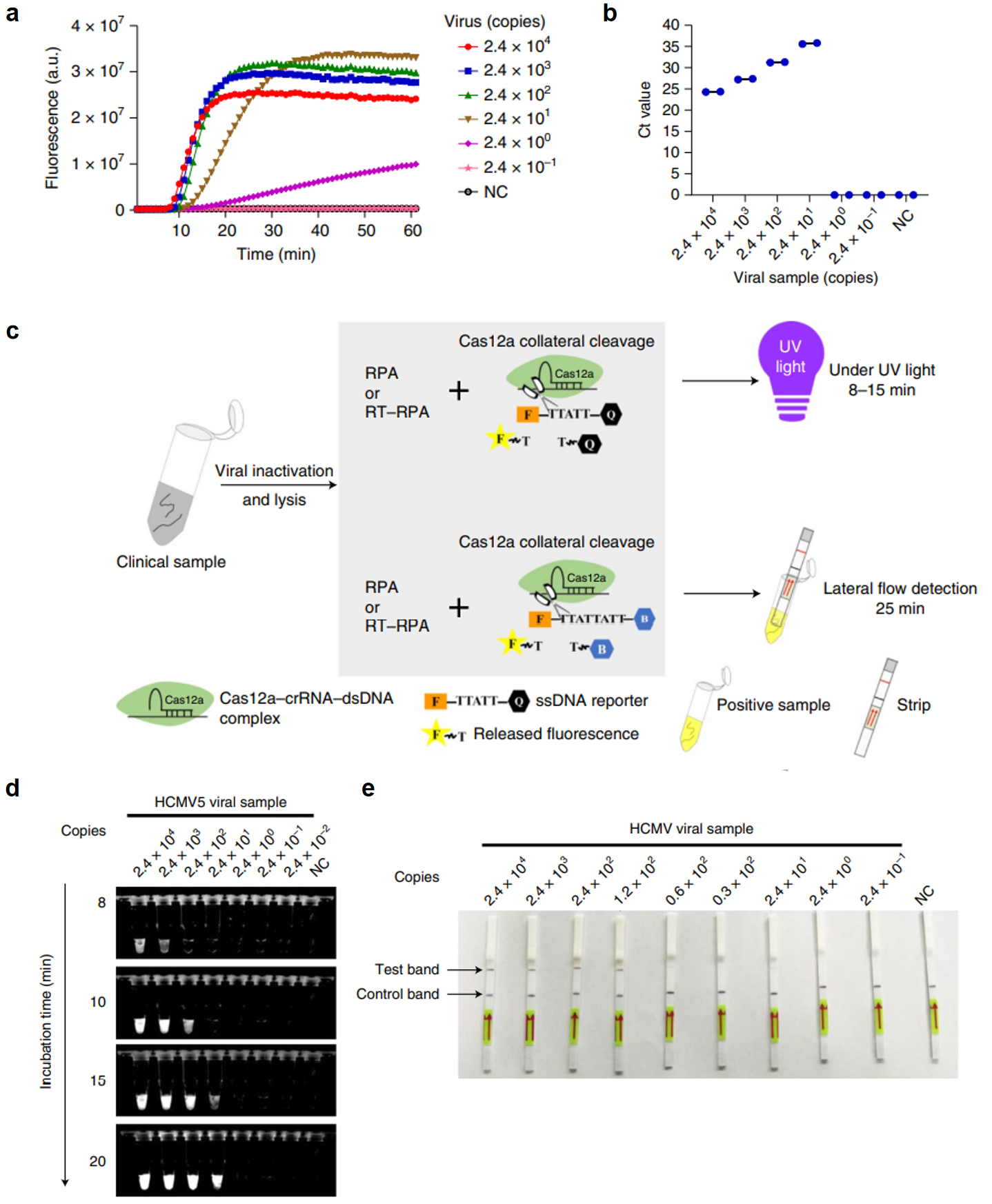
图四 sPAMC单管法用于HCMV检测[14]
最后,研究者采用sPAMC单管法来开发新冠病毒的分子诊断方法。基于表现最优的三种非典型次优PAM基序VTTV、TTVV和TCTV可设计出更多的crRNA用于试剂盒的开发(图五a)。研究者将基于RPA/Cas12a/非典型次优PAM的sPAMC和基于LAMP/Cas12b的STOPCovid进行头对头比较实验,发现sPAMC在DNA和RNA的最低检测浓度(图五b)和检测时间(图五d)上都显著优于STOPCovid(图五c和e)。在新冠病毒鼻咽拭子104份阳性样本和100份阴性样本的临床测试结果中,sPAMC的敏感性为94.2%,特异性为100.0%(图五f-h)。sPAMC最低可检测到RT-qPCR的Ct值为35.8的样本,并且所有阳性样本在15分钟的反应时间都出现了阳性信号(图五g)。相比之下,STOPCovid.v1无法稳定检测Ct值在31.0以上的样品,灵敏度为78.8%。此外,sPAMC的新冠病毒检测与其他常见病毒没有交叉反应,特异性好。

图五 sPAMC单管法用于新冠病毒检测[14]
综上所述,sPAMC单管法是一种具有快速、高灵敏度、高可靠性和灵活性相结合的Cas12a介导的核酸检测方法,可快速用于开发各种特定病毒核酸的即时检验(POCT),并可开发核酸检测试剂盒用于实验室以外护理点的快速病毒诊断。

GTP研发中心拥有自主知识产权的“人造精子细胞”介导的半克隆技术,可在体外实现“人造精子细胞”的多位点改造并进行功能元件测试,最后将“人造精子细胞”通过卵母细胞注射可以一步法获得基因改造小鼠。基于“人造精子细胞”单倍体打靶高效率的优势,可快速构建定制化小鼠,如基因组安全位点大片段敲入、条件性基因敲除/敲入和人源化基因改造等。

如有需要欢迎联系我们,GTP研发中心将竭诚为您设计最佳方案,制定专属于您的基因改造小鼠。
网址:http://www.sibcb.ac.cn/gtp/
邮箱:gtp_order@sibcb.ac.cn
地址:上海市岳阳路320号
责任编辑:多一百
新媒体运营:suway
参考文献
1. Kuiken T, Fouchier RA, Schutten M, Rimmelzwaan GF, van Amerongen G, van Riel D, Laman JD, de Jong T, van Doornum G, Lim W et al: Newly discovered coronavirus as the primary cause of severe acute respiratory syndrome. Lancet 2003, 362(9380):263-270.
2. Zaki AM, van Boheemen S, Bestebroer TM, Osterhaus AD, Fouchier RA: Isolation of a novel coronavirus from a man with pneumonia in Saudi Arabia. N Engl J Med 2012, 367(19):1814-1820.
3. Chang SY, Bowman BH, Weiss JB, Garcia RE, White TJ: The origin of HIV-1 isolate HTLV-IIIB. Nature 1993, 363(6428):466-469.
4. Wang C, Horby PW, Hayden FG, Gao GF: A novel coronavirus outbreak of global health concern. Lancet 2020, 395(10223):470-473.
5. Vogels CBF, Brito AF, Wyllie AL, Fauver JR, Ott IM, Kalinich CC, Petrone ME, Casanovas-Massana A, Catherine Muenker M, Moore AJ et al: Analytical sensitivity and efficiency comparisons of SARS-CoV-2 RT-qPCR primer-probe sets. Nat Microbiol 2020, 5(10):1299-1305.
6. Piepenburg O, Williams CH, Stemple DL, Armes NA: DNA Detection Using Recombination Proteins. Plos Biology 2006, 4(7):1115-1121.
7. Tsugunori N, Hiroto O, Harumi M, Toshihiro Y, Keiko W, Nobuyuki A, Tetsu H: Loop-mediated isothermal amplification of DNA. Nucleic Acids Research 2000(12):E63.
8. Gootenberg JS, Abudayyeh OO, Lee JW, Essletzbichler P, Dy AJ, Joung J, Verdine V, Donghia N, Daringer NM, Freije CA: Nucleic acid detection with CRISPR-Cas13a/C2c2. Science 2017, 356(6336):438-442.
9. Chen JS, Ma E, Harrington LB, Da Costa M, Tian X, Palefsky JM, Doudna JA: CRISPR-Cas12a target binding unleashes indiscriminate single-stranded DNase activity. Science 2018, 360(6387):eaar6245.
10. Broughton JP, Deng X, Yu G, Fasching CL, Servellita V, Singh J, Miao X, Streithorst JA, Granados A, Sotomayor-Gonzalez A et al: CRISPR-Cas12-based detection of SARS-CoV-2. Nat Biotechnol 2020, 38(7):870-874.
11. Patchsung M, Jantarug K, Pattama A, Aphicho K, Uttamapinant C: Clinical validation of a Cas13-based assay for the detection of SARS-CoV-2 RNA. Nature Biomedical Engineering 2020:1-10.
12. Joung J, Ladha A, Saito M, Kim NG, Woolley AE, Segel M, Barretto RPJ, Ranu A, Macrae RK, Faure G et al: Detection of SARS-CoV-2 with SHERLOCK One-Pot Testing. N Engl J Med 2020, 383(15):1492-1494.
13. Arizti-Sanz J, Freije CA, Stanton AC, Petros BA, Myhrvold C: Streamlined inactivation, amplification, and Cas13-based detection of SARS-CoV-2. Nature Communications 2020, 11(1):5921.
14. Lu S, Tong X, Han Y, Zhang K, Zhang Y, Chen Q, Duan J, Lei X, Huang M, Qiu Y et al: Fast and sensitive detection of SARS-CoV-2 RNA using suboptimal protospacer adjacent motifs for Cas12a. Nat Biomed Eng 2022, 6(3):286-297.



