技术分享:基于CRISPRa-SAM小鼠的疾病造模方法
基因过表达常用于基因功能和疾病机制研究。CRISPRa(CRISPR-based transcriptional activation)系统为基因过表达提供了一个灵活高效的工具。它通过催化失活的Cas9(dCas9)融合转录激活子,结合到靶基因的启动子区域激活靶基因的转录,实现过表达的目的[1, 2]。在CRISPRa的系统中,SAM(Synergistic Activation Mediator)系统的激活效果稳定,适合在体研究[3]。SAM系统需要三个元件:(1)MS2发夹结构融合到sgRNA序列上,(2)MS2结合蛋白(MCP)与转录激活因子p65和HSF1相融合,(3)dCas9与转录激活因子VP64相融合。CRISPRa-SAM系统利用sgRNA特异性识别并结合到靶基因的启动子上,招募MCP和dCas9蛋白,从而在多种转录激活因子的协同作用下,高效模拟细胞内的转录激活[4]。
2021年5月,Nature Communications上发表了一种CRISPRa-SAM小鼠,可以结合体内递送系统来模拟多种疾病表型,包括高胆固醇血症和转甲状腺素淀粉样变(ATTR)的疾病模型[5]。
研究者首先构建了CRISPRa-SAM小鼠,在基因组安全位点Rosa26(R26)定点插入CRISPRa-SAM系统所需元件。在工具鼠中,将CRISPRa-SAM系统的两个固定元件dCas9-VP64和MCP-p65-HSF1通过2A肽连接,并在上游插入loxP-stop-loxP(LSL)终止盒以防止R26内源启动子驱动该元件表达(图一a,R26LSL-SAM)。在Cre重组酶存在下,LSL终止盒被去除,启动CRISPRa-SAM系统的两个元件表达(图一a和b,R26SAM)。然后将CRISPRa-SAM系统靶点识别的第三个元件sgRNA也通过打靶构建在R26上,命名为R26#a[Gene],其中sgRNA由U6启动子驱动,几个sgRNA序列被串联在一起(图一c)。另外,激活sgRNA的位置设计在转录起始位点(TSS)上游的300 bp以内(图一d)。
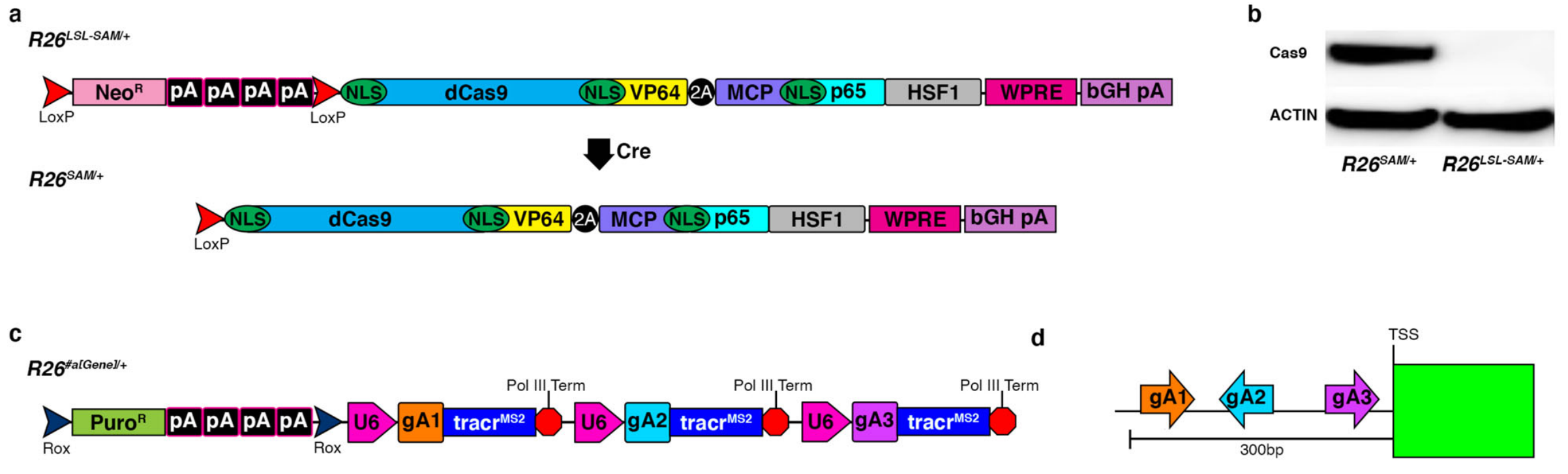
图一 CRISPRa-SAM小鼠的工作原理示意图[5]
接着,研究者对CRISPRa-SAM小鼠的在体基因激活能力进行测试,设计sgRNA靶向激活Ttr基因的表达。将R263aTtr与R26SAM两品系小鼠交配获得R26SAM/3aTtr小鼠,ELISA检测到血清中TTR蛋白含量维持在高出对照组2.5倍的水平(图二a)。qPCR结果表明所有被测组织中Ttr表达均显著升高,内源性高表达的组织如肝脏表达上调了2.5倍,内源性低表达的组织如肺上调高达98,000倍(图二b)。RNA-Seq结果表明,在各个组织中仅有Ttr基因的表达被激活,Ttr基因组上相邻的其他基因表达并没有受影响(图二c)。为了进一步可视化证明这一结论,将R26SAM/3aTtr小鼠与TtrLacZ小鼠进行交配,后者是将Ttr基因组编码序列替换为编码β半乳糖苷酶的lacZ基因(图二d)。检测R26SAM/3aTtr;TtrLacZ/+小鼠发现,与对照组相比所有组织器官都有很强的lacZ阳性染色,包括本底低表达的组织器官如脾脏(图二e)。以上结果表明,CRISPRa-SAM小鼠可全身性高效特异激活靶基因。
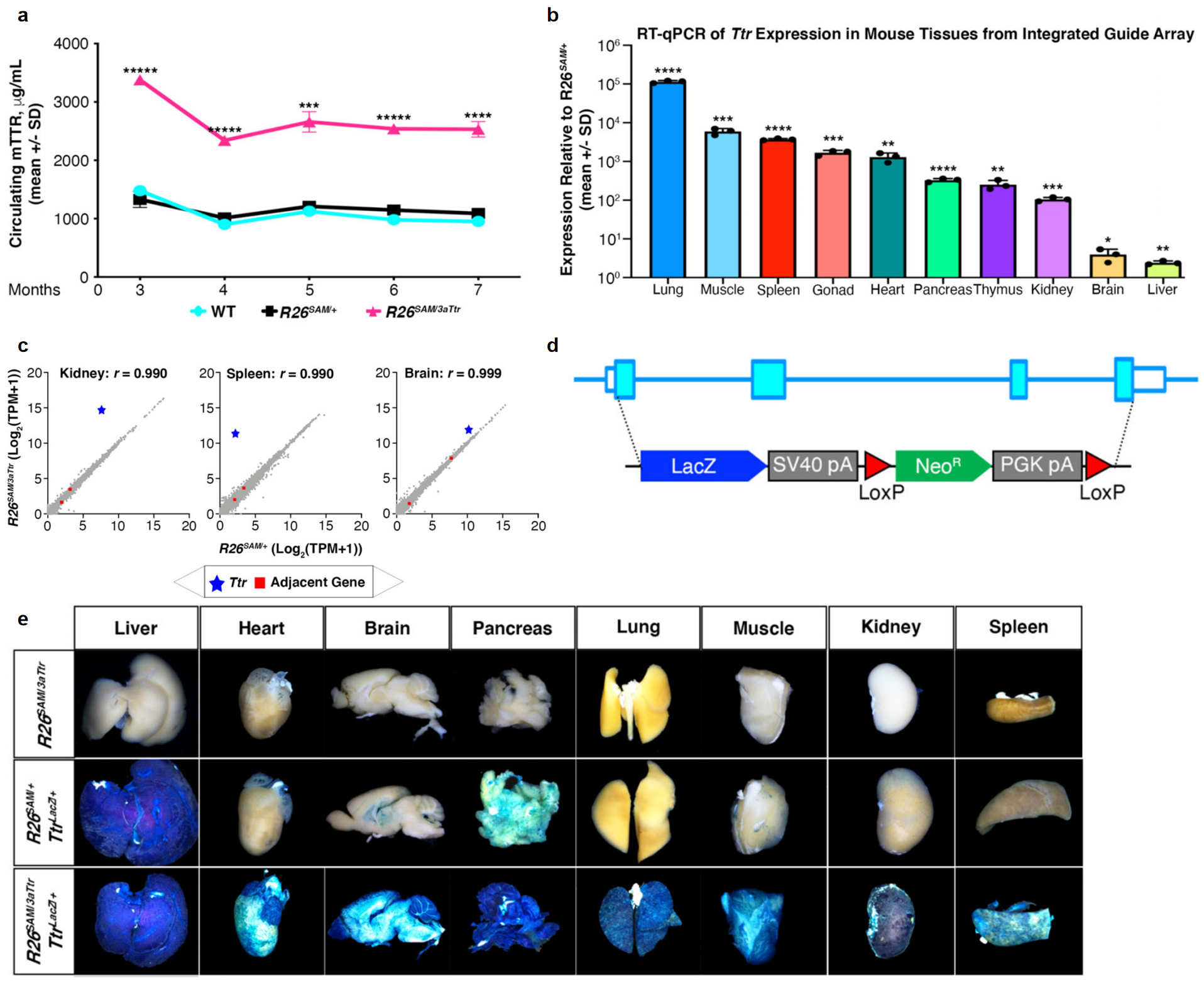
图二 CRISPRa-SAM小鼠的在体基因激活能力验证[5]
利用CRISPRa-SAM小鼠的高效在体激活能力,可快速构建各种疾病模型。为了构建高胆固醇血症疾病模型,研究者选择了Pcsk9靶点进行测试(图三)。PCSK9是一个丝氨酸蛋白酶,主要在肝脏合成,它能够减少肝细胞中低密度脂蛋白受体(LDLR)的数量。PCSK9与位于细胞膜表面的LDLR结合后,内吞回胞质内,促进溶酶体对LDLR的降解。LDLR是胆固醇稳态的关键,它负责细胞对LDL的摄取和降解。因此,PCSK9的含量升高会导致LDLR数量减少,使得血浆中LDL水平增高,造成高胆固醇血症[6]。在工具小鼠R26SAM中,利用AAV8病毒载体来递送靶向Pcsk9的激活sgRNA,与对照组相比均可检测到血浆中LDL胆固醇含量的显著升高,其中注射了AAV8Pcsk9-gA1的小鼠水平升高最为显著(图三a和d);同样地,总胆固醇水平也显著升高(图三b和e);对高密度脂蛋白HDL的影响较小(图三c和f)。雄鼠相比于雌鼠的表型更为明显。以上结果表明,利用CRISPRa-SAM小鼠可快速构建高胆固醇血症疾病模型。

图三 CRISPRa-SAM小鼠的高胆固醇血症模型构建[5]
最后,研究者利用工具小鼠R26SAM来构建转甲状腺素淀粉样变(ATTR)疾病模型。ATTR是由于错误折叠的转甲状腺素(TTR)蛋白在组织中的积聚而导致,主要发生在神经和心脏。ATTR的研究模型通常采用人源化TTR小鼠(TTRHu/Hu),将小鼠基因组的Ttr基因替换为人的TTR基因(图四a)。可是TTRHu/Hu小鼠虽然仅表达人源TTR蛋白,但与正常人血浆中(NHS)的TTR蛋白表达水平仍有显著差距(图四b),无法模拟ATTR疾病表型。于是研究者在纯合TTRHu/Hu;R26SAM/SAM小鼠中,利用AAV8来递送靶向Ttr的激活sgRNA,与对照组相比可显著提高TTR蛋白的含量(图四c),其表达量显著高于正常人血浆水平。因此,该小鼠模型能更好地模拟ATTR疾病表型。
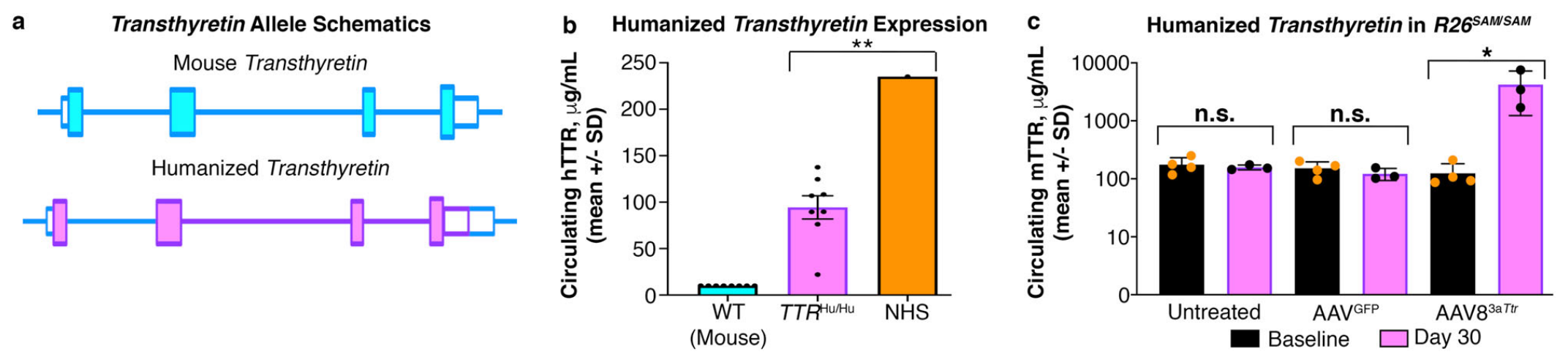
图四 CRISPRa-SAM小鼠的转甲状腺素淀粉样变模型构建[5]
综上所述,研究者将CRISPRa-SAM系统的两个固定元件定点插入在基因组安全位点,来构建工具小鼠R26SAM,使得两个元件可以在小鼠体内全身性稳定表达;并且单拷贝的插入形式可以被稳定遗传,方便小鼠繁殖与鉴定。另外,将CRISPRa-SAM系统靶点识别的第三个元件sgRNA利用AAV等载体进行体内递送,方便快速构建质粒,更换各种靶点,增加应用灵活性;并能根据体内递送系统的属性,靶向不同组织,实现组织特异性地激活靶基因,从而加速在体研究和药物研发的进程。

GTP研发中心拥有自主知识产权的“人造精子细胞”介导的半克隆技术,可在体外实现“人造精子细胞”的多位点改造并进行功能元件测试,最后将“人造精子细胞”通过卵母细胞注射可以一步法获得基因改造小鼠。基于“人造精子细胞”单倍体打靶高效率的优势,可快速构建定制化小鼠,如基因组安全位点大片段敲入、条件性基因敲除/敲入和人源化基因改造等。更多复杂的调控元件可以通过多次体外细胞打靶构建在同一株“人造精子细胞”中,经细胞水平质控后,一步法获得基因改造小鼠。

如有需要欢迎联系我们,GTP研发中心将竭诚为您设计最佳方案,制定专属于您的基因改造小鼠。
网址:http://www.sibcb.ac.cn/gtp/
邮箱:gtp_order@sibcb.ac.cn
地址:上海市岳阳路320号
责任编辑:张一鸣
新媒体运营:suway
参考文献
1. Lundh M, Pluciñska K, Isidor MS, Petersen PSS, Emanuelli B: Bidirectional manipulation of gene expression in adipocytes using CRISPRa and siRNA. Mol Metab 2017, 6(10):1313-1320.
2. Matharu N, Rattanasopha S, Tamura S, Maliskova L, Wang Y, Bernard A, Hardin A, Eckalbar WL, Vaisse C, Ahituv N: CRISPR-mediated activation of a promoter or enhancer rescues obesity caused by haploinsufficiency. Science 2019, 363(6424):eaau0629.
3. Chavez A, Tuttle M, Pruitt BW, Ewen-Campen B, Chari R, Ter-Ovanesyan D, Haque SJ, Cecchi RJ, Kowal EJK, Buchthal J et al: Comparison of Cas9 activators in multiple species. Nature methods 2016, 13(7):563-567.
4. Konermann S, Brigham MD, Trevino AE, Joung J, Abudayyeh OO, Barcena C, Hsu PD, Habib N, Gootenberg JS, Nishimasu H et al: Genome-scale transcriptional activation by an engineered CRISPR-Cas9 complex. Nature 2015, 517(7536):583-588.
5. Hunt C, Hartford SA, White D, Pefanis E, Hanna T, Herman C, Wiley J, Brown H, Su Q, Xin Y et al: Tissue-specific activation of gene expression by the Synergistic Activation Mediator (SAM) CRISPRa system in mice. Nat Commun 2021, 12(1):2770.
6. Shapiro MD, Tavori H, Fazio S: PCSK9: From Basic Science Discoveries to Clinical Trials. Circulation research 2018, 122(10):1420-1438


