技术分享:肠道非模式菌的遗传工程改造方法
数量庞大的肠道菌群寄居在人体肠道内,可以帮助宿主消化吸收、保护肠道和调节生理健康,与宿主及外部环境保持生态平衡。肠道菌群的失调不仅会引起宿主的胃肠道疾病和代谢性疾病,还与神经系统、呼吸系统、心血管系统、肿瘤等疾病息息相关。肠道菌群主要通过菌群代谢产生的小分子代谢物如短链脂肪酸、胆汁酸、色氨酸、氨基酸等,来交互影响宿主健康。为此,科学家们致力于解析肠道菌群与人类健康之间的作用机理,对肠道菌群进行遗传工程改造来功能性验证疾病机制,从而开发工程肠道益生菌来阻止疾病发展或治疗疾病。
近年来,科学家们对模式菌如大肠杆菌、多形类杆菌的研究已经很完备,可对其进行各种遗传工程改造[1]。然而肠道菌群中多为非模式菌,研究依然存在困难。一方面多数菌种还未完成基因组测序,无法确定引入外源DNA的方式[1];另一方面还未能建立高效、标准化的非模式菌遗传工程改造方法。2022年1月,美国康奈尔大学的郭春君团队在Cell上发表了用于肠道非模式菌中厚壁菌门梭菌纲的遗传工程改造方法,并利用该方法获得的baiH基因敲除的工程改造菌来研究其影响宿主生理功能[2]。
研究者通过对多个参数进行优化,最终确定了38种梭菌纲菌株的质粒导入条件。以此为基础,采用CRISPRi-dCpf1和第Ⅱ类内含子(Group Ⅱ intron)两种基因编辑手段来对这些梭菌纲菌株分别进行基因敲降(KD)和基因敲入(KI)的遗传工程改造。
基因敲降的实现是利用CRISPRi-dCpf1系统中的dCpf1/crRNA复合物与靶基因的模板DNA链相结合,阻断靶基因的转录延伸,从而抑制靶基因表达[3]。dCpf1相比Cas9和Cpf1,不会引发DNA双链断裂,质粒毒性更小,结合效率更高[3, 4]。研究者利用lacZα表达来指示导入片段的表达情况,并设计靶向lacZα的2个gRNA(G1、G2)来实现对lacZα的敲降(图一Ai)。将CRISPRi-dCpf1系统利用优化后的质粒导入条件转入梭菌纲菌株中,在多种菌株中均可发现同时携带dCpf1、gRNA和lacZα的三种过表达元件的质粒导入,可显著性抑制lacZα的表达(图一Aii),表明基因敲降系统构建成功。
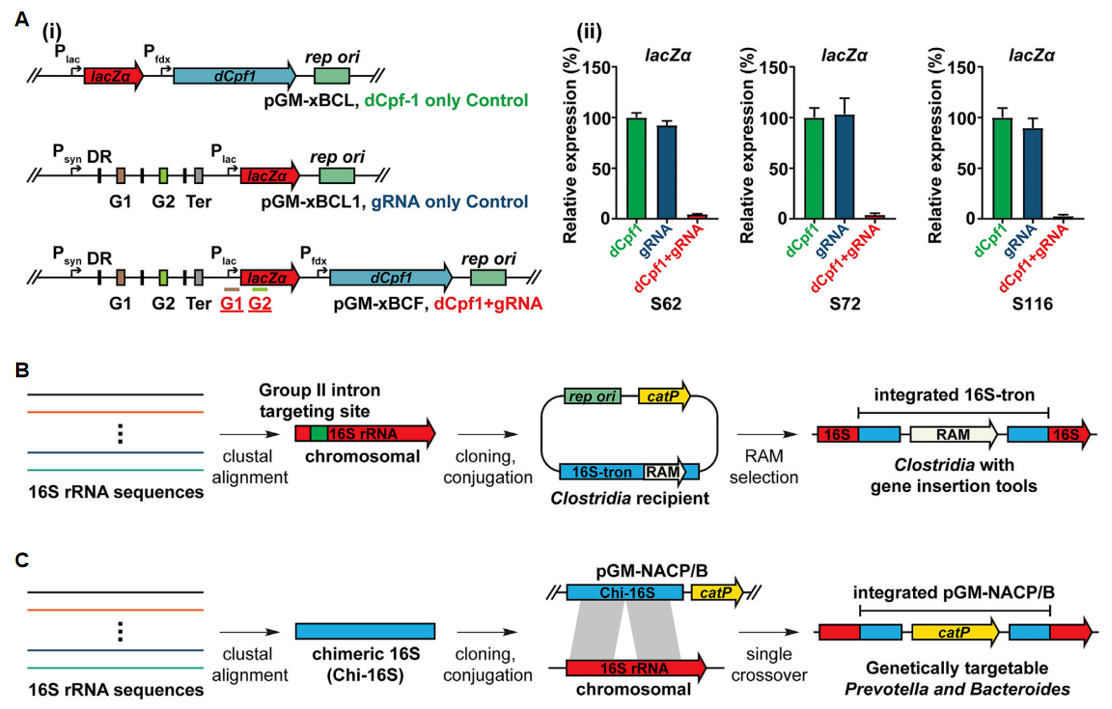
图一 非模式肠道菌群的CRISPRi-dCpf1与第II类内含子系统的打靶示意图[2]
基因敲入的实现需要选择合适的基因组位点来进行操作,研究者选择了16S rRNA来进行测试。首先它是多拷贝基因,由于KI导致的一个拷贝基因被破坏并不会致死;其次它在菌株中高度保守,可作为KI工具位点用于不同菌株中,既节约时间又便于克隆和测序[5]。研究者利用第II类内含子归巢的原理,将携带逆转录激活筛选标志物(RAM)的内含子序列定点插入菌株染色体上16S rRNA上的靶位点中(图一B)。实验发现,在18个可靶向的梭菌菌株中有15个菌株的染色体成功插入16S-tron。另外,研究者还利用同源重组将嵌合16S(Chi-16S)序列结合自杀基因系统,成功引入其他多个菌种的16S rRNA位点上(图一C)。这些数据表明,在不同的菌株中可利用上述两种方法来实现KI。
接下来,研究者利用上述方法来对肠道菌群进行遗传工程改造,从而功能性验证肠道菌群对宿主疾病的互作影响。在肠道菌群的作用下,胆酸等初级胆汁酸通过7α-脱羟基化转变为次级胆汁酸,从而易引发肠道炎症[6]。研究者通过进一步实验发现在具有复杂菌群的小鼠体内,移植baiH敲除突变株的小鼠丧失了7α-脱羟基化的活性,影响了肠道胆汁酸和菌群的分布,具有更好的抗炎症表型(图二)。
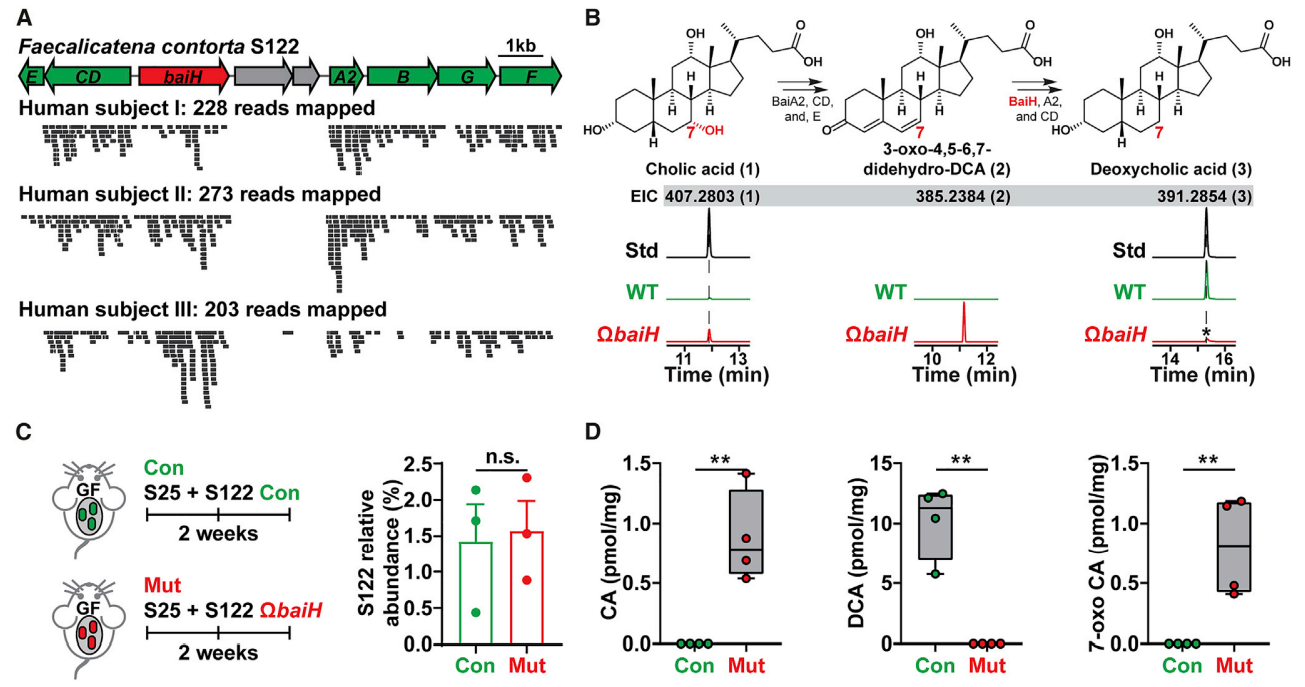
图二 肠道菌群的baiH基因敲除影响小鼠肠道炎症的发生[2]
综上所述,研究者成功建立了肠道非模式菌中厚壁菌门梭菌纲等菌株的遗传工程改造方法,将大大推动肠道菌群与人体健康的互作机制研究,有望实现重大慢性疾病的预防和治疗。

GTP研发中心拥有自主知识产权的“人造精子细胞”介导的半克隆技术,可在体外实现“人造精子细胞”的多位点改造并进行功能元件测试,最后将“人造精子细胞”通过卵母细胞注射可以一步法获得基因改造小鼠。基于“人造精子细胞”单倍体打靶高效率的优势,可快速构建定制化小鼠,如基因组安全位点大片段敲入、条件性基因敲除/敲入和人源化基因改造等。多个复杂调控元件可以通过多次体外细胞打靶构建在同一株“人造精子细胞”中,经细胞水平质控后,一步法获得基因改造小鼠。

如有需要欢迎联系我们,GTP研发中心将竭诚为您设计最佳方案,制定专属于您的基因改造小鼠。
网址:http://www.sibcb.ac.cn/gtp/
邮箱:gtp_order@sibcb.ac.cn
地址:上海市岳阳路320号
责任编辑:张一鸣
新媒体运营:suway
参考文献
1. Waller MC, Bober JR, Nair NU, Beisel CL:Toward a genetic tool development pipeline for host-associated bacteria. CurrOpin Microbiol 2017, 38:156-164.
2. Jin WB, Li TT, Huo D, Qu SP, Li XV,Arifuzzaman M, Lima SF, Shi HQ, Wang AL, Putzel GG et al: Genetic manipulationof gut microbes enables single-gene interrogation in a complex microbiome. Cell2022, 185(3):547-562.
3. Kim SK, Kim H, Ahn WC, Park KH, Woo EJ, LeeDH, Lee SG: Efficient Transcriptional Gene Repression by Type V-A CRISPR-Cpf1from Eubacterium eligens. Acs Synth Biol 2017, 6(7):1273-1282.
4. Zhang XC, Wang JM, Cheng QX, Zheng X, ZhaoGP, Wang J: Multiplex gene regulation by CRISPR-ddCpf1. Cell Discov 2017,3:17018.
5. Isenbarger TA, Carr CE, Johnson SS, FinneyM, Church GM, Gilbert W, Zuber MT, Ruvkun G: The Most Conserved Genome Segmentsfor Life Detection on Earth and Other Planets. Origins Life Evol B 2008,38(6):517-533.
6. Sinha SR, Haileselassie Y, Nguyen LP,Tropini C, Wang M, Becker LS, Sim D, Jarr K, Spear ET, Singh G et al: Dysbiosis-InducedSecondary Bile Acid Deficiency Promotes Intestinal Inflammation. Cell HostMicrobe 2020, 27(4):659-670.

