技术分享:一个集合Vika、Flp、Dre和Cre重组系统的报告基因小鼠模型
Tips:上一期我们分享了Cre-loxP重组酶系统,Cre重组酶是一种位点特异性重组酶,通过与一个34 bp的核苷酸序列,即loxP位点结合,使其发生删除、重组或颠换。loxP位点的反向回文序列是Cre重组酶的识别和结合区域,另外中间的间隔序列为同源重组区和可变区,决定了loxP的方向。本文将介绍其他位点特异性重组酶系统:Flp-FRT系统、Dre-rox系统和Vika-vox系统。
位点特异性重组酶(SSR)是一种重要的基因组工程工具,可实现基因组精确改造,用于合成生物学[1]、条件诱变[2]和谱系追踪[3]等。目前已有报道显示可成功在小鼠体内同时使用Cre-loxP、Flp-FRT和Dre-rox这三种SSR系统[4],但是后来发现的新型Flp-like[5, 6]和Cre-like[7-9]重组系统,还都没有在转基因小鼠中进行有效性和特异性测试。
2018年9月,Scientific Reports上报道了一个集合Vika、Flp、Dre和Cre重组系统的报告基因小鼠模型,简称为MuX(multi-reporter mouse line for loci-of-recombination (X))小鼠[10]。研究人员首先在小鼠胚胎干细胞系(mESC)中交叉测试各种SSR系统的效率和特异性,证明了Cre-loxP、Vika-vox、Dre-rox和Flpo-FRT这四个SSR系统均能高效进行重组,且四个SSR系统之间不会发生交叉反应,具有系统特异性(图一)。其中Flpo是Flp的变体,Flp重组酶的最佳反应温度为30℃,为了更好的用于细胞系与动物体内的重组反应开发出了Flpo重组酶,其在37℃表现出较好的耐热性。
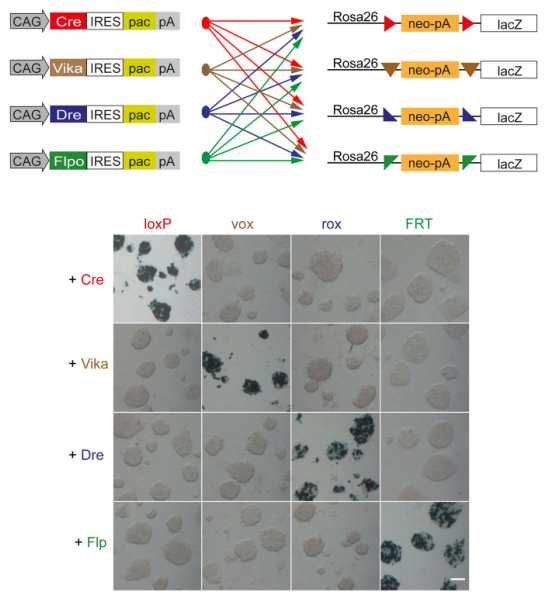
图一 mESC中分别测试四个重组酶系统的特异性[10]
鉴于四个SSR系统的特异性,研究者将这四个SSR系统合并设计在Rosa26安全位点的一个报告基因MuX载体中,并进行mESC打靶建系与测试,EGFP荧光表达结果显示Rosa26-MuX系统在mESC中测试成功(图二)。于是,研究者将携带MuX系统的mESC进行8细胞胚胎注射,获得嵌合小鼠,再通过与野生型小鼠交配获得生殖系遗传的MuX小鼠。MuX小鼠通过分别与携带Vika、Flpo、Dre和Cre重组酶的转基因小鼠进行交配,检测E10.5天的子代胚胎,可以看到各组织中EGFP的均质表达(图三)。同样地,成年子代的不同器官组织内EGFP也均质表达(图四)。
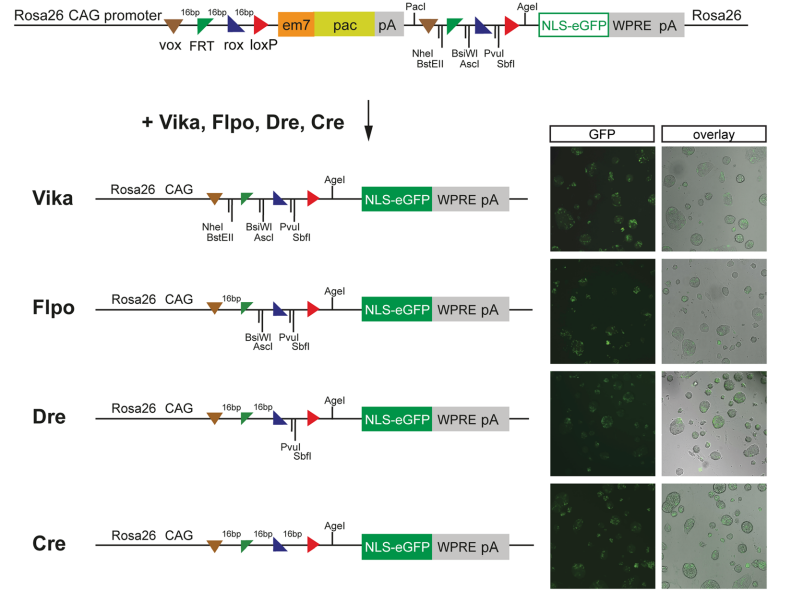
图二 Rosa26-MuX系统在mESC中打靶并进行重组效率测试[10]
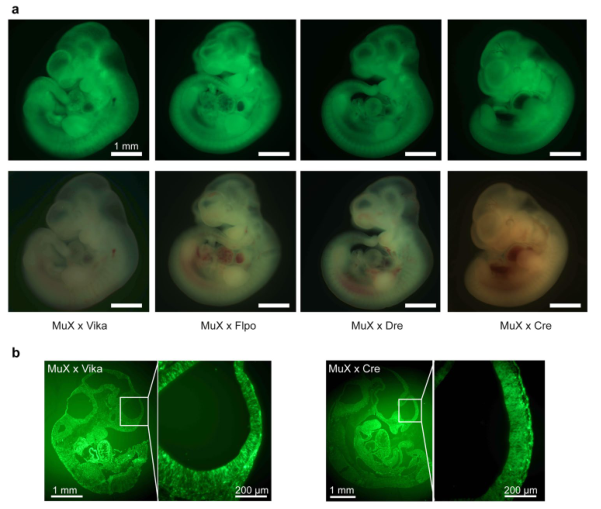
图三 Rosa26-MuX小鼠子代E10.5天胚胎的重组效率测试[10]
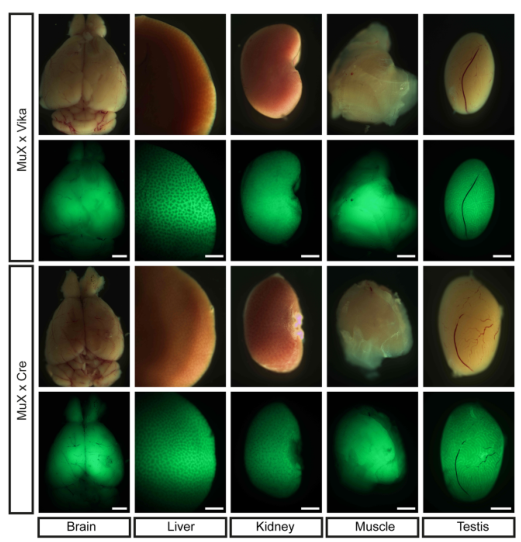
图四 Rosa26-MuX小鼠成年子代的重组效率测试[10]
综上所述,MuX小鼠集合Vika-vox、Flpo-FRT、Dre-rox和Cre-loxP这四个SSR系统的报告基因于一体,四个SSR系统重组效率高、特异性好,方便研究者灵活使用。针对每一种重组酶系统来构建一个对应的报告基因小鼠是很麻烦的,MuX小鼠集合四个SSR系统的报告基因于一身,大大减少了小鼠笼位数量,节约了时间和经费,并且促进了基于复杂重组酶系统的在体基因组工程设计和研究。在条件诱变方面,新的位点特异性重组酶可以在一些长链非编码RNA或高同源性基因的某些关键序列进行特异性切除。在细胞命运谱系追踪方面,可以通过使用多个重组酶和报告基因在不同组织中发现新的细胞类群等。多个SSR系统在一种动物体内的组合,还可以在创新研发以及生物医学应用上挖掘出更大的潜力。

GTP研发中心拥有自主知识产权的“人造精子细胞”介导的半克隆技术,可在体外实现“人造精子细胞”的多位点改造并进行功能元件测试,最后将“人造精子细胞”通过卵母细胞注射可以一步法获得基因改造小鼠。基于“人造精子细胞”单倍体打靶高效率的优势,可快速构建定制化小鼠,如基因组安全位点大片段敲入、条件性基因敲除/敲入和人源化基因改造等。
多个重组酶与报告基因系统可以通过在体外细胞打靶构建在同一株“人造精子细胞”中,经细胞水平质控后,一步法快速获得基因改造小鼠。“人造精子细胞”单倍体打靶效率更高,且无需漫长的嵌合体小鼠繁殖交配等待。

如有需要欢迎联系我们,GTP研发中心将竭诚为您设计最佳方案,制定专属于您的基因改造小鼠。
网址:http://www.sibcb.ac.cn/gtp/
邮箱:gtp_order@sibcb.ac.cn
地址:上海市岳阳路320号
责任编辑:张一鸣
新媒体运营:suway
参考文献
1. Chakravarti D, Cho JH, Weinberg BH, Wong NM, Wong WW: Synthetic biology approaches in cancer immunotherapy, genetic network engineering, and genome editing. Integr Biol (Camb) 2016, 8(4):504-517.
2. Bouabe H, Okkenhaug K: Gene targeting in mice: a review. Methods Mol Biol 2013, 1064:315-336.
3. Weissman TA, Pan YA: Brainbow: new resources and emerging biological applications for multicolor genetic labeling and analysis. Genetics 2015, 199(2):293-306.
4. Plummer NW, Evsyukova IY, Robertson SD, de Marchena J, Tucker CJ, Jensen P: Expanding the power of recombinase-based labeling to uncover cellular diversity. Development 2015, 142(24):4385-4393.
5. Chandras C, Zouberakis M, Salimova E, Smedley D, Rosenthal N, Aidinis V: CreZOO--the European virtual repository of Cre and other targeted conditional driver strains. Database (Oxford) 2012, 2012:bas029.
6. Nagy A, Mar L, Watts G: Creation and use of a cre recombinase transgenic database. Methods Mol Biol 2009, 530:365-378.
7. Suzuki E, Nakayama M: VCre/VloxP and SCre/SloxP: new site-specific recombination systems for genome engineering. Nucleic Acids Res 2011, 39(8):e49.
8. Karimova M, Abi-Ghanem J, Berger N, Surendranath V, Pisabarro MT, Buchholz F: Vika/vox, a novel efficient and specific Cre/loxP-like site-specific recombination system. Nucleic Acids Res 2013, 41(2):e37.
9. Karimova M, Splith V, Karpinski J, Pisabarro MT, Buchholz F: Discovery of Nigri/nox and Panto/pox site-specific recombinase systems facilitates advanced genome engineering. Sci Rep 2016, 6:30130.
10. Karimova M, Baker O, Camgoz A, Naumann R, Buchholz F, Anastassiadis K: A single reporter mouse line for Vika, Flp, Dre, and Cre-recombination. Sci Rep 2018, 8(1):14453.

