技术分享:桥接重组酶ISCro4实现兆碱基级人类基因组重排
基因组是承载生物复杂性的基础信息源代码,从编码单个酶活性到协调整个细胞网络、直至协调生物个体层级行为[1]。这种基因型与表型之间的关联,可能由跨越兆碱基级基因组的突变组合所决定,突破了人们对最简单基因组的理解。CRISPR基因组工程等RNA引导技术的革命性发展,使得人们通过可编程DNA改造手段探索生物功能的能力发生了质的飞跃[2]。然而,这些方法依赖于同源定向修复或先导编辑等复杂的多步机制,导致基因组的编辑长度受限[3]。与CRISPR相比,Cre等DNA重组酶虽然可操控数千碱基对的DNA片段,但需要预先设置识别位点才能实现哺乳动物的基因组重组,且在重组后残留疤痕序列[4]。尽管通过融合RNA引导系统或定向进化可改变重组酶的序列特异性,但此类工具大多局限于基因整合,且会留下插入位点疤痕序列[5]。
作为RNA引导的DNA重组酶,桥接重组酶有望兼并CRISPR的灵活特异性和传统重组酶的高载荷量优势[6]。桥接重组酶通过可编程的桥接RNA(bRNA)双特异性识别靶DNA和供体DNA,实现了DNA插入、倒位和切除(图一A)。相比之下,CRISPR相关转座酶通过向导RNA识别靶DNA,需要依赖固定的DNA供体序列[7]。而bRNA包含了可编程的两个环,一个与靶DNA结合(TBL),另一个与供体DNA结合(DBL)。对桥接重组酶IS621的结构研究表明,两个重组酶二聚体分别结合bRNA环区,随后结合同源DNA形成四聚体突触复合物(图一B)。在DNA与重组酶形成共价键后发生链交换,产生霍利迪连接体中间结构,最终解离形成重组产物(图一C)。已有研究成功验证了桥接重组酶改造原核基因组的可行性,下一步需开发能在人类细胞中高效基因重组的桥接重组酶。
2025年9月,Science期刊报道了桥接重组酶直系同源物ISCro4的发现与工程化改造,实现了在人类基因组的插入、倒位及切除。首先,通过改造桥接RNA(bRNA),优化后bRNA体系特异性高达82%。其次,通过深度突变扫描获得增强型三重突变体ISCro4,将高活性突变体与优化bRNA结合,在人类基因组中实现了高达20%的插入效率。再次,运用ISCro4完成了精准且无痕的基因组重排,成功实现0.92 Mb大片段倒位与0.13 Mb大片段切除,且无明显的距离依赖性。最后,通过切除镰状细胞贫血症BCL11A增强子及弗里德希共济失调症相关重复序列,为其临床治疗潜力提供了概念验证[8]。
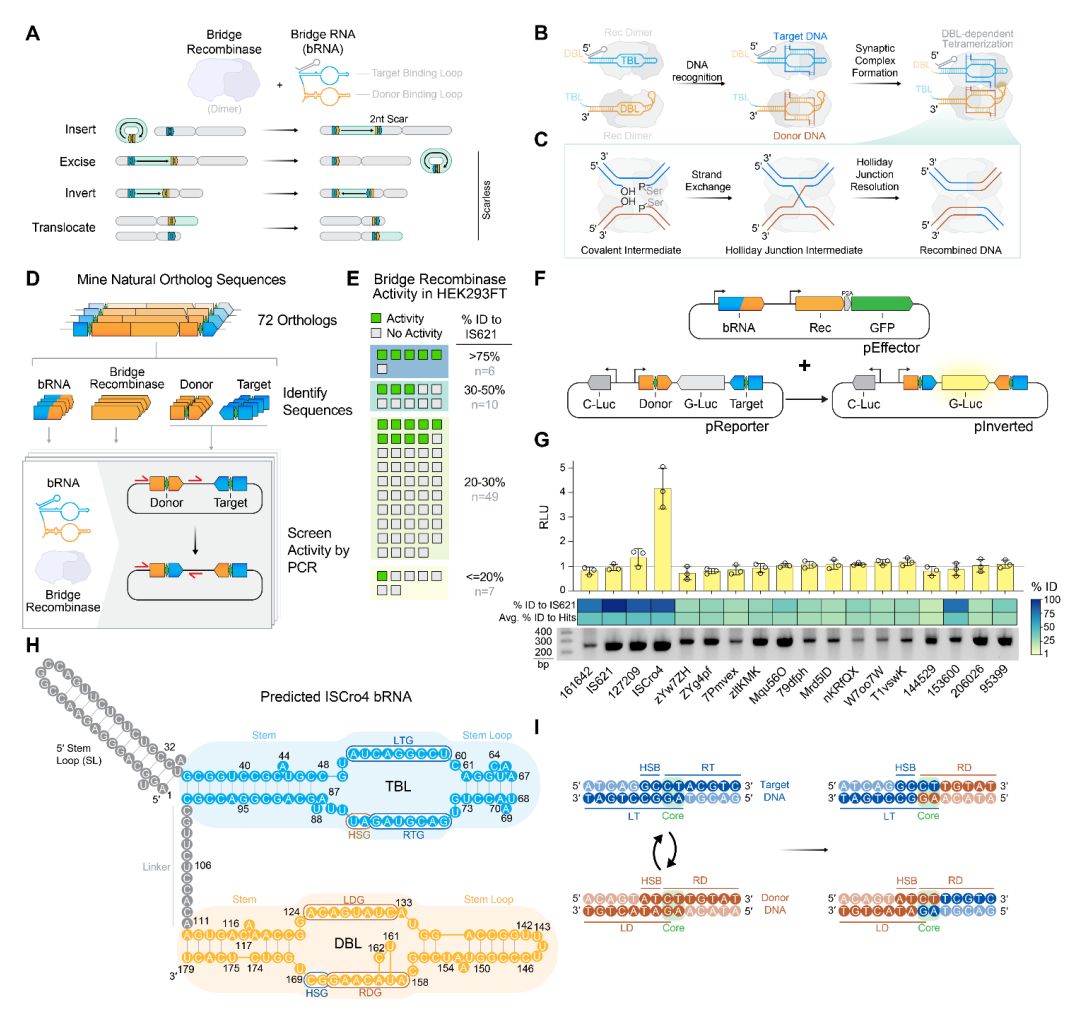
图一 在人类细胞中筛选活性桥接重组酶[8]
基于桥接重组酶IS110元件数据库[6],研究者预测了其他72种桥接重组酶编码序列及其对应bRNA。基于比较基因组学比对IS110元件及其边缘序列,构建了这72种桥接重组酶的天然靶序列和供体序列,并在人胚胎肾293FT细胞中进行倒位报告基因检测筛选(图一D)。其中18种(25%)重组酶检测到倒位连接产物,证实至少部分桥接重组酶在人类细胞中无需原核宿主因子即可发挥作用(图一E)。利用高斯荧光素酶倒位实验对这18种功能性同源酶进行定量分析(图一F),结果显示仅一种同源酶的活性超过检测限,其活性值较背景提升4倍(图一G)。该活性酶来自啮齿柠檬酸杆菌,在ISFinder数据库中命名为ISCro4[9],与已知的IS621高度同源(重组酶相似性88%,bRNA 86.6%)。预测ISCro4能通过bRNA的靶DNA结合环(TBL)和供体DNA结合环(DBL)分别识别靶标和供体DNA底物中的14个核苷酸,在二核苷酸核心序列周围实现保守性重组(图一H和I)。以上数据表明,桥接重组酶ISCro4在人类细胞中展现出活性。
单个bRNA分子同时识别两种DNA底物,但之前IS621的结构研究表明四聚体复合物在重组过程中可能独立使用这两个RNA环。因此研究者尝试分别表达TBL与DBL(图二A)。利用mCherry倒位报告系统对比完整bRNA与两种分裂型bRNA的效果,发现分裂型bRNA不仅保持功能活性,还促使mCherry平均荧光强度(MFI)检测的重组效率提升约2.1倍,且无需5’端的茎环结构(图二B和C)。TBL含有一个12 bp的连续茎区,仅单个碱基(A44)突出,而DBL茎区中间有三个未配对位点,两侧分别为3个和4个配对碱基(图一H)。为此研究者探究增加茎区长度对活性的影响。通过插入碱基与天然未配对连接区互补配对,将TBL茎区延长未观察到活性提升,相比之下,将较短的DBL茎区分别延长6 bp(DBL2)和11 bp(DBL3)后,活性分别达到DBL1的1.3倍和1.4倍(图二D绿色框)。接着测试在两个环区内替换与删除碱基的效果,通过最大化碱基配对数量来增强bRNA的结构稳定性。在分裂型bRNA体系中分别评估各环区的突变效果,发现TBL的替换与删除突变效果更显著,连同A:U→G:C配对修饰(A61G+U73C)进行组合,观察到活性的累加提升,最终获得TBL3,活性达TBL1的2.1倍(图二E)。引入天然5'茎环结构的TBL4可进一步轻微提升活性(图二E)。将完整bRNA改造为包含TBL4和DBL3,并通过GC二核苷酸连接区衔接。在完整bRNA体系中观察到活性提升3.8倍(图二F和G),将其命名为增强型bRNA(图二H)。在分裂型bRNA体系中,TBL4+DBL3组合也是最佳,活性较WT bRNA提升6.4倍(图二F和G)。
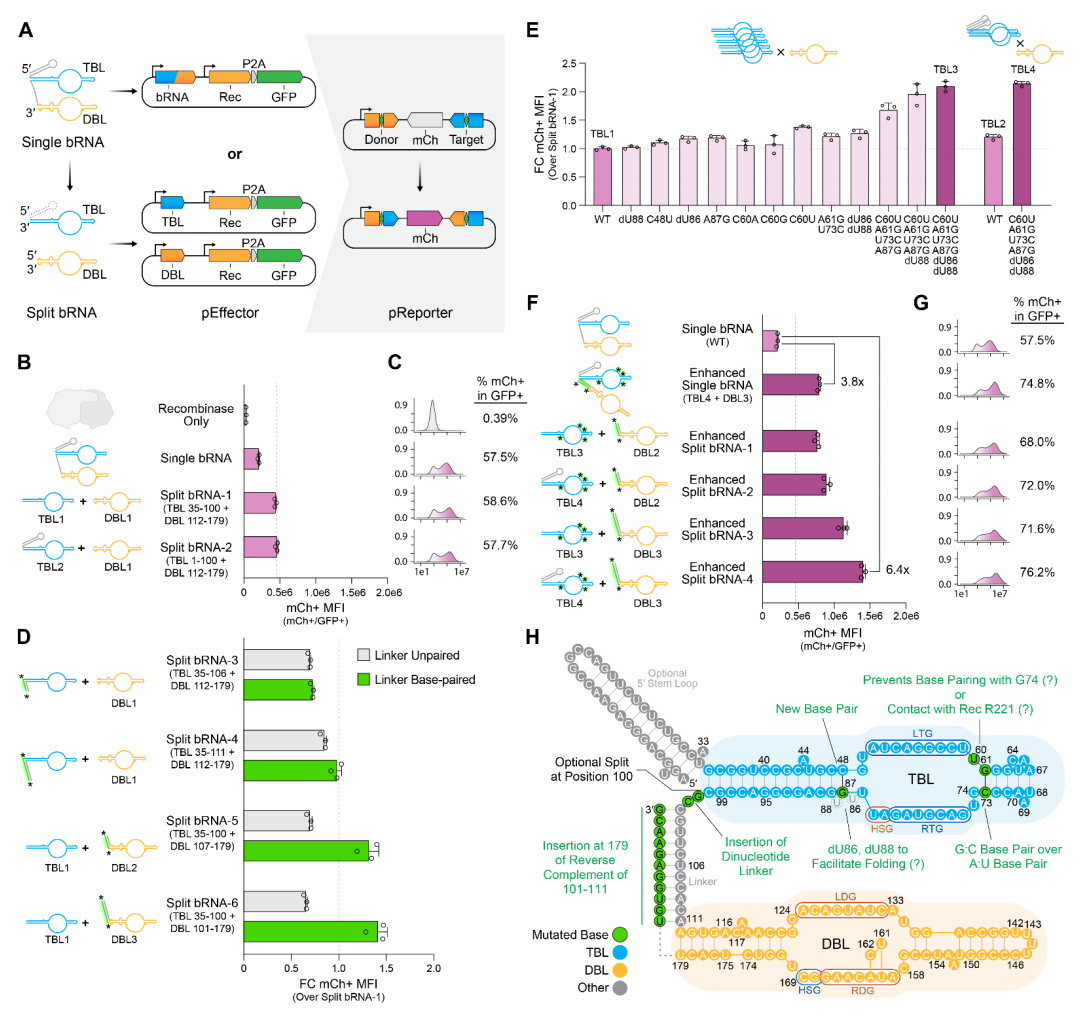
图二 工程化改造bRNA实现高效重组[8]
ISCro4的bRNA天然识别的14核苷酸靶序列仅在人类基因组中出现一次,这为序列特异性插入提供了理想测试模型。研究者将携带重组酶与WT bRNA的质粒以及含有WT供体序列和嘌呤霉素筛选质粒共转染至人类细胞(图三A)。通过深度测序检测全基因组范围内的相对插入效率,发现所有插入位点的共有序列与供体序列高度匹配,而非预期靶标序列,且91.3%的插入事件与质粒供体序列的相似度高于预期基因组靶标(图三B和C)。之前结构和生化研究表明,IS621介导的这种"供体-供体"反应很可能是由不同bRNA的两个DBL所介导的转座体,而非一个TBL和一个DBL所导致(图三D)[10]。DBL-DBL转座体的形成可能是DBL独特的茎环结构促使四聚体的稳定性。采用mCherry倒位报告系统比较WT bRNA介导的"靶标-供体"与"供体-供体"重组效率,确认了两类重组均可有效发生,并且单一的DBL也足以介导高效重组(图三E和F)。
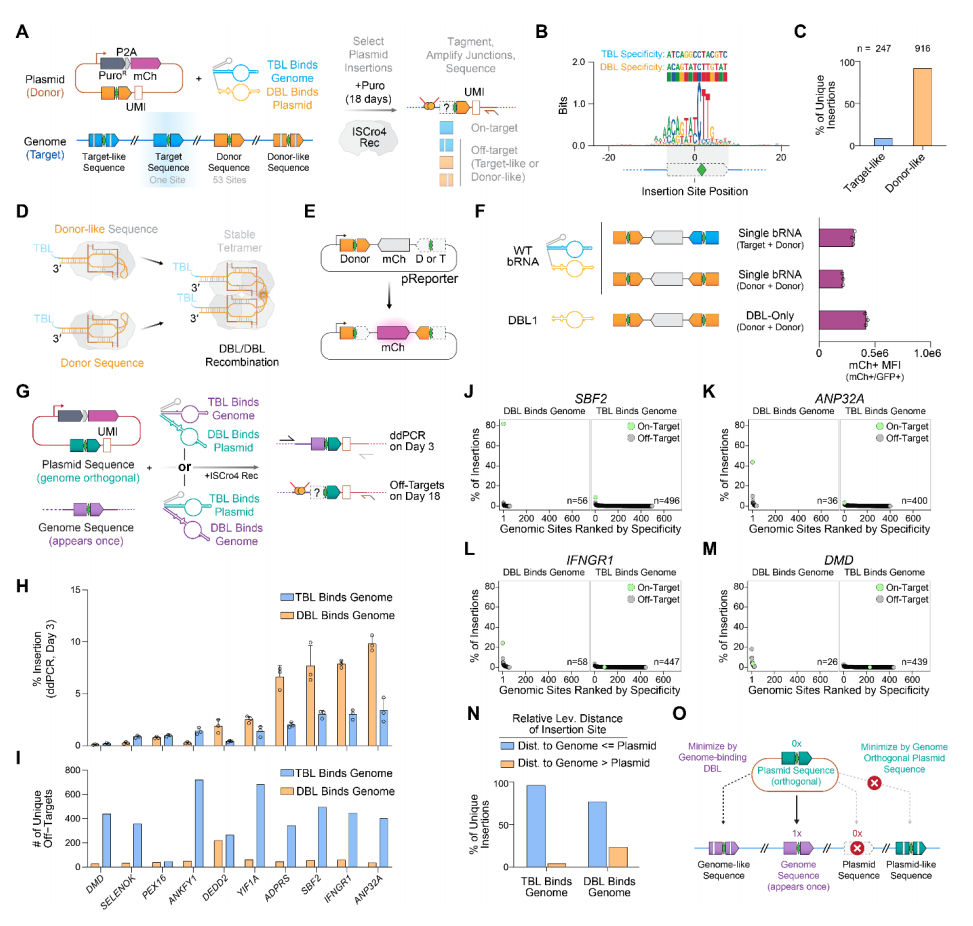
图三 DBL与基因组的结合可实现高特异性插入[8]
为促进靶向插入并减少仅由DBL介导的重组,研究者实施三项改进:1)在递送的质粒供体上编码基因组中不存在的独特bRNA识别序列,即基因组正交序列;2)采用增强型bRNA;3)使DBL识别基因组序列,TBL识别供体质粒序列。为验证该策略,研究者筛选了10个含独特14 nt序列的基因组位点,并与嘌呤霉素抗性质粒中的基因组正交供体序列相配对(图三G)。每个位点设计两种增强型bRNA:一种由TBL识别基因组而DBL识别质粒,另一种则相反。在转染后第三天通过液滴数字聚合酶链式反应(ddPCR)评估两种方向的重组效率,发现TBL结合基因组的插入效率为0.18-3.38%,而DBL结合基因组为0.10-9.82%(图三H),且有8个位点的DBL靶向基因组的特异性显著优于TBL,表现为显著降低了脱靶率(图三I),在4个位点观察到稳定的靶向插入,包括三个最高效位点SBF2、ANP32A和IFNGR1(图三J-M)。进一步分析表明,在质粒供体上编码基因组正交序列还能有效消除两种bRNA靶向的类质粒脱靶事件(图三N)。以上实验结果筛选出在平衡特异性的同时实现效率最大化的策略,即在供体质粒上使用基因组正交序列以及设计DBL靶向基因组,是实现ISCro4基因组插入的最佳方案(图三O)。此外,通过深度突变扫描,研究者筛选到ISCro4的S30T、P54Q和S243H突变组合成一个三重突变体,活性提升约1.5倍。
桥接重组酶的独特优势在于可设计识别几乎任意基因组序列对,无需如其他方法般预先设置重组酶的识别位点[11]。为在人类细胞中验证该功能,研究者筛选基因组中独特的序列对,要求其核心序列匹配而第6、7位点不匹配。根据两序列的相对链方向,位点间重组可产生基因组内倒位或切除,通过标签化建库与扩增子测序进行定量(图四A)。设计了两种增强型bRNA(TBL结合位点1以及DBL结合位点2,或者互换),搭配野生型ISCro4重组酶,分别检测了七组倒位和七组切除,成功实现最长倒位929,524 bp(0.92 Mb,效率5.62%;图四B)与最长切除134,143 bp(0.13 Mb,效率0.80%;图四C)。在两类基因组重组实验中,再次观察到bRNA的靶向会影响结果,表明重组效率主要取决于重组酶和bRNA本身的特性,而非特定序列对及其基因组背景。
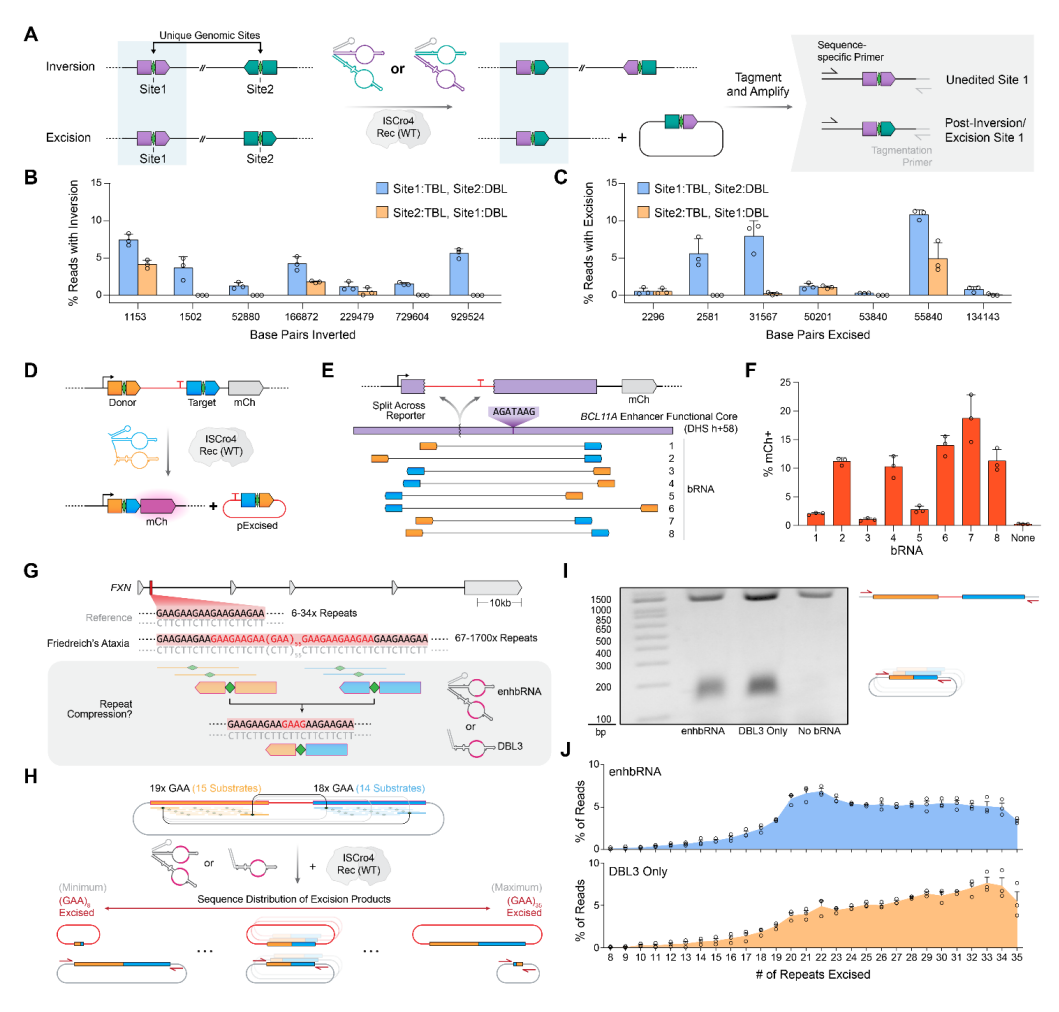
图四 ISCro4介导基因组重组的应用[8]
桥接重组技术可编程切除特定基因组区间的能力,为高分辨率功能基因组学研究开辟了新途径。转录因子BCL11A的增强子对其表达至关重要,在红细胞中靶向敲除该区域已成为治疗镰状细胞贫血和β地中海贫血的疗法[12]。作为概念验证,研究者采用包含BCL11A增强子区的报告质粒,切除后才能表达mCherry,从而定量检测切除效率(图四D和E)。通过对TBL和DBL结合位点的多种组合测试,实现了最高18.7%的稳健切除效率(图四F),表明桥接重组技术介导的切除适用于用户自定义的基因组区间。
弗里德希共济失调是一种神经肌肉疾病,由FXN基因内含子1中GAA三核苷酸重复序列扩增引起(图四G)。健康个体该重复序列仅6次,而患者可多达1700次[13]。运用桥接重组酶切除过量重复序列可实现无疤痕且保留阅读框的精准操作,从而恢复正常基因型。然而此类重组需要TBL和DBL同时结合相同底物,可能导致双环相互竞争抑制。为此研究者探索能否单独使用DBL重组(图三F)来清除异常重复区域。设计增强型bRNA的双环或单一增强型DBL3识别跨越5个GAA重复单元的14核苷酸序列。任意两个潜在结合位点之间均可发生重组,最终导致不同范围的重复序列切除,从而产生不同分子的多样化结果(图四G)。为在报告质粒体系中验证该策略,研究者在序列两侧设置GAA重复区域,对比完整bRNA与单一DBL的切除效率(图四H)。通过该区域扩增定量检测完整与切除报告基因的比例(图四I)。正如预期,bRNA组与单一DBL组均观察到一系列不同结果,质粒中被切除的重复单元数最少8个,最多达35个。值得注意的是,单一DBL在切除更多重复单元方面表现更优,40%发生切除的质粒中超过80%(≥29个)重复单元被清除(图四J)。
综上所述,该研究首次开发了可在人类基因组中进行插入、倒位及切除的桥接重组酶ISCro4。重点阐述其关键优化策略,特别是利用桥接RNA(bRNA)的供体结合环(DBL)与基因组进行碱基配对,揭示了两个bRNA结合环之间功能与机制的差异。通过系统性工程化改造,增强型ISCro4系统显著提升了bRNA与重组酶的活性,利用人类基因组中合适的特异性序列对,即可实现高达20%的插入效率,以及无痕编辑兆碱基尺度的基因组DNA重排。该研究提供了一种新的基因座与染色体尺度的基因组重组技术,无需预先设置识别位点就能进行任意序列间的DNA重组,为解析生物体的序列与功能关系、理解细胞功能与人类疾病机制开辟了新的路径。

GTP研发中心拥有自主知识产权的类精子干细胞介导的半克隆技术,可在体外实现类精子干细胞的多位点改造并进行功能元件测试,最后将类精子干细胞通过卵母细胞注射可以一步法获得基因改造小鼠。基于类精子干细胞单倍体打靶高效率的优势,可快速构建定制化小鼠,如基因组安全位点大片段敲入、条件性基因敲除/敲入和人源化基因改造等。多个复杂调控元件可以通过多次体外细胞打靶构建在同一株类精子干细胞中,经细胞水平质控后,一步法获得基因改造小鼠。

如有需要欢迎联系我们,GTP研发中心将竭诚为您设计最佳方案,制定专属于您的基因改造小鼠。
网址:http://www.sibcb.ac.cn/gtp/
地址:上海市岳阳路320号
责任编辑:甜点
新媒体运营:suway
参考文献
1. Hartwell LH, Hopfield JJ, Leibler S, Murray AW: From molecular to modular cell biology. Nature 1999, 402(6761 Suppl):C47-52.
2. Hsu PD, Lander ES, Zhang F: Development and applications of CRISPR-Cas9 for genome engineering. Cell 2014, 157(6):1262-1278.
3. Liao H, Wu J, VanDusen NJ, Li Y, Zheng Y: CRISPR-Cas9-mediated homology-directed repair for precise gene editing. Mol Ther Nucleic Acids 2024, 35(4):102344.
4. Pinglay S, Lalanne JB, Daza RM, Kottapalli S, Quaisar F, Koeppel J, Garge RK, Li X, Lee DS, Shendure J: Multiplex generation and single-cell analysis of structural variants in mammalian genomes. Science 2025, 387(6733):eado5978.
5. Anzalone AV, Gao XD, Podracky CJ, Nelson AT, Koblan LW, Raguram A, Levy JM, Mercer JAM, Liu DR: Programmable deletion, replacement, integration and inversion of large DNA sequences with twin prime editing. Nature biotechnology 2022, 40(5):731-740.
6. Durrant MG, Perry NT, Pai JJ, Jangid AR, Athukoralage JS, Hiraizumi M, McSpedon JP, Pawluk A, Nishimasu H, Konermann S et al: Bridge RNAs direct programmable recombination of target and donor DNA. Nature 2024, 630(8018):984-993.
7. Lampe GD, King RT, Halpin-Healy TS, Klompe SE, Hogan MI, Vo PLH, Tang S, Chavez A, Sternberg SH: Targeted DNA integration in human cells without double-strand breaks using CRISPR-associated transposases. Nat Biotechnol 2024, 42(1):87-98.
8. Perry NT, Bartie LJ, Katrekar D, Gonzalez GA, Durrant MG, Pai JJ, Fanton A, Martins JQ, Hiraizumi M, Ricci-Tam C et al: Megabase-scale human genome rearrangement with programmable bridge recombinases. Science 2025:eadz0276.
9. Siguier P, Perochon J, Lestrade L, Mahillon J, Chandler M: ISfinder: the reference centre for bacterial insertion sequences. Nucleic Acids Res 2006, 34(Database issue):D32-36.
10. Hiraizumi M, Perry NT, Durrant MG, Soma T, Nagahata N, Okazaki S, Athukoralage JS, Isayama Y, Pai JJ, Pawluk A et al: Structural mechanism of bridge RNA-guided recombination. Nature 2024, 630(8018):994-1002.
11. Yarnall MTN, Ioannidi EI, Schmitt-Ulms C, Krajeski RN, Lim J, Villiger L, Zhou W, Jiang K, Garushyants SK, Roberts N et al: Drag-and-drop genome insertion of large sequences without double-strand DNA cleavage using CRISPR-directed integrases. Nat Biotechnol 2023, 41(4):500-512.
12. Frangoul H, Altshuler D, Cappellini MD, Chen YS, Domm J, Eustace BK, Foell J, de la Fuente J, Grupp S, Handgretinger R et al: CRISPR-Cas9 Gene Editing for Sickle Cell Disease and β-Thalassemia. N Engl J Med 2021, 384(3):252-260.
13. Bürk K: Friedreich Ataxia: current status and future prospects. Cerebellum Ataxias 2017, 4:4.
转载请注明来源于【GTP研发中心】
声明:本文仅用于分享,不代表GTP研发中心立场,若有侵权,请及时联系我们第一时间更正!



