技术分享:人类蛋白质互作图谱预测模型
蛋白质是细胞的重要组成成分和功能执行者。解析蛋白质的相互作用伙伴及其蛋白质复合物的三维结构,对于理解蛋白质的功能至关重要[1]。尽管已有酵母双杂交和亲和纯化质谱等大规模实验方法用于鉴定人类蛋白质组规模的蛋白质-蛋白质相互作用(PPI),但它们在非生理条件下检测PPI时,仍存在较高的假阳性和假阴性问题[2]。以人类蛋白质互作组学为例,预测包含了7.4万至20万对PPI,而来自UniProt、BioGrid和STRING等PPI数据库的实验数据却提示可能存在超过100万对潜在的人类PPI[3]。值得注意的是,这些数据库中高置信度PPI的重合度较低,仅有3988对PPI被三家数据库共同认定为高置信度的相互作用(图一A)。
为解析人类蛋白质互作组学,科研人员开发了多种计算方法来辅助实验研究,例如通过分析已知相互作用伙伴的同源性、预测接触面的算法以及蛋白质间的功能联系来预测PPI[4]。PPI接触面残基的协同进化分析已与AlphaFold(AF)、RoseTTAFold(RF)等三维结构预测技术相结合,已成功在细菌和酵母中实现蛋白质组规模的PPI鉴定[5]。主要通过三个步骤来评估PPI概率:1)构建两个蛋白质的跨物种直系同源蛋白的多序列比对(MSA);2)将同物种的序列拼接成配对MSA(pMSA);3)基于协同进化与复合物结构建模来推断这两个蛋白质间的残基-残基相互作用概率[6]。尽管类似方法已用于预测特定人类蛋白质的相互作用,但由于庞大的计算规模和有限的动物基因组数量,人类蛋白质组规模的从头筛选PPI仍然面临挑战[7]。前期开发的轻量化深度学习网络尽管在细菌和酵母的快速PPI筛选中表现优异,但在人类PPI预测中的精度显著下降[8]。虽然AlphaFold2精度更高,但其巨额计算成本使其难以应用于蛋白质组尺度的研究。
2025年10月,Science期刊发布了迄今为止最全面的人类蛋白质-蛋白质相互作用(PPI)的高精度预测模型,系统性描绘了人类蛋白质互作图谱。首先,通过收集30 PB未拼接的21,415种真核生物的基因组数据,将多序列比对(MSA)的深度提高7倍,显著增强了协同进化分析信号。其次,基于2亿个预测蛋白质结构的结构域相互作用数据集,开发了新的深度学习网络。由此,系统性预测出17,849对高置信度PPI,其中3,631对为首次发现的全新互作关系,这些预测的蛋白质复合物为了解人类生物学与疾病机制提供了丰富见解[9]。
为提高人类PPI预测的精度,增强协同进化分析的信号,需解决现有各物种蛋白质注释不足的问题。生物学家的研究通常依赖于现有数据库中已注释的蛋白质序列,然而在各个物种的原始基因组测序数据中标注蛋白质序列信息却不是个常规工作。从2024年6月NCBI数据库上收集的36,840种真核生物基因组,仅有约20%的7,355个物种被注释蛋白质信息,并且可能还存在拼接与注释不全或错误等问题。于是,研究者挖掘30 PB的NCBI基因组与SRA数据库,从21,415种真核生物中提取全基因组与全转录组数据集,重点关注高等真核生物中的脊索动物与节肢动物两大门类。通过开发新型生物信息学流程,利用剪接位点识别的序列比对工具和模式生物参考蛋白集[10],从各数据集中组装蛋白编码序列(图一B)。所有预测蛋白序列均与人类直系同源蛋白进行比对,并采用双向最佳匹配标准区分直系/旁系同源物。将最终比对结果命名为omicMSA,涵盖21,415个物种,涉及9,905属、2,727科、626目。相比UniRef100数据库中仅含的3,082个物种,omicMSA显著拓展了蛋白质序列的分类学多样性(图一C),深度也比UniRef100提升了数倍(图一D)。将omicMSA与其他常用策略构建的MSA进行比较,发现omicMSA在所有测试方法中均表现最佳(图一E-G,红色曲线)。以上数据表明,omicMSA利用更广泛的物种分类数据,获取了更强的协同进化分析的信号,提升了PPI预测的准确率。
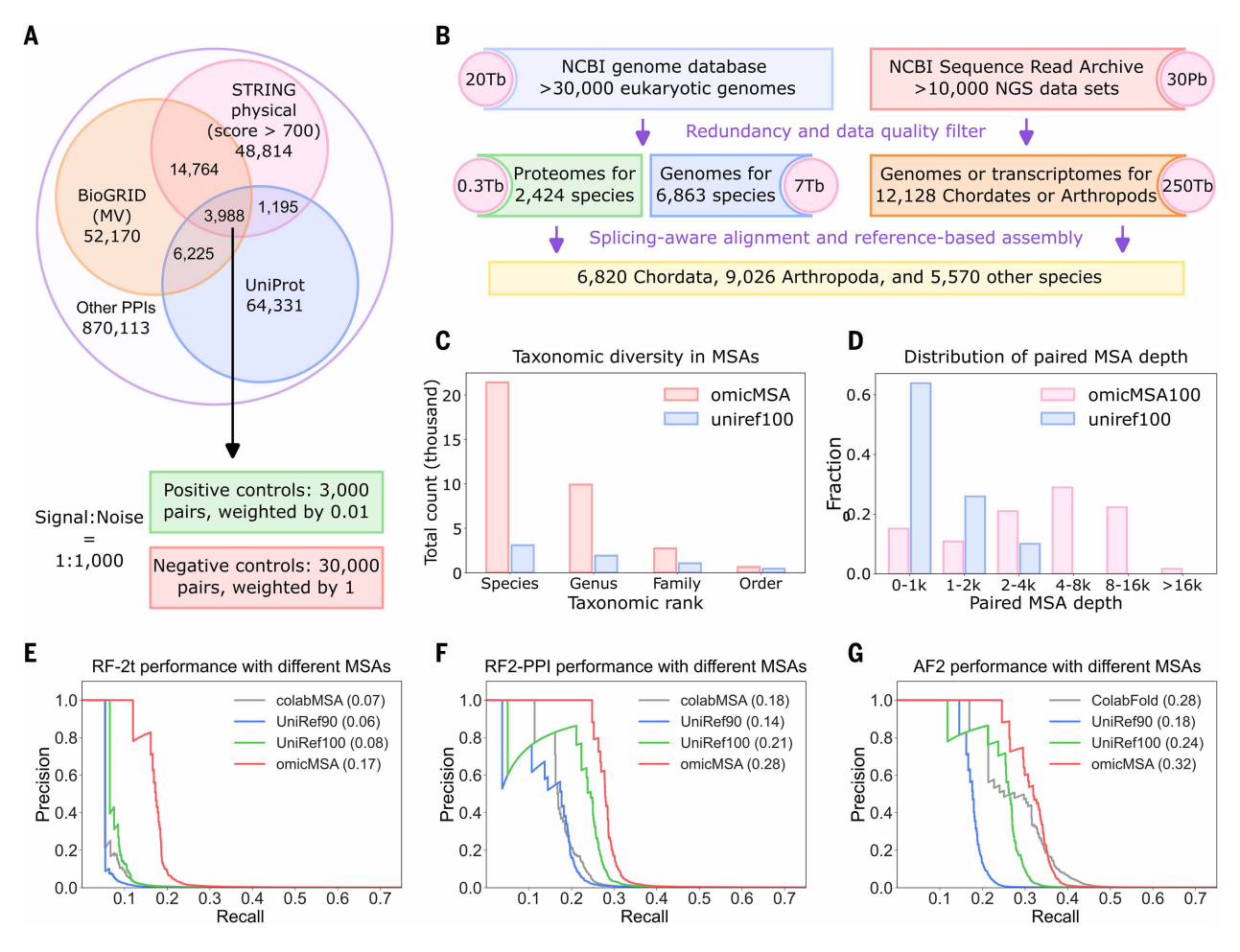
图一 omicMSA显著增强协同进化分析信号,提升PPI预测准确率[9]
为提升蛋白质复合物三维结构建模能力,AlphaFold-multimer与AlphaFold3均采用PDB数据库中的PPI数据进行训练。然而,此类训练策略与推断目标未必能增强深度学习网络从非相互作用蛋白质中区分出真实PPI的能力。在序列一致性低于30%的阈值下,PDB数据库(2023年12月)共有24,358个异源PPI聚类,其中包含了大量人类蛋白质及其近缘同源物,剔除这些复合物后仅剩13,231个有效的异源PPI聚类(图二A左),这一训练数据规模严重制约了模型的判别能力。根据蛋白质结构特征和PDB条目分类结构域的同源性,研究者先前开发了一种方法,将AF2模型分割成结构域,用于重新整合InterPro中UniProt条目的结构特征和结构域注释(图二B)。在过滤掉50%序列相似性后,从5370万个AFDB模型中,识别出1240万个的多结构域蛋白,共计包含2240万个高质量的结构域-结构域对。通过筛选至少具有25个残基接触且相互作用置信度高的结构域对,最终获得100万对结构域-结构域相互作用(DDI)。在序列一致性低于30%的阈值下,去除人类蛋白近缘同源物后,得到了223,440个DDI聚类,规模达PDB PPI训练集的17倍(图二A右)。

图二 从AFDB提取的DDI数据用于深度学习网络,快速实现PPI预测[9]
基于RF2架构,研究者联合PDB PPI与AFDB DDI数据集开发了RF2-PPI,整合了MSA、相互作用残基和三维结构等特征(图二C)。在识别真实PPI方面,RF2-PPI相比先前的RF2-lite提升了1.7倍(图二D)。当与omicMSA结合使用时,RF2-PPI略优于广泛使用的ColabFold(图二D)。尽管AFmm和AF3在蛋白质复合物三维结构建模方面表现卓越,但在区分真实PPI与随机配对时性能远逊于RF2-PPI(图二D)。AF2与omicMSA结合使用能获得最佳性能(图二D)。RF2-PPI和AF2的预测性能均与接触面大小相关,大接触面的强相互作用更易被准确预测(图二E)。为改进计算效能,研究者通过减少RF2-PPI中类Evoformer模块数量并优化数据处理流程,最终使其速度较前代RF2-lite提升50%,比AF2快约20倍(图二F)。于是,研究者采用RF2-PPI与AF2协同策略:前者负责大规模蛋白质组PPI筛查,后者进一步为候选PPI生成高质量三维结构模型。
研究者对19,528个蛋白质形成的1.91亿对蛋白质进行PPI无偏搜索。为了使搜索更容易处理,根据UniProt细胞室(CC)注释,将分析范围限制在位于同一细胞室内的蛋白质,以及亚细胞定位未知的未被充分研究的蛋白质。通过直接耦合分析(DCA)、RF2-PPI和AF2,依次分析了5380万对同一细胞室蛋白质和5740万对未被注释定位的蛋白质(图三A)。经RF2-PPI预测获得190万对RFIntProb>0.3的候选对(图三B中),再经AF2生成三维模型并计算AFIntProb,最终在80%预估精度下获得6,763对预测PPI(图三B)。预测随机配对蛋白质的高置信度相互作用需要采用接近0.99的严格阈值(图三C和D),而存在先验证据时,RFIntProb或AFIntProb阈值可显著降低且保持同等置信水平。基于STRING的523万遗传关联对,研究者以80%精度预测出9,428对PPI,90%精度下为7,201对(图三A)。基于UniProt、BioGRID和STRING的物理相互作用先验证据,以80%精度预测出21,960对PPI,90%精度下为14,129对PPI(图三A)。综上,共计获得29,256对预估精度80%的PPI,相当于覆盖人类蛋白质互作组的12-32%,其中17,849对PPI达到90%预估精度。研究者后续深度分析该高置信度预测集,并将全部预测数据共享至http://prodata.swmed.edu/humanPPI。
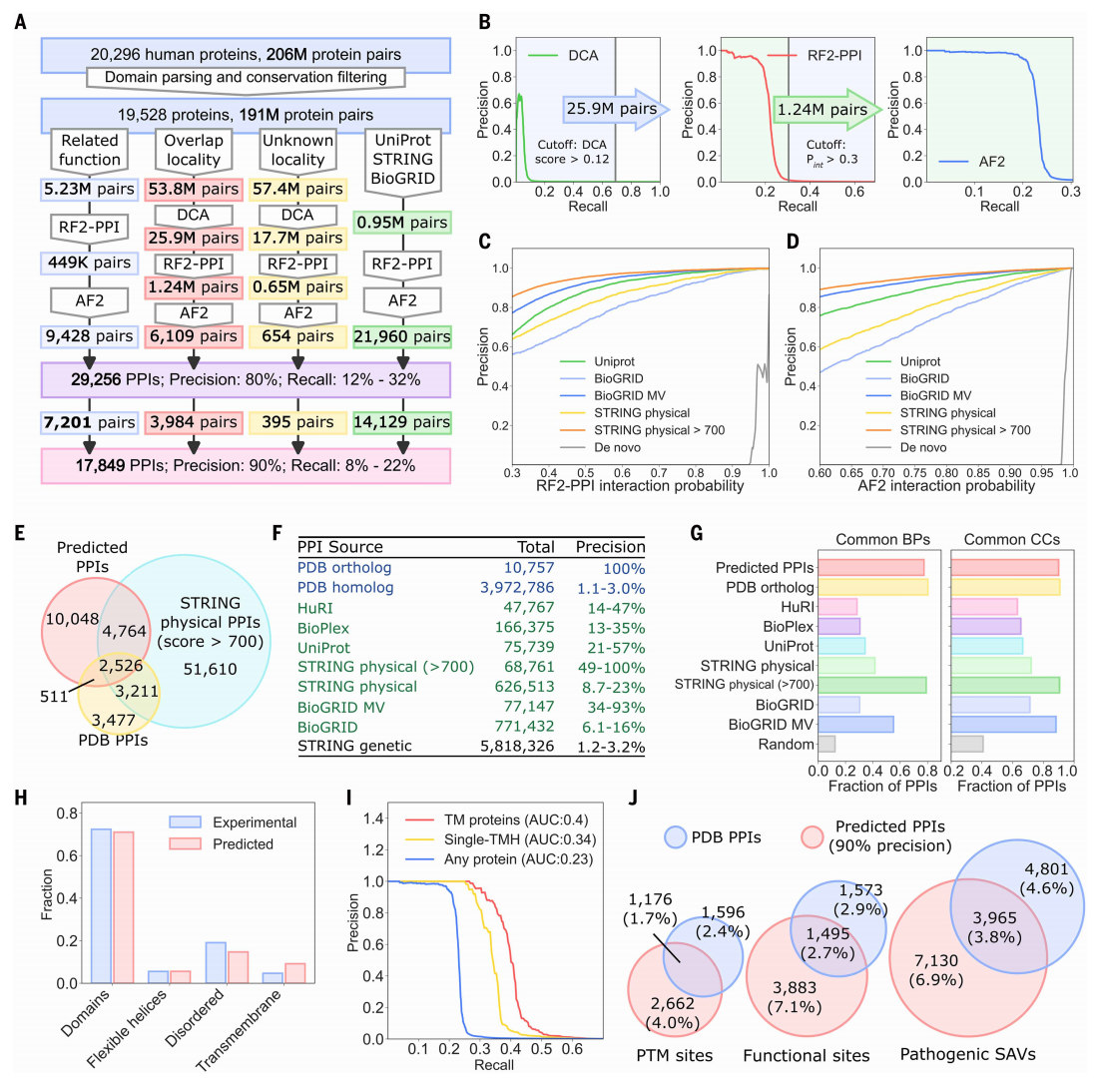
图三 全蛋白质组范围的PPI筛选流程及其统计结果[9]
将预测结果与其他PPI数据库进行对比(图三E),分析显示直系同源PDB模板的PPI几乎全为真实相互作用(图三F)。预测PPI的生物学功能和亚细胞定位与直系同源PDB模板PPI最为相似(图三G)。通过将预测PPI的结构特性与9,725个直系同源PDB模板的人类PPI对比发现,大部分预测PPI主要由球状结构域介导,而结构域间连接子介导的PPI在预测中占比低于PDB复合物(图三H)。与实验确定的蛋白质复合物相比,预测的PPI中涉及跨膜螺旋(TMH)的比例更高(图三H)。通过对跨膜蛋白(TMP)配对进行测试,该方法能以更高精度预测更多跨膜蛋白相互作用(图三I)。数千个翻译后修饰(PTM)位点和功能位点(活性中心及底物/辅因子结合位点)定位于实验确定的和预测的PPI接触面上,分别占人类蛋白质中所有此类注释位点的8.1%和12.7%(图三J)。以及约1.6万个(15.3%)疾病相关单氨基酸变异(SAV)被定位至预测或实验确定的PPI接触面上(图三J)。以上数据表明,PPI的破坏是遗传性疾病的常见机制,该预测数据可作为阐释人类疾病分子基础的重要资源。
在高置信度预测的17,849对PPI中,3,037对(17%)在直系同源PDB模板,另有11,181对(63%)存在于PPI数据库,其余3,631对(20%)为未被收录的全新相互作用,其中有1,413对PPI涉及功能未知的蛋白质(图四A)。优先选取2,700对既未收录于PPI数据库也无同源PDB模板的PPI进行深度分析,通过UniProt关键词进行功能分类,发现这些未知PPI在特定功能类别中显著富集(图四B)。聚焦其中几个功能类别,这些预测揭示了未被认知的功能机制,助力解析疾病发生机理(图四C-H)。例如,预测到G蛋白偶联受体143与BLOC-3复合体组分HPS1之间存在相互作用(图四C),C5a过敏毒素趋化受体2(C5AR2)与中性粒细胞弹性蛋白酶(ELANE)之间存在相互作用(图四D),去泛素化酶OTUD4与E3泛素连接酶ASB8之间存在直接相互作用(图四E)。预测到其他未表征PPI涉及在特定免疫细胞分化和活化中起作用的蛋白质。例如,杀伤细胞凝集素样受体G亚家族成员1(KLRG1)是一种跨膜受体,可抑制自然杀伤细胞和效应T细胞的活性。预测显示,KLRG1的胞外结构域与Toll样受体3(TLR3)发生相互作用,并且一种罕见遗传性免疫缺陷病相关的SAV位于KLRG1与TLR3的相互作用界面(图四F红框)。超过半数先前未知的相互作用(2,700对中的1,556对)涉及跨膜蛋白,它们常位于细胞器如线粒体、内质网、高尔基体或溶酶体中(图四B)。在线粒体中预测到许多PPI与构成呼吸电子传递链的线粒体复合物组装有关。例如,发现细胞色素c氧化酶亚基2(MT-CO2,复合体IV的一个亚基)与细胞色素c氧化酶组装蛋白COX20(已知的复合体IV组装因子)之间具有相互作用(图四G)。未表征蛋白C8orf82与由ETFA和ETFB组成的电子转移黄素蛋白(ETF)复合物预测到存在相互作用(图四H),表明C8orf82可能作为分子伴侣促进ETF复合物组装,或在特定代谢条件下作为调节亚基调控ETF活性。位于预测PPI接触面的疾病相关SAV(图四G和H)为这些相互作用的功能相关性提供了支持。以上数据表明,这些预测PPI可以为人类疾病分子机制研究提供新的视角。

图四 预测PPI提供蛋白质功能新见解[9]
最后,通过整合预测结果和直系同源PDB模板的PPI,研究者成功预测出404个多蛋白复合物,其中每个组分至少都与两个其他组分存在相互作用。可将这些复合物中的PPI划分为三类:i)具有实验结构数据的相互作用;ii)PPI数据库中的高置信度条目;iii)缺乏或仅有薄弱实验证据支持的相互作用。重点关注第三类,通过预测这些复合物的存在并构建其三维结构,为未来研究开辟了新的方向。例如,预测微管蛋白聚谷氨酰化酶(TPG)还存在三个新的亚基:TBC1D19、CSTPP1和SANBR,每个预测亚基均与多个已知TPG组分存在相互作用(图五A)。另一个由NOL10、UTP25、DDX47、ESF1、ABT1和DDX49组成的复合体,可能将基础转录调控与核糖体生物发生联系起来(图五B)。还预测了多个由纤毛与鞭毛生物发生相关蛋白组成的复合体(图五C)。预测到Complex Portal数据库中139个已知人类蛋白质复合体的新增组分,每个新增组分与已知复合体的至少两个亚基存在相互作用,并且主要与该复合体发生直接作用。例如,预测参与rRNA加工的核蛋白LYAR与端粒维持复合体的两个蛋白DKC1和GAR1存在相互作用(图五D)。还为糖基磷脂酰肌醇-N-乙酰葡糖胺转移酶(GPI-GnT)复合体鉴定出一个新的亚基(图五E)。以及预测到MKS过渡区复合体的一个新的组分,通过整合该复合体各组分的预测PPI,成功构建了横跨胞外区、跨膜区与胞质区的大型复合体模型(图五F)。
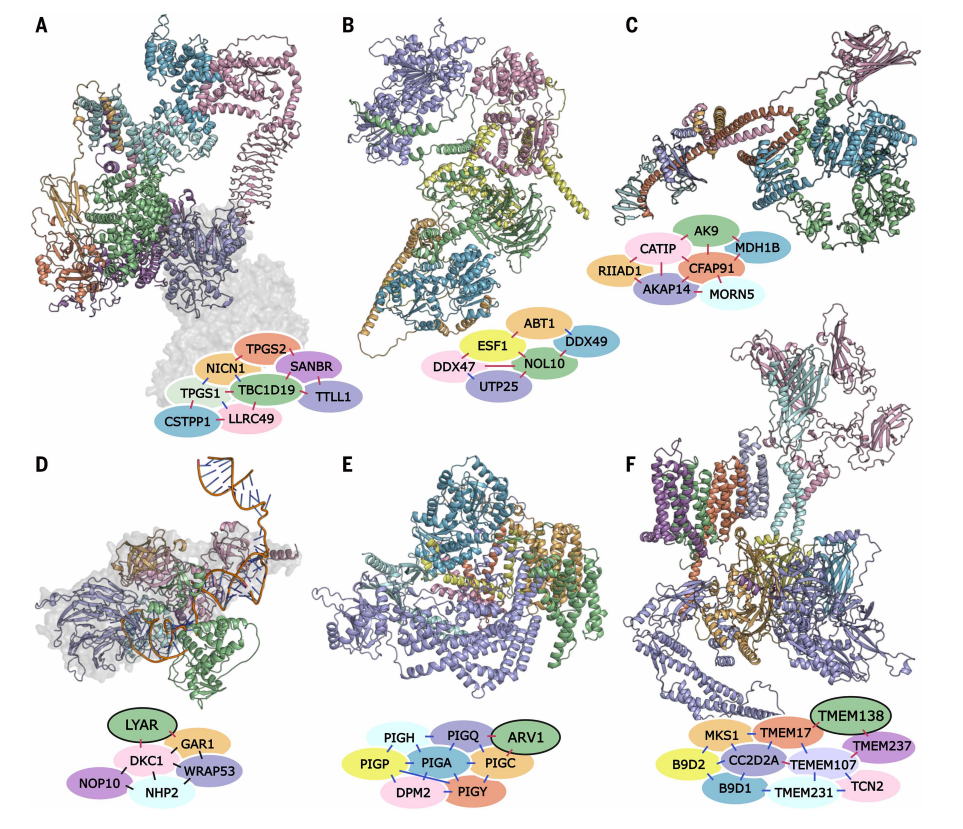
图五 预测高阶寡聚蛋白质复合物[9]
综上所述,该研究开发了一个可扩展的计算流程,基于海量的数据,通过整合协同进化分析与深度学习网络技术,成功实现了从2亿对可能的人类蛋白质组合中预测出1.7万对高置信度的蛋白质-蛋白质相互作用(PPI),发布了迄今为止最全面的人类蛋白质互作图谱。不仅大幅拓宽对蛋白质相互作用网络的认识,更为理解人类生物学、解析疾病机制、推动精准医疗提供了重要的数据资源。

GTP研发中心拥有自主知识产权的类精子干细胞介导的半克隆技术,可在体外实现类精子干细胞的多位点改造并进行功能元件测试,最后将类精子干细胞通过卵母细胞注射可以一步法获得基因改造小鼠。基于类精子干细胞单倍体打靶高效率的优势,可快速构建定制化小鼠,如基因组安全位点大片段敲入、条件性基因敲除/敲入和人源化基因改造等。多个复杂调控元件可以通过多次体外细胞打靶构建在同一株类精子干细胞中,经细胞水平质控后,一步法获得基因改造小鼠。

如有需要欢迎联系我们,GTP研发中心将竭诚为您设计最佳方案,制定专属于您的基因改造小鼠。
网址:http://www.sibcb.ac.cn/gtp/
地址:上海市岳阳路320号
责任编辑:甜点
新媒体运营:suway
参考文献
1. Rolland T, Taşan M, Charloteaux B, Pevzner SJ, Zhong Q, Sahni N, Yi S, Lemmens I, Fontanillo C, Mosca R et al: A proteome-scale map of the human interactome network. Cell 2014, 159(5):1212-1226.
2. Rao VS, Srinivas K, Sujini GN, Kumar GN: Protein-protein interaction detection: methods and analysis. Int J Proteomics 2014, 2014:147648.
3. Szklarczyk D, Kirsch R, Koutrouli M, Nastou K, Mehryary F, Hachilif R, Gable AL, Fang T, Doncheva NT, Pyysalo S et al: The STRING database in 2023: protein-protein association networks and functional enrichment analyses for any sequenced genome of interest. Nucleic Acids Res 2023, 51(D1):D638-d646.
4. Zhao H, Petrey D, Murray D, Honig B: ZEPPI: Proteome-scale sequence-based evaluation of protein-protein interaction models. Proceedings of the National Academy of Sciences of the United States of America 2024, 121(21):e2400260121.
5. Humphreys IR, Zhang J, Baek M, Wang Y, Krishnakumar A, Pei J, Anishchenko I, Tower CA, Jackson BA, Warrier T et al: Essential and virulence-related protein interactions of pathogens revealed through deep learning. bioRxiv 2024.
6. Cong Q, Anishchenko I, Ovchinnikov S, Baker D: Protein interaction networks revealed by proteome coevolution. Science 2019, 365(6449):185-189.
7. Burke DF, Bryant P, Barrio-Hernandez I, Memon D, Pozzati G, Shenoy A, Zhu W, Dunham AS, Albanese P, Keller A et al: Towards a structurally resolved human protein interaction network. Nat Struct Mol Biol 2023, 30(2):216-225.
8. Humphreys IR, Zhang J, Baek M, Wang Y, Krishnakumar A, Pei J, Anishchenko I, Tower CA, Jackson BA, Warrier T et al: Protein interactions in human pathogens revealed through deep learning. Nat Microbiol 2024, 9(10):2642-2652.
9. Zhang J, Humphreys IR, Pei J, Kim J, Choi C, Yuan R, Durham J, Liu S, Choi HJ, Baek M et al: Predicting protein-protein interactions in the human proteome. Science 2025:eadt1630.
10. Li H: Protein-to-genome alignment with miniprot. Bioinformatics 2023, 39(1):btad014.
11. Hauser AS, Attwood MM, Rask-Andersen M, Schiöth HB, Gloriam DE: Trends in GPCR drug discovery: new agents, targets and indications. Nat Rev Drug Discov 2017, 16(12):829-842.
12. Genova M, Grycova L, Puttrich V, Magiera MM, Lansky Z, Janke C, Braun M: Tubulin polyglutamylation differentially regulates microtubule-interacting proteins. Embo j 2023, 42(5):e112101.



