技术分享:迭代重组酶技术实现超大片段DNA精准无痕编辑
基因组编辑技术作为生命科学领域的一项革命性突破,为基础研究和应用开发提供了强大的技术支撑。精准染色体编辑技术对于基因操纵、疾病治疗和作物改良等方面至关重要[1]。新一代基因组编辑工具要求能够支持染色体水平的改造,包括插入、删除、倒位和易位,实现多基因编辑和通过调控基因组结构变异来调节植物性状和治疗遗传疾病[2]。此外,精准的染色体重排使得体内构建人工染色体成为可能,已在分子育种、合成生物学和分子治疗方面显示出巨大的潜力。
传统的大片段DNA编辑工具通常依赖于双链断裂(DSB)和非同源末端连接(NHEJ),可能会引入额外的插入或缺失,以及细胞损伤[3]。近几年出现了不依赖于DSB的大片段DNA操纵技术,包括使用成对pegRNA的先导编辑已在哺乳动物细胞中实现大片段DNA编辑[4, 5],以及将位点特异性重组酶与CRISPR系统相结合的PASSIGE、PASTE和PrimeRoot等系统,也能在哺乳动物细胞和植物中进行大片段DNA插入[6, 7]。然而,这些技术在编辑效率、尺度、精准性及类型多样性等方面仍存在明显不足。
2025年8月,Cell期刊报道了一项研究,开发了超大片段DNA精准无痕编辑技术——可编程染色体工程PCE和RePCE,能够在植物和人类细胞中实现千碱基到兆碱基级别的无痕DNA操纵。研究者在位点特异性重组酶Cre-Lox系统的基础上,通过高通量工程获得了逆向重组降低10倍的Lox位点,并应用人工智能辅助重组酶工程方法(AiCErec)获得了Cre变体,其重组效率是野生型的3.5倍。结合Re-pegRNA介导的无痕策略进一步提高了编辑精度,实现在染色体水平上进行无痕插入、删除、置换、倒位和易位。该研究显著拓宽了基因组编辑在分子育种、疾病治疗和合成生物学中的应用范围[8]。
将Cre-Lox系统与CRISPR结合时,显示出精确的大片段DNA操纵潜力[9]。然而,传统的Lox位点的逆向重组活性和Cre有限的DNA重组效率限制了它们的广泛应用。传统Cre系统利用的成对LoxP位点,具有相等的正向和逆向重组活性。尽管已经努力减少逆向重组,例如使用Lox66/Lox71、LoxKR3/Lox71和LoxTC9/Lox71对,但这些改造仍然没有完全解决可逆性问题。因此,研究者设计了一种策略来快速筛选重组位点(RS),在保持Lox位点高重组效率的同时最小化可逆性。首先,构建了两个质粒文库(P1和P2),每个文库都包含一个Lox位点。在P1-Lox位点的左臂(umLA)区域(中心序列的前半部分)引入了A、T、C和G的统一突变,称为Lox-umLA。同样,在P2质粒中另一个Lox位点的右臂(umRA)区域(中心序列的后半部分)引入了统一突变,称为Lox-umRA(图一A)。当P1和P2之间的发生正向重组,将产生一个同时含有LA和RA突变的质粒Lox-dm,以及另一个含有LoxP的质粒。当发生逆向重组时,将再回复到Lox-umLA和Lox-umRA的质粒(图一A)。其次,利用分裂的GFP荧光蛋白,分别构建相应的重组和逆向重组报告系统来评估重组位点变体(图一B)。再次,将P1和P2质粒文库、以及Cre表达质粒通过原生质体转化到植物细胞中(图一C)。最后,利用GFP筛选、Lox-dm的扩增和深度测序,检测LA和RA区域每个位置的碱基丰度,确定Cre重组酶对Lox位点的碱基偏好(图一C-E)。
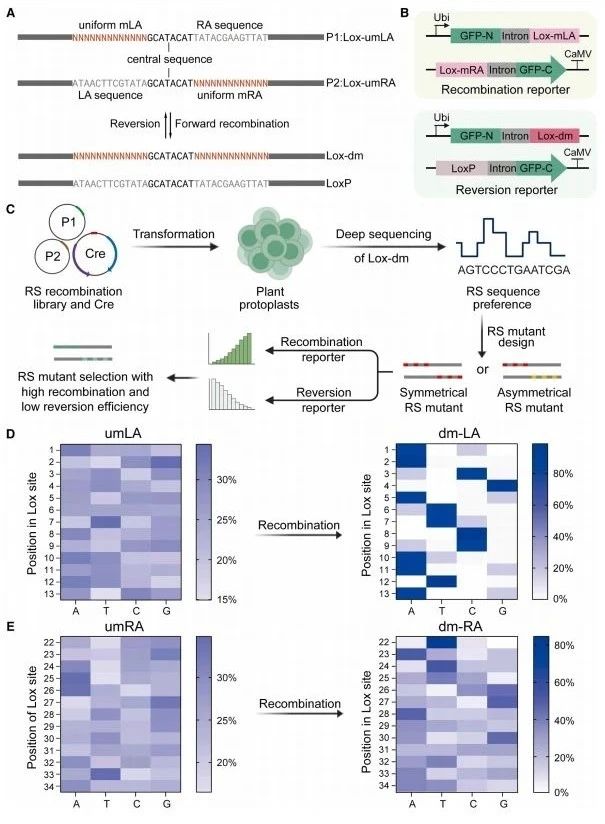
图一 开发重组位点快速改造方法及其文库构建[8]
通过转化植物原生质体和深度测序确定Cre在LA和RA区域Lox位点的碱基偏好结果(图一D和E),发现LA区域的碱基高度保守,而RA区域的碱基更灵活(图二A)。于是,研究者选择两种LA变体:Lox71和一种基于碱基偏好新生成的变体LoxPF(图二B)。然后设计对称变体,包括Scm1(LoxPF/LoxPSR)、Scm2(Sm1Lox66/Sm1Lox71)、Scm3(Sm2Ox66/Sm2Ox71)和Scm4(Sm3Lox66/Sm3Lox71),以及非对称变体包括Acm1-6(LoxPF/LoxAR1-6)、Acm7-12(Lox71/LoxAR1-16)和LoxPF/LoxPR。使用重组和逆向重组报告系统评估这些Lox变体组合的重组频率和可逆性,并通过流式细胞术测量荧光(图一C)。结果表明,基于碱基偏好的Lox变体组合显著降低了可逆性,同时保持了较高的正向重组活性(图二C和D)。最显著的变体组合是Acm8,由Lox71和LoxAR2组成,表现出与Lox71/Lox66相当的重组活性,可逆性显著降低,重组增加了10倍以上(图二D)。
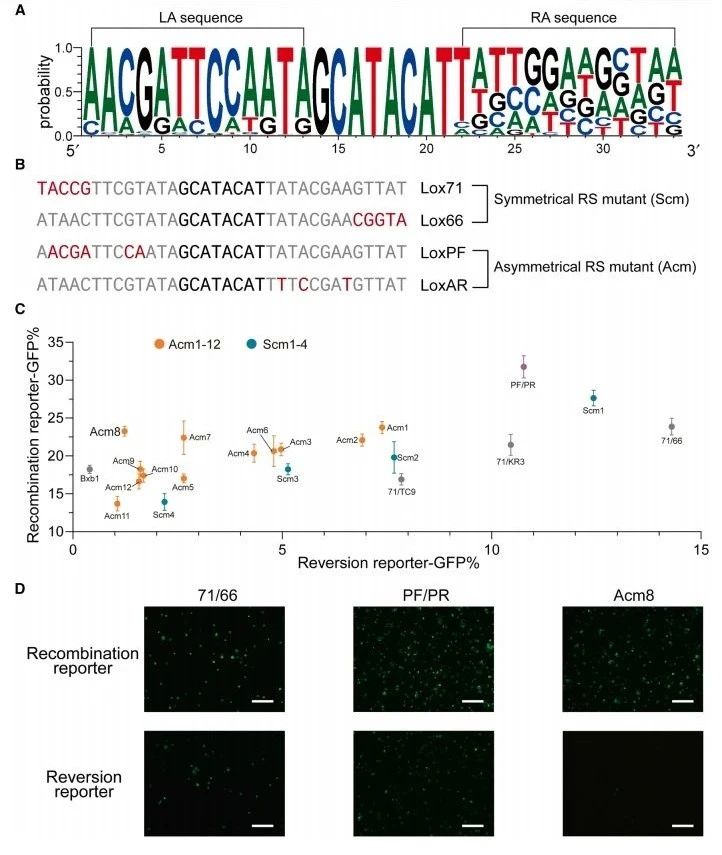
图二 设计筛选具有高重组活性且低可逆性的Lox变体[8]
Cre和其他重组酶的有限重组效率限制了它们在生物技术应用中的扩展。由于位点特异性重组酶系统固有的不可编程性,提高这些工程酶的重组能力的相关研究很少。在重组过程中,酪氨酸重组酶作为四聚体,通过Holliday连接中间体促进DNA链交换[10]。这一过程取决于重组酶的多聚化,提示重组酶的功能限制可能与多聚化有关。为了测试这种可能性,研究者进一步扩展了AiCE,一种基于进化和结构基准有效地识别具有高适应性单突变的人工智能驱动的蛋白质工程通用策略[11],开发了可用于重组酶工程的方法AiCErec。AiCErec结合了突变适应度和生物分子相互作用之间的关系(图三A),专门用于工程重组酶和其他多聚体蛋白,如转座子元件。使用AiCErec,研究者设计了24个Cre单点突变(图三B)。转化水稻原生质体的荧光分析和流式细胞术显示,8个变体的重组效率是野生型Cre的1.7-2.4倍。将效率更高突变组合成功能单元,构建了27个Cre变体,称为cm1-cm27,涉及2到5个突变的组合。测试这些变体并鉴定出10种重组活性显著提高的Cre突变组合,其中最有效的组合cm24的重组效率是野生型的3.5倍(图三C)。测试其中五种效率最高的组合,使用液滴数字PCR(ddPCR)评估GFP(720 bp)和Act1-GFP(2.4 kb)片段在六个水稻靶标上的插入效率。结果表明,变体cm24在内源性靶位点插入大片段DNA的平均效率是野生型的3.2倍(图三D)。使用AlphaFold3模型预测Cre-cm24的结构,评估其五个突变H40E、D141Q、A275P、K276P和Q281G如何影响酶的构象。在野生型结构中,残基H40、K276和Q281预计会与DNA链相互作用,而突变残基40E、276P和281G的位置离DNA更远,表明相互作用减少,这可能有助于Holliday连接解离。此外,位于K276附近的A275P可能增强酶的结构稳定性,同时保持重组活性,而D141Q突变可能减少与E138的静电排斥,影响亚基C的F142和亚基D的L339之间的相互作用(图三E)。这种改变可能促进了链交换过程中多聚体亚基的分离,有助于链交换并提高重组效率。以上结果表明,AiCErec可用于位点特异性重组酶的工程改造,获得有效的Cre变体。
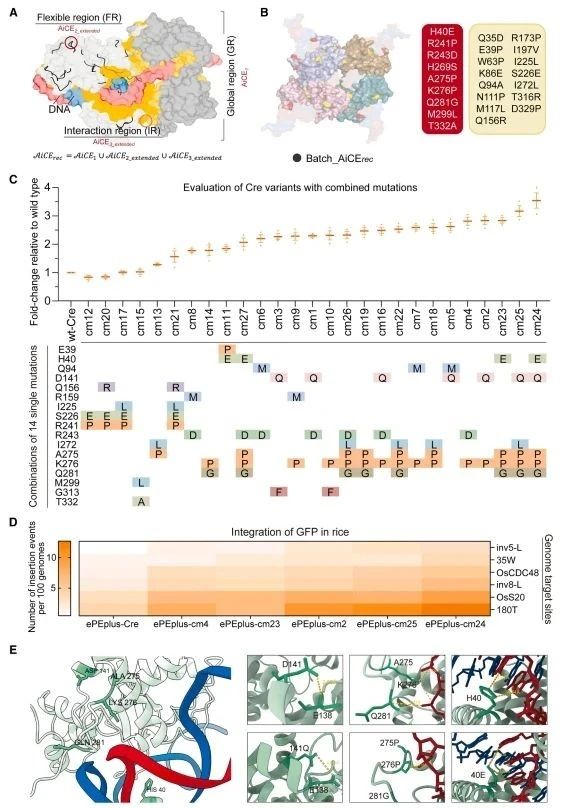
图三 人工智能辅助设计具有增强DNA重组活性的Cre变体[8]
基于上述的开发和改造,研究者结合Acm8和Cre变体cm24,开发了迭代的Cre-Lox系统,并将其与先导编辑系统集成,以赋予可编程性。通过设计重组位点的着陆位置和方向,迭代Cre-Lox系统可以在染色体尺度上生成精确的插入、缺失、置换、倒位和易位,将此系统称为PCE(图四A)。评估PCE将720 bp、2.4 kb和18.8 kb的DNA片段插入玉米和小麦原生质体的六个靶位点的频率(图四B和C),并和PrimeRoot作比较。结果表明,在玉米原生质体中,PCE的有效性是PrimeRoot的1.8-5.3倍,在小麦原生质体中插入GFP的有效性比PrimeRoot高3.7-6.2倍,插入18.8 kb片段的有效性高3.9-11.8倍。选择两个小麦基因组安全位点Ta6D和Ta7D,比较PrimeRoot和PCE精确插入GFP基因的有效性。发现PCE在两个靶位点的GFP整合频率分别为9.7%和5.4%,显著高于PrimeRoot在两个位点的0.5%(图四D)。设计pegRNA,在水稻原生质体的四个基因组区域插入LoxAR2和反向Lox71(rLox71),随后比较通过NHEJ DNA修复途径和PCE产生的靶向倒位的频率,发现PCE的频率比NHEJ高17.6倍(图四E)。还设计构建了用于植物的具有23 bp逆转录模板的Lox-pegRNA,插入Lox后测试倒位的频率,PCE对inv-35W和inv-315H的精确倒位率分别为26.2%和20.1%,显著高于PrimeRoot(图四F)。以上数据表明,PCE系统能够精确整合各种植物物种的大片段DNA,在植物中产生精确、大规模基因组倒位方面优于传统方法,为基因组工程提供了一种强大的工具。
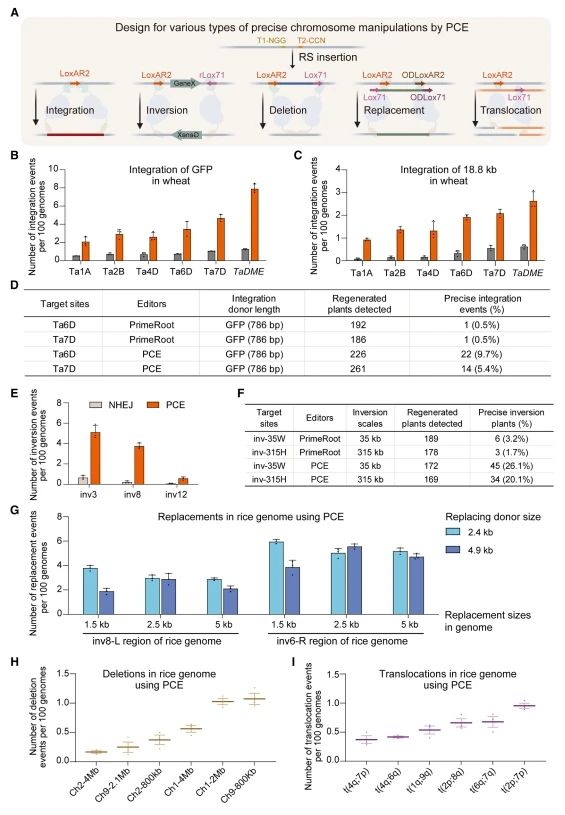
图四 植物高效染色体编辑PCE系统的开发与测试[8]
接下来,研究者测试PCE系统在千碱基水平上实现靶向DNA置换,以及在染色体水平上实现靶向缺失和易位的能力。在特定区域两侧插入两个正交的LoxAR2位点,以及一个含有所需DNA片段的外源供体,该片段两侧是Lox71位点,从而可以置换靶基因组区域(图四A)。使用PCE,以1.5、2.5和5 kb的间隔将正交LoxAR2序列插入水稻基因组的两个区域,用含有正交Lox71序列的2.4或4.9 kb供体DNA片段置换中间的基因组区域。原生质体转化后,ddPCR显示编辑效率高达5.9%(图四G)。通过插入靶区侧翼的LoxAR2和Lox71位点,水稻原生质体中9号染色体着丝粒和1号染色体短臂2 Mb片段的缺失频率达到1.0%(图四H)。将LoxAR2插入在水稻1、4和6号染色体长臂以及2号染色体短臂上的靶位点,Lox71位点插入到6、7、8和9号染色体长臂以及7号染色体短臂上的靶位点,易位效率可达1.0%(图四I)。以上数据表明,PCE能够在植物中实现从千碱基到兆碱基水平的跨尺度DNA操作,促进了DNA结构的大规模编辑。
通过双pegRNA策略精确插入重组位点需要NGG和CCN PAM,限制了靶向范围。为了克服这一限制,研究者开发了一个基于植物先导编辑器(ePPEplus)的无PAM先导编辑系统,SpRY-ePPEplus。通过二代测序测量了LoxAR2插入效率,SpRY-ePPEplus几乎对所有PAM序列都产生了最高的插入频率(图五A),突出了它在最小PAM限制下插入重组位点的实用性。位点特异性重组酶和其他转座子系统的内在特性导致编辑后特定序列的保留。为了克服这一局限性,研究者开发了一种无痕的染色体编辑策略。这种方法利用了额外的pegRNA,称为Re-pegRNA,用于在大片段DNA操作后重新启动和编辑重组位点序列(图五B)。通过结合PCE系统、SpRY-ePPEplus框架和无痕概念,开发了一种基于Re-pegRNA的无痕染色体编辑系统,称为RePCE,该系统可在PAM最小限制的情况下运行。为测试RePCE的无痕编辑能力,建立了无痕和无PAM-荧光报告(SPR)系统。还设计了Lox-pegRNA,用于在非NGG PAM靶位点插入LoxAR2序列,并设计了Re-pegRNA靶向两个残留的Lox位点,实现GFP重组后的无痕修复,通过评估荧光来评估RePCE的真正无痕编辑能力(图五C)。将RePCE和SPR系统转化水稻原生质体,仅当引入两对Re-pegRNA时才观察到荧光原生质体(图五D),证实了RePCE进行精确且无痕编辑的能力。通过在内源性靶位点使用四种不同的PAM(NGA、NAT、NTA和NGC)评估GFP插入,进一步证实RePCE在没有NGG PAM的性能(图五E)。这些结果突出了RePCE在跨不同PAM序列的大片段DNA编辑中的有效性,并证实了其无痕编辑的潜力。
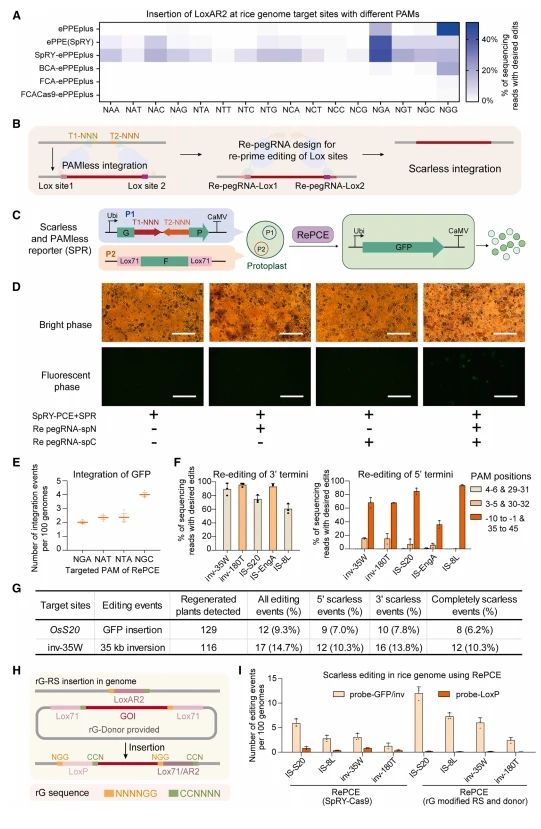
图五 无痕染色体编辑器RePCE的开发[8]
随后,评估RePCE系统在内源性基因组位点进行无痕大片段DNA操纵的能力。针对剩余Lox位点设计了Re-pegRNA来消除5'末端的LoxP和3'末端的Lox71/AR2。两端高达90%的Lox序列被Re-pegRNA重新编辑,从而实现了无痕操作(图五F)。进一步在水稻愈伤组织中证实了SpRY介导的RePCE系统的无痕编辑能力(图五G)。为了提高无痕编辑效率,研究者提出了一种“rG” 策略,即重新设计的Lox-pegRNA和具有GG基序的供体。通过将N3NGG和CCNN3基序人工引入重组位点和供体序列,编辑后位于Lox位点侧翼的NGG和CCTN-PAM可用于设计使用SpCas9重新编辑的Re-pegRNA(图五H),使得无痕编辑效率有了显著提高(图五I)。以上结果表明,RePCE是植物无痕染色体编辑的一种高效工具,显著提高了精确和无痕大片段DNA操纵的潜力。
最后,研究者评估RePCE在人类细胞中的无痕编辑能力。为测试插入效率,构建了由CMV启动子驱动的PCE系统,分别在AAVS1、ACTB、LMNB1和HEK3中选择了四个靶位点,并构建了由hU6启动子驱动的Lox-pegRNA和Re-pegRNA。转染HEK293T细胞后,检测到GFP的插入率高达18.6%,LoxP的插入率几乎为零(图六A),表明当使用Re-pegRNA时,大多数插入事件都被重新编辑以消除LoxP序列。深度测序显示,Lox序列的重新编辑效率超过90%(图六B)。为研究RePCE模拟遗传疾病相关染色体结构变化的潜力,选择了两种疾病尤因肉瘤(ES)和非小细胞肺癌(NSCLC)。前者通常由11号染色体和22号染色体之间的易位引起EWSR1和FLI1的融合,后者由12 Mb倒位引起的2号染色体上ALK和EML4的融合(图六C和D)。转染后,使用探针LoxP和探针trl/inv通过ddPCR分别检测到1.9%的染色体融合效率和1.2%的染色体倒位效率(图六E)。随后的深度测序显示,在编辑区域的5'末端和3'末端,无痕编辑效率高达90%(图六F)。为评估RePCE系统在哺乳动物细胞中的特异性,使用Cas-OFFinder,确定了18个潜在的脱靶位点。二代测序显示,在预测的脱靶pegRNA位点没有Lox插入,只有少数indels(图六G)。ddPCR结果进一步证实,在最相似的LoxAR2脱靶位点上插入的GFP极少(图六H),证实了RePCE系统在人类细胞中的高度特异性。以上数据表明,RePCE系统可用于人类细胞疾病相关位点的无痕大片段DNA插入、染色体融合和倒位。
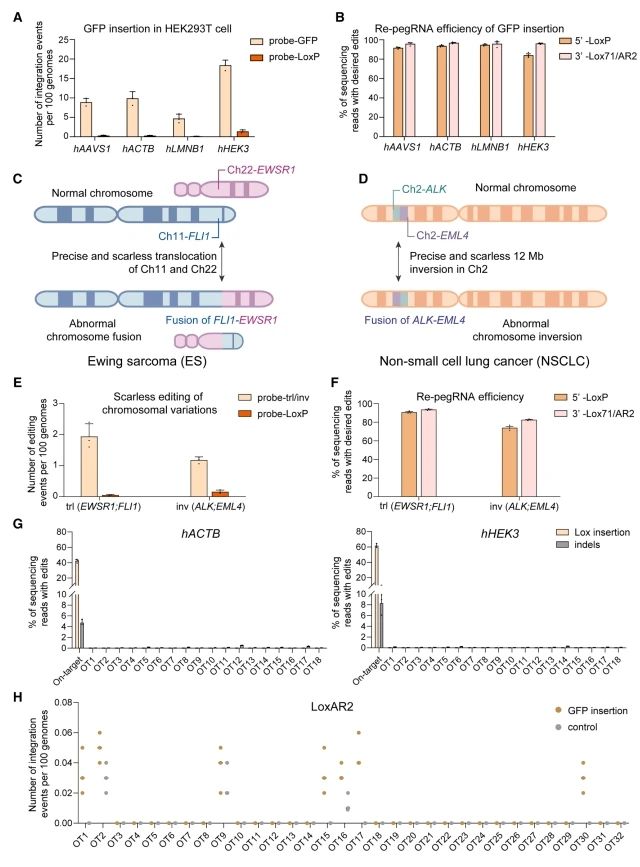
图六 RePCE在人类细胞中实现无痕染色体编辑[8]
综上所述,该研究开发了一种无痕染色体编辑工具,可以在植物和人类细胞中进行高效的大片段DNA操纵,应用范围从千碱基到兆碱基级别。在Cre-Lox系统的基础上,研究者设计出新的重组位点Lox变体降低了10倍逆向重组,并筛选出重组效率提升3.5倍的Cre变体。进而,开发了一种可编程染色体工程(PCE)系统,先使用先导编辑器和pegRNA在靶位点插入重组位点Lox变体,然后用Cre变体进行DNA重组。PCE系统应用于细胞和植物时,实现了高达18.8 kb的精确DNA插入、4 Mb的缺失、5 kb的置换、12 Mb的倒位和染色体易位,效率达到26.2%。在此基础上,研究者还设计了一种无痕染色体编辑策略RePCE,使用Re-pegRNA在大片段DNA操作后重新启动和消除Lox,实现无缝基因编辑,进一步提高了编辑精度。PCE和RePCE为不同物种大片段DNA的高效和无痕操纵提供了强大的工具,提升了真核生物基因组的操纵尺度和能力,拓宽了基因组编辑在分子育种、疾病治疗和合成生物学中的应用范围。

GTP研发中心拥有自主知识产权的类精子干细胞介导的半克隆技术,可在体外实现类精子干细胞的多位点改造并进行功能元件测试,最后将类精子干细胞通过卵母细胞注射可以一步法获得基因改造小鼠。基于类精子干细胞单倍体打靶高效率的优势,可快速构建定制化小鼠,如基因组安全位点大片段敲入、条件性基因敲除/敲入和人源化基因改造等。多个复杂调控元件可以通过多次体外细胞打靶构建在同一株类精子干细胞中,经细胞水平质控后,一步法获得基因改造小鼠。

如有需要欢迎联系我们,GTP研发中心将竭诚为您设计最佳方案,制定专属于您的基因改造小鼠。
网址:http://www.sibcb.ac.cn/gtp/
地址:上海市岳阳路320号
责任编辑:多一百
新媒体运营:suway
参考文献
1. Li B, Sun C, Li J, Gao C: Targeted genome-modification tools and their advanced applications in crop breeding. Nat Rev Genet 2024, 25(9):603-622.
2. Park CY, Sung JJ, Kim DW: Genome Editing of Structural Variations: Modeling and Gene Correction. Trends in biotechnology 2016, 34(7):548-561.
3. Leibowitz ML, Papathanasiou S, Doerfler PA, Blaine LJ, Sun L, Yao Y, Zhang CZ, Weiss MJ, Pellman D: Chromothripsis as an on-target consequence of CRISPR-Cas9 genome editing. Nature genetics 2021, 53(6):895-905.
4. Choi J, Chen W, Suiter CC, Lee C, Chardon FM, Yang W, Leith A, Daza RM, Martin B, Shendure J: Precise genomic deletions using paired prime editing. Nature biotechnology 2022, 40(2):218-226.
5. Jiang T, Zhang XO, Weng Z, Xue W: Deletion and replacement of long genomic sequences using prime editing. Nature biotechnology 2022, 40(2):227-234.
6. Wang C, Qu Y, Cheng JKW, Hughes NW, Zhang Q, Wang M, Cong L: dCas9-based gene editing for cleavage-free genomic knock-in of long sequences. Nat Cell Biol 2022, 24(2):268-278.
7. Anzalone AV, Gao XD, Podracky CJ, Nelson AT, Koblan LW, Raguram A, Levy JM, Mercer JAM, Liu DR: Programmable deletion, replacement, integration and inversion of large DNA sequences with twin prime editing. Nature biotechnology 2022, 40(5):731-740.
8. Sun C, Li H, Liu Y, Li Y, Gao R, Shi X, Fei H, Liu J, Liang R, Gao C: Iterative recombinase technologies for efficient and precise genome engineering across kilobase to megabase scales. Cell 2025.
9. Sun C, Lei Y, Li B, Gao Q, Li Y, Cao W, Yang C, Li H, Wang Z, Li Y et al: Precise integration of large DNA sequences in plant genomes using PrimeRoot editors. Nature biotechnology 2024, 42(2):316-327.
10. Van Duyne GD: A structural view of cre-loxp site-specific recombination. Annual review of biophysics and biomolecular structure 2001, 30:87-104.
11. Fei H, Li Y, Liu Y, Wei J, Chen A, Gao C: Advancing protein evolution with inverse folding models integrating structural and evolutionary constraints. Cell 2025.



