技术分享:转录因子直接竞争错配修复驱动DNA突变
尽管真核DNA复制的保真度极高,但在每个复制周期中仍会发生核苷酸的错误掺入,从而产生突变,驱动遗传疾病的发生和基因组进化。人类基因组的突变率差异很大,范围从横跨百万碱基如晚期复制区的高突变率,到单个核苷酸如CpG背景下的C>T高突变率[1]。介于这两种极端之间,转录因子(TF)的蛋白结合调控位点在体细胞中的突变也惊人地丰富。这种高频突变模式涉及多种因素和癌症类型。黑色素瘤细胞的基因组数据分析显示,TF结合位点(TFBS)的突变率增加和DNA修复减少具有相关性[2],表明TF可能通过直接竞争修复来增加突变,但这仅适用于紫外线诱导引起的大量损伤,其修复依赖于核苷酸切除修复途径。相比之下,大约三分之二的癌症突变来源于复制错误,即没能通过错配修复(MMR)途径去除而留下的错配突变。因此,尚不清楚体细胞突变在多种癌症类型的TFBS中为何如此普遍。有研究证明,一系列结构域家族的TF蛋白表现出与DNA错配损伤的高亲和力结合[3],提示TF的直接结合可能干扰了错配修复的识别与进行,导致体细胞突变的增加。在真核生物中,MutSα(MSH2和MSH6蛋白的异二聚体复合物)是识别DNA复制产生的错配并启动MMR修复的主要功能酶。因此,充分探究TF结合、MutSα识别与修复、DNA突变产生三者之间的关联,将使人们更广泛地理解遗传变异和基因组演化的突变机制。
2025年7月,Cell期刊在线报道了一项研究,发现转录因子(TF)与真核错配修复(MMR)的主要酶MutSα直接竞争,快速识别并结合DNA错配,从而干扰MMR修复,导致DNA突变产生(图一)。使用酵母遗传分析量化TF结合位点的突变积累,TF与MutSα竞争导致细胞中TF结合位点的突变增加,证明了这种TF诱导的突变机制。这一发现也存在于人类癌症细胞,由MYC结合错配引起的突变在MMR功能正常的细胞中增多,TF与MMR系统竞争是癌症中TF结合位点体细胞高频突变的关键决定因素。这种竞争在不同家族的MutSα同源物和TF中普遍存在。该研究揭示了一种新的TF诱导的突变机制,对研究TF结合位点罕见遗传变异疾病和调控基因组DNA演化具有重要意义[4]。
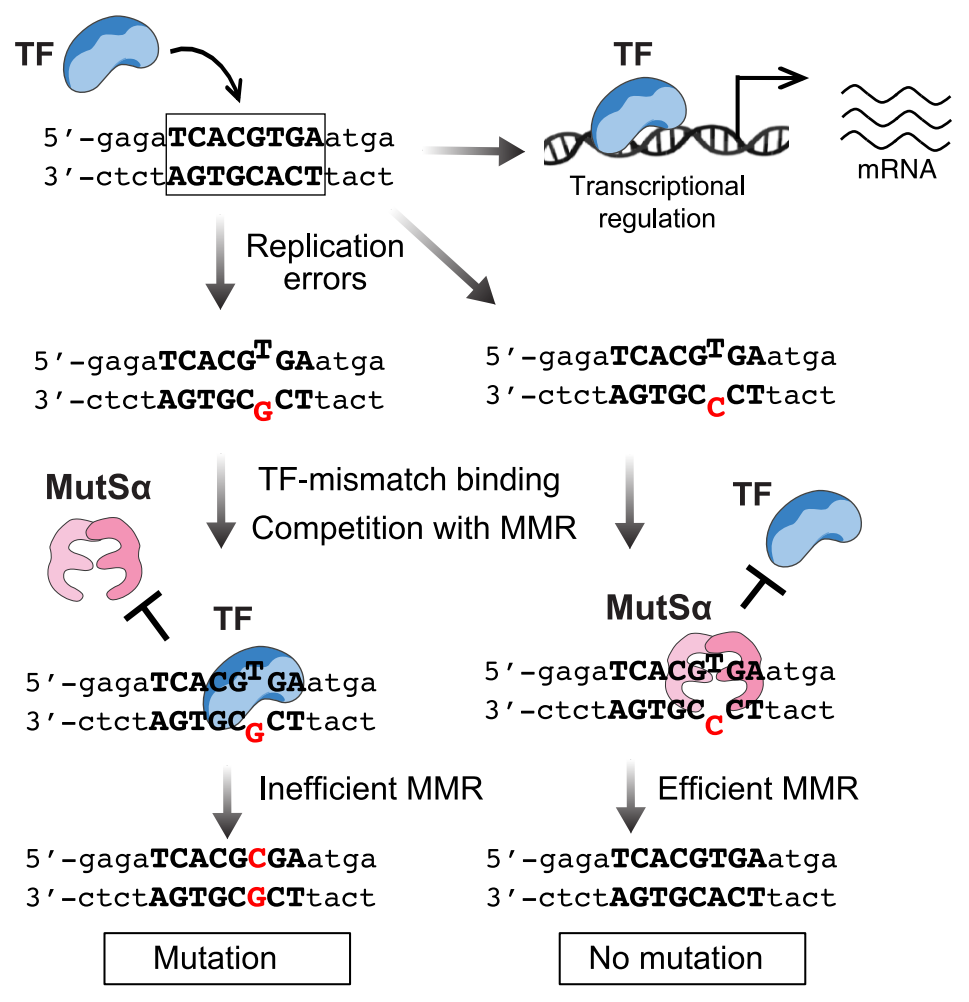
图一 转录因子与错配修复竞争诱导DNA突变[4]
之前研究证明,TF蛋白表现出与DNA错配损伤的高亲和力结合[3]。然而,由于目前没有可用的MutSα-TF竞争数据,因此尚不清楚这种结合是否足够强到竞争MutSα识别DNA错配。为此,研究者使用之前开发的饱和错配结合分析(SaMBA)[3]技术来定量测量MutSα单独或与TF竞争时结合数千个含有错配DNA序列的情况。选择酿酒酵母TF Cbf1作为研究对象,基于两个14 bp的序列设计错配DNA文库:一个包含高亲和力位点TCACGTGA的Cbf1靶序列[5],和一个具有相同核苷酸组成但预计不会被Cbf1结合的杂乱位点(SS,图二A)。对于这两个序列,计算生成所选位点及其直接侧翼的所有可能错配。然后在高密度DNA芯片上从头合成DNA分子,作为单链探针,自杂交形成互补位点和所有可能的单错配变体(图二A和B)。将双链芯片与重组全长MutSα、Cbf1或MutSα和Cbf1以1:2的比例孵育,以匹配它们在酵母细胞中的相对丰度。根据芯片上每个DNA点的蛋白质荧光(MutSα)或抗体荧光(Cbf1)定量每种蛋白质的结合。正如预期,与基因组靶标类似,Cbf1与所选结合位点(TFBS)结合强烈,而与杂乱位点(SS)的结合在随机位点范围内(图二C)。杂乱位点的错配与Cbf1结合较差,而结合位点的错配显示出广泛的结合亲和力,其中大多数在基因组靶标范围内(图二D)。对MutSα的高通量结合数据表明,C-C错配的识别率很低,T-G错配具有很高的亲和力结合(图二E)。此外,发现大多数错配表现出广泛的MutSα结合亲和力,具体取决于序列背景。数据显示了显著的可重复性(图二F)、与独立结合亲和力数据[6]的高度相关性(图二G)。
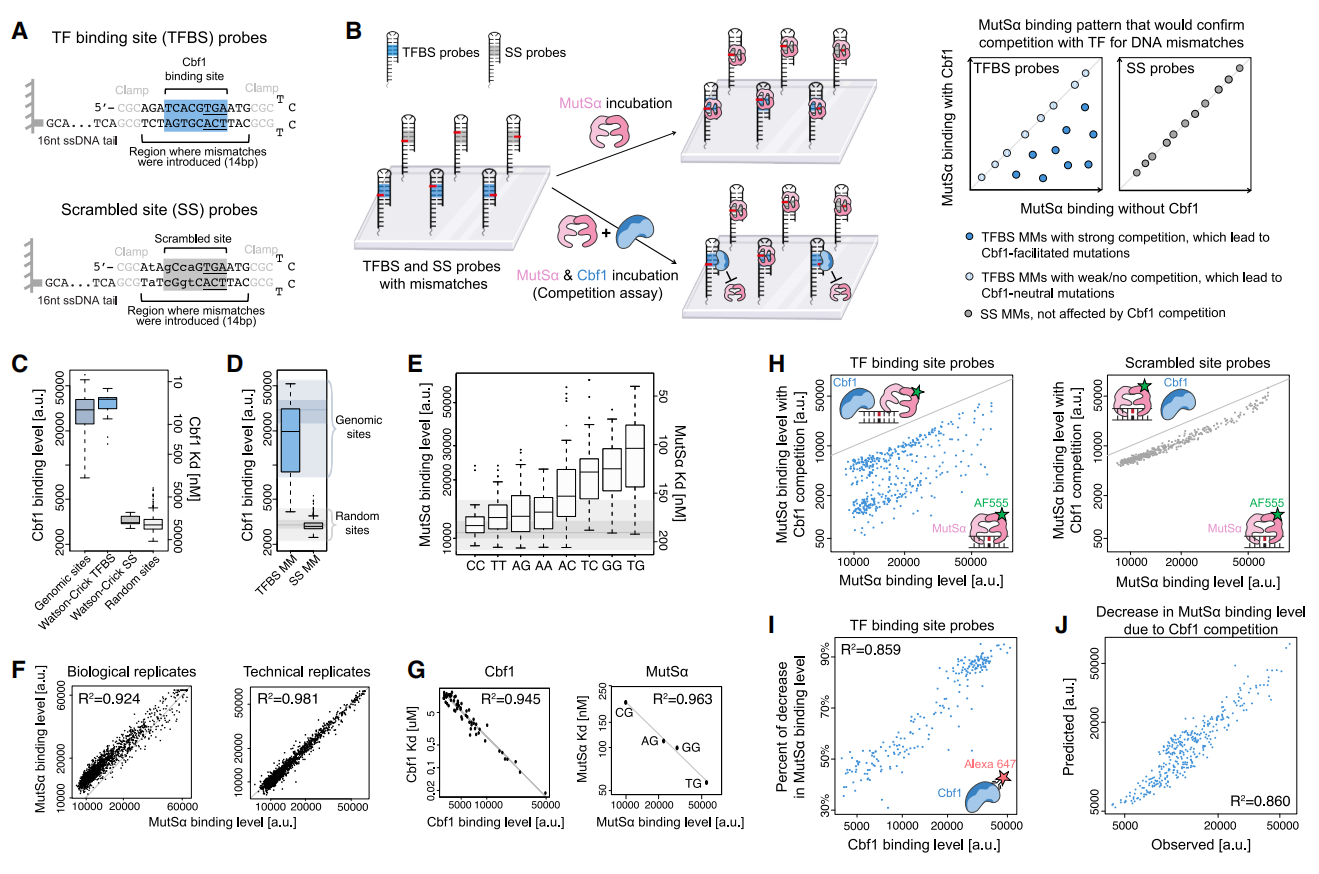
图二 转录因子Cbf1与MutSα竞争结合DNA 错配[4]
比较Cbf1存在或不存在时的MutSα-DNA结合水平,结果显示出一种强烈的竞争性结合模式(图二H)。在杂乱位点引入的错配中,仅观察到Cbf1对MutSα结合的适度和非特异性影响(图二H,右),这与TF对这些探针的低亲和力相一致(图二D)。相比之下,在Cbf1结合位点引入的错配中,观察到在Cbf1存在的情况下MutSα结合显著减少,竞争效应的大小(与对角线的距离)在错配探针之间差异很大(图二H,左),并与Cbf1对错配序列的结合亲和力相关(R2=0.859;图二I)。此外,使用从SaMBA测量中得出的Cbf1和MutSα结合亲和力数据(图二G),发现Cbf1和MutSα之间直接竞争的简单生物物理模型:Cbf1导致MutSα识别错配的减少(图二J)。以上结果表明,Cbf1结合对MutSα识别错配产生了广泛影响,MutSα不能有效识别TF结合的DNA错配。
为了测量Cbf1错配结合对活细胞突变形成的影响,研究者采用LYS2逆转试验来测量酵母中的突变[7]。LYS2是酿酒酵母的一个非必需基因,它包含一个约150 bp的区域,被称为逆转窗口,该区域在功能上是不必要的,常被用于研究插入的异源DNA序列的诱变特性。研究者在LYS2逆转窗口插入了一个59 bp的DNA片段,该片段包含Cbf1结合位点或者杂乱位点,插入位点使TGA三联体充当过早的终止密码子(图三A)。分别将这些菌株称为“TFBS菌株”和“SS菌株”。为了防止插入位点形成核小体,从而促进Cbf1的直接结合,设计了DNA片段使结合位点和杂乱位点两侧都有刚性Poly (dA:dT),在基因组中产生无核小体区域[8]。使用染色质免疫沉淀和定量实时PCR(ChIP-qPCR)进行验证,证实Cbf1能有效结合插入LYS2逆转窗口的TFBS位点,但不能结合杂乱位点。尽管LYS2基因在全培养基中不是必需的,但对于赖氨酸缺乏培养基中的存活至关重要。于是,在细胞积累72小时的自发突变后,将细胞转移到赖氨酸缺乏的培养基中,以选择LYS2+细胞。这些细胞成功将逆转窗口中的TGA转变为有义密码子,从而获得功能性LYS2蛋白。这种自发突变可能通过16种可能性错配引入,但不同错配对Cbf1具有不同的结合亲和力、不同水平的MutSα-Cbf1竞争,从而对MMR产生不同的影响。假设在Cbf1结合位点的TGA三联体第一个位置发生T-G错配,Cbf1以高亲和力结合并显著降低MutSα结合,导致细胞的错配识别和修复效率低下,那么预计相应突变(TGA>CGA)的发生率会增加,这是一种Cbf1促进突变(图三A)。相比之下,Cbf1对同一位置的T-C错配结合较差,预计Cbf1不会干扰这种错配的修复,导致相应突变的发生率较低(TGA>GGA),这是一种Cbf1中性突变(图三A)。如果Cbf1与错配的结合影响了MutSα结合和MMR进程,那么预计Cbf1促进突变的发生率将明显高于Cbf1中性突变(图三A)。为排除影响细胞局部突变率的其他影响,使用SS菌株作为对照(图三A右侧)。
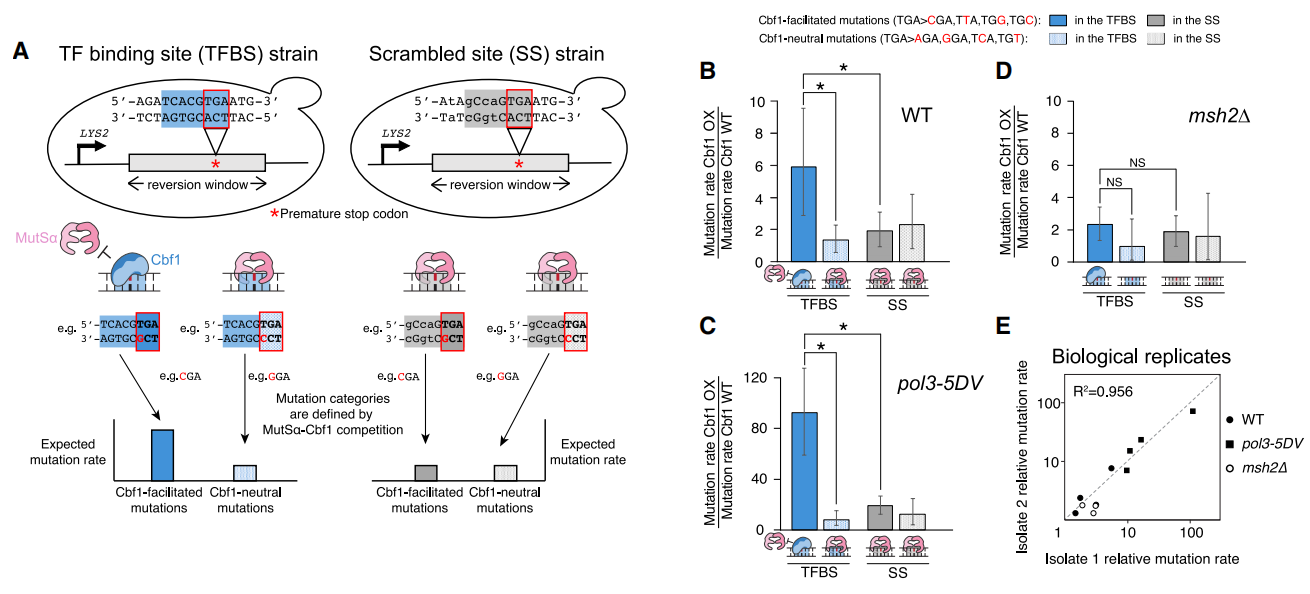
图三 TF结合DNA错配导致活细胞突变增加[4]
研究者测量了TFBS菌株和SS菌株的LYS2+逆转率,其中Cbf1表达量要么以内源性水平存在(WT),要么是过表达(OX)。通过对每种菌株至少40个回复突变进行测序,确定了TGA过早终止密码子的突变。比较了WT和过表达Cbf1的菌株突变情况,将突变类型分为4种Cbf1促进突变和4种Cbf1中性突变。在过表达Cbf1后,Cbf1促进突变增加了约6倍,而同一TFBS菌株Cbf1中性突变的增加为1.3倍(图三B,蓝色)。在杂乱位点上,两组之间没有观察到显著差异(图三B,灰色)。此外,在结合位点的Cbf1促进突变率明显高于杂乱位点的促进突变率(图三B)。为进一步证实Cbf1促进突变率的增加是由于Cbf1结合DNA复制过程产生错配所造成的,先将pol3-5DV等位基因引入TFBS和SS酵母菌株中,以禁用复制性DNA聚合酶Polδ的校对能力,已知这会增加Polδ错误掺入率并导致强烈的突变表型。正如预期,TFBS和SS菌株的总体突变率都有所增加,其中TFBS菌株中Cbf1促进突变率增加了92倍,而SS菌株的Cbf1促进突变率和中性突变率之间没有显著差异(图三C),表明pol3-5DV菌株也表现出由于TF强烈结合错配引起的突变大幅增多。此外,通过缺失MSH2来消除MMR系统,MSH2是编码MutSα酶两种成分之一的基因。在MMR缺陷(msh2Δ)细胞中,Cbf1促进突变和中性突变发生率之间,以及TFBS和SS菌株之间都没有观察到显著差异(图三D),表明这一过程确实需要MMR系统来增强TF结合错配引起的突变。
为了确定上述酵母中的TF诱导突变是否延展到人类细胞,研究者进一步分析了MYC结合位点的癌症体细胞突变数据。MYC是一种强效癌蛋白TF,在人类癌症中表现出异常过度表达和广泛的基因组占有率[9]。使用SaMBA技术进行MYC-MutSα竞争实验,基于一个MYC结合位点和具有相同核苷酸组成的相应杂乱位点,计算所有可能的错配变体,并在高密度芯片上合成了错配DNA文库,最后与MutSα单独或与MYC竞争性孵育,孵育浓度模拟人类细胞中的相对表达水平(图四A)。结果显示,在MYC结合位点的DNA错配上,MYC与MutSα存在有效竞争,但在杂乱位点上则没有(图四B),TFBS上的MYC促进突变显著增加,且MYC存在时MutSα结合的减少与MYC结合能力相关(R2=0.89,图四C和D)。考虑出现在人类基因组非编码区的所有假定MYC结合位点,不仅包括规范的E-box位点CACGTG,还包括E-box样变体CACATG、CACGCG和CACGAG。使用癌症体细胞突变数据集[10],来自10例具有微卫星不稳定性(MSI)和90例具有微卫星稳定性(MSS)表型的肿瘤。汇编了非编码人类基因组中这些假定MYC结合位点的所有突变。已包含足够多的MSI(一种由MMR缺陷引起的高变表型)样本,可以分析MMR缺陷细胞中MYC位点的突变率。结果显示,与对照位点相比,MYC结合位点的促进突变率显著增加,这种增加趋势仅存在于MSS样本中,而不存在于MMR缺陷的MSI样本中(图四E)。对于MYC中性突变,都没有增加趋势(图四F和G)。以上结果表明,MYC与MutSα在识别特定复制错误方面的竞争,确实会导致MYC结合位点的突变增加。
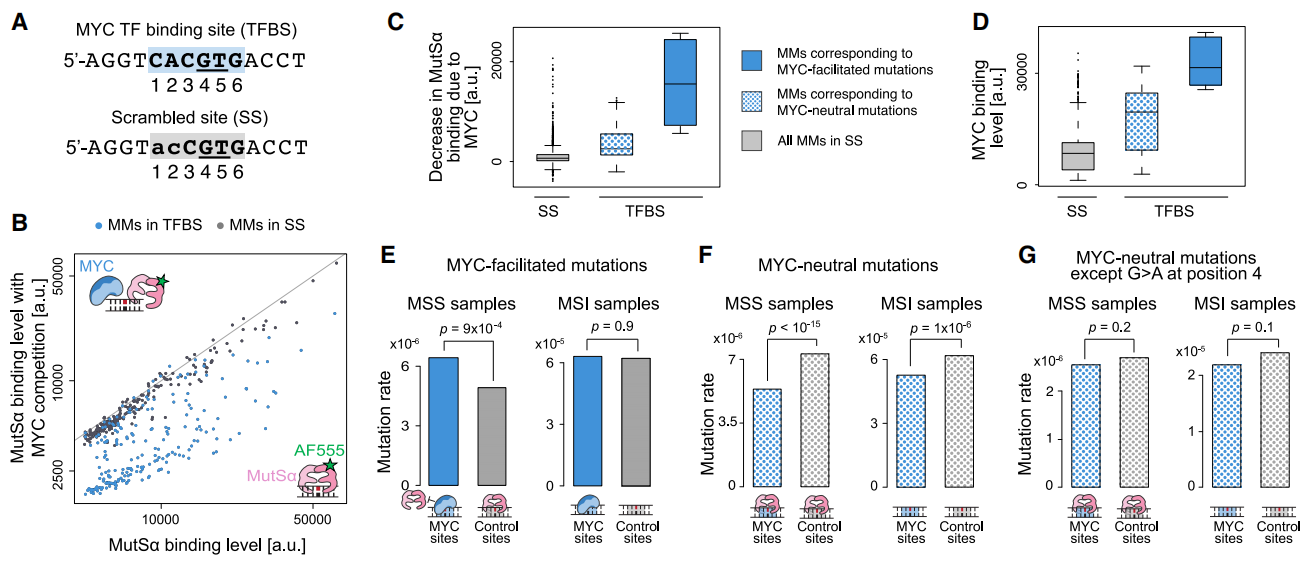
图四 MYC-MutSα竞争与MYC结合位点的较高突变率相关[4]
上述测试的酵母Cbf1和人MYC蛋白都是真核TF bHLH家族成员。研究者还对另外三种人源MAX、CREB1和EGR1进行了TF-MutSα竞争实验,发现它们都对MutSα错配识别产生了强烈的影响。TF与MutSα竞争的普遍性也延伸到MutSα同源物,如重组人MutSα。以上结果表明,这种TF与MutSα竞争可能在不同结构域家族TF之间以及不同生物体的MutSα蛋白之间广泛存在。
综上所述,研究者利用体外SaMBA技术发现转录因子(TF)与DNA错配的结合具有高亲和力,可以有效竞争真核错配修复(MMR)主要酶MutSα对错配的识别。利用开发的酵母诱变系统监测活细胞中TF相关突变的积累,发现这种直接竞争干扰了MMR进程,增加了TF结合位点上复制错误引起的突变。这一现象也存在于人类癌症细胞,表现为由MYC结合错配引起的突变在MMR正常功能细胞中的增多,表明TF与MMR竞争是癌症中TF结合位点出现体细胞高频突变的关键决定因素。该研究为TF结合位点出现罕见遗传变异提供了一种全新视角的分子机制,对研究疾病发生和基因组演化的DNA突变调控具有重要意义。

GTP研发中心拥有自主知识产权的类精子干细胞介导的半克隆技术,可在体外实现类精子干细胞的多位点改造并进行功能元件测试,最后将类精子干细胞通过卵母细胞注射可以一步法获得基因改造小鼠。基于类精子干细胞单倍体打靶高效率的优势,可快速构建定制化小鼠,如基因组安全位点大片段敲入、条件性基因敲除/敲入和人源化基因改造等。多个复杂调控元件可以通过多次体外细胞打靶构建在同一株类精子干细胞中,经细胞水平质控后,一步法获得基因改造小鼠。

如有需要欢迎联系我们,GTP研发中心将竭诚为您设计最佳方案,制定专属于您的基因改造小鼠。
网址:http://www.sibcb.ac.cn/gtp/
地址:上海市岳阳路320号
责任编辑:多一百
新媒体运营:suway
参考文献
1. Supek F, Lehner B: Scales and mechanisms of somatic mutation rate variation across the human genome. DNA repair 2019, 81:102647.
2. Sabarinathan R, Mularoni L, Deu-Pons J, Gonzalez-Perez A, López-Bigas N: Nucleotide excision repair is impaired by binding of transcription factors to DNA. Nature 2016, 532(7598):264-267.
3. Afek A, Shi H, Rangadurai A, Sahay H, Senitzki A, Xhani S, Fang M, Salinas R, Mielko Z, Pufall MA et al: DNA mismatches reveal conformational penalties in protein-DNA recognition. Nature 2020, 587(7833):291-296.
4. Zhu W, Zhang Y, Sahay H, Wasserman H, Afek A, Williams J, Shaltz S, Johnson C, Pinheiro K, MacAlpine DM et al: DNA mutagenesis driven by transcription factor competition with mismatch repair. Cell 2025.
5. Gordân R, Shen N, Dror I, Zhou T, Horton J, Rohs R, Bulyk ML: Genomic regions flanking E-box binding sites influence DNA binding specificity of bHLH transcription factors through DNA shape. Cell Rep 2013, 3(4):1093-1104.
6. Srivatsan A, Bowen N, Kolodner RD: Mispair-specific recruitment of the Mlh1-Pms1 complex identifies repair substrates of the Saccharomyces cerevisiae Msh2-Msh3 complex. The Journal of biological chemistry 2014, 289(13):9352-9364.
7. Kim N, Jinks-Robertson S: Guanine repeat-containing sequences confer transcription-dependent instability in an orientation-specific manner in yeast. DNA repair 2011, 10(9):953-960.
8. Suter B, Schnappauf G, Thoma F: Poly(dA.dT) sequences exist as rigid DNA structures in nucleosome-free yeast promoters in vivo. Nucleic acids research 2000, 28(21):4083-4089.
9. Lin CY, Lovén J, Rahl PB, Paranal RM, Burge CB, Bradner JE, Lee TI, Young RA: Transcriptional amplification in tumor cells with elevated c-Myc. Cell 2012, 151(1):56-67.
10. Wang K, Yuen ST, Xu J, Lee SP, Yan HH, Shi ST, Siu HC, Deng S, Chu KM, Law S et al: Whole-genome sequencing and comprehensive molecular profiling identify new driv000 00er mutations in gastric cancer. Nature genetics 2014, 46(6):573-582.



