技术分享:转录因子的结合与靶基因调控呈现低重叠性
真核细胞表达的DNA序列特异性转录因子(TF)可作用于靶基因的增强子和启动子的调控位点,从而调节转录和染色质结构。TF通过与辅因子或基础转录机器的直接相互作用来调节转录的起始、延伸或者染色质修饰[1]。经大规模筛选表明,在没有其他TF的情况下,只有一小部分TF具有很强的激活功能,这意味着大多数TF是通过联合作用来调节基因转录[2, 3]。为了更好地理解单个TF在全基因组范围内的作用,需要明确TF的功能和重要性在不同基因上的变化,如何界定相同调控元件上不同TF的功能、区分靶基因的启动子类型和单个基因的染色体背景等。尽管有研究表明,单个TF具有强烈的激活或抑制功能,可以调控邻近基因,但早期尝试将TF结合与“靶基因”调节相关联的研究产生了令人困惑的结果。例如,通过综合分析TF基因缺失或小干扰RNA耗竭,发现部分TF结合位点并无调控功能,或者TF在不同基因上具有相反的调控作用[4, 5]。然而,这些研究大多依赖于稳态RNA的分析,并且是在长期缺失TF的情况下进行的,很难区分直接效应或者间接效应。
酿酒酵母是研究TF特异性、协同性和功能的优秀模型。尽管酵母缺乏已知的长程增强子,但其许多转录机制与高等真核生物相保守。酵母仅有约150个TF,总量不到人类的十分之一,使其更适合进行大规模全基因组的TF结合与功能比较。现有大量的TF-TF相互作用、TF调控“靶基因”、以及TF共调节靶基因集的遗传学生化数据[6, 7] 。将酵母蛋白的编码基因根据辅因子依赖性进行分类,大多数酵母基因的表达依赖于TFIID和Bdf1/2辅因子,这类基因多为组成型表达;而一部分受调控的基因转录呈现辅因子冗余特性,称为辅因子冗余型基因(CR),可依赖于TFIID或SAGA。在酵母中,大多数TF被认为是通过结合上游激活序列来调节转录的,这些激活序列位于受调节基因上游的几百个碱基对以内[8]。然而,多个TF如何协同调节单个基因,TF如何促进辅因子的特异性,以及某些TF是否对基因类型具有特异性,这些都是重要且未决的问题。
2025年6月,Nature期刊报道了一项研究,通过绘制酵母近乎所有转录因子(TF)的结合位点及其靶基因调控图谱,对TF的作用及其调控全基因组的转录机制提出了独特的见解。与预期相反,仅有极少数TF专一地承担基因激活或抑制功能,绝大多数TF具有双重功能。尽管几乎所有的蛋白质编码基因都受到一个或多个TF的调控,但TF结合和基因调控之间的重叠性有限。TF的快速耗竭揭示了存在许多远端TF结合位点的调控靶点,提示存在其他远端的非经典调控机制[9]。
首先,研究者绘制了酵母几乎所有序列特异性转录因子(TF)的结合位点图谱。这些TF包括带注释的激活因子、阻遏因子和推定的调节因子(图一a)。为了进行比较,还绘制了一些染色质重塑因子、转录辅因子和转录起始前复合物相关蛋白的结合位点图谱。将每种因子单独融合微球菌核酸酶(MNase),使用染色质内源性切割测序(ChEC-seq)探测其在基因组DNA上的结合位置,根据每个TF-MNase融合引导的DNA切割绘制其染色体位置。由于酵母不含长程增强子,研究者在转录起始位点(TSS)-400至 +200 bp的区间内分析TF与蛋白质编码基因DNA的结合。该区间包含77%的TF结合位点(图一b)。在检测的5891个启动子中,有5467个检测到至少与一个TF相互作用。每个启动子与1-137个TF相互作用,中位数为15个TF。而单个TF结合的启动子数量范围很广,从1-3355个不等,中位数为624个启动子(图一c)。
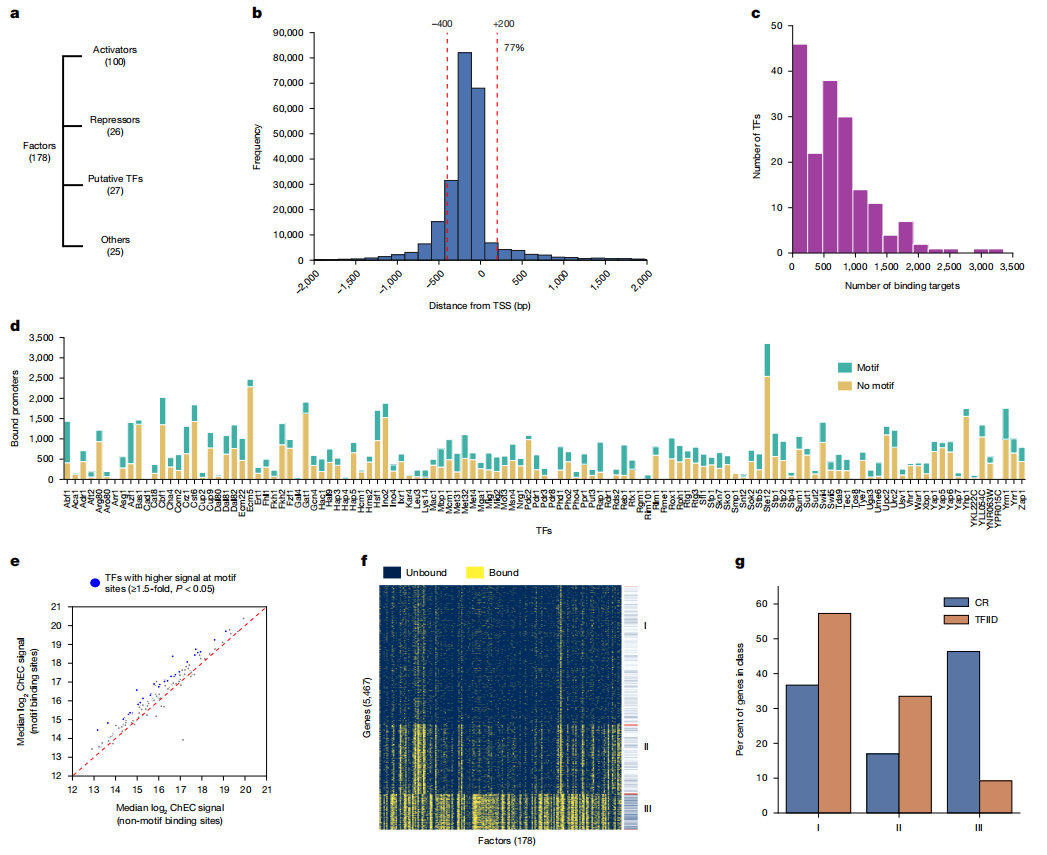
图一 全基因组范围内的酵母转录因子结合图谱[9]
评估TF结合位点的DNA序列特异性,47个TF(例如Abf1和Reb1)在其50%或更多的结合位点上存在匹配的基序;而对于大多数TF,只有一小部分结合位点存在基序,范围7-49%,中位数为34%(图一d)。令人惊讶的是,对于大多数具有已知DNA结合基序的TF(120个中有87个),ChEC信号在含基序启动子和无基序启动子之间没有显著统计学差异(图一e),表明对于大多数TF来说,序列基序不能可靠地预测体内TF-启动子的结合。使用二元TF结合数据的无监督k-means聚类将基因分为三个不同的类别。簇I包括所分析总基因的53%,其启动子结合相对较少的TF,中位数为每个启动子结合7个TF。簇II约占26%的基因,结合了中等数量的TF,每个启动子的结合TF中位数为24。而簇III约占14%的基因,与许多TF相结合,每个启动子的结合TF中位数为67(图一f)。簇I和簇II富含TFIID依赖性基因,而簇III富含CR基因,具有最宽的核小体耗竭区(NDR),包含许多高表达基因,并且高度富集了位于染色体相互作用域边界附近的基因(图一f和g)。
接下来,研究者系统评估TF的全基因组功能,以确定DNA结合与发挥功能之间的关联程度。使用生长素诱导的degron(AID)系统[10]检测了TF快速耗竭后新生RNA水平的变化。将AID与几乎所有序列特异性TF融合,获得了126株酵母菌株。菌株用吲哚-3-乙酸(IAA)处理后,用4-硫代尿嘧啶(4TU)处理以标记新转录的RNA,然后进行新生RNA富集、RNA测序和蛋白质印迹分析(图二a)。在Gcn4、Cbf1、Cyc8或Reb1耗竭后的三到四个时间点收集RNA(图二b)。在添加IAA 30分钟后,这4个克隆菌株中分别有43、603、2973和1349个基因的转录发生了改变。其中Cyc8的缺失改变了近一半蛋白质编码基因的转录。根据每个基因的调节性TF数量计算z评分,确定了211个基因具有异常数量的调节性TF(图二c和d)。其余4725个基因由至少一个TF调节,每个基因有1-21个调节性TF,包括85%的TFIID依赖性基因和75%的CR基因。与TFIID依赖性基因(3个TF)相比,影响CR基因(7个TF)的TF中位数更多;同样,与其他两个簇(3个TF)相比,簇III基因受更多TF(6个TF)的调控。TF的快速耗竭会导致许多基因的上调和下调,这种现象适用于大多数TF。图二e显示了TF耗竭后下调的受调控基因的百分比。单一的激活性TF(例如Msn2和Gcn4)或抑制性TF(例如Rox1和Sum1)相对较少,大多数酵母TF(超过66%)具有双重作用(图二f)。
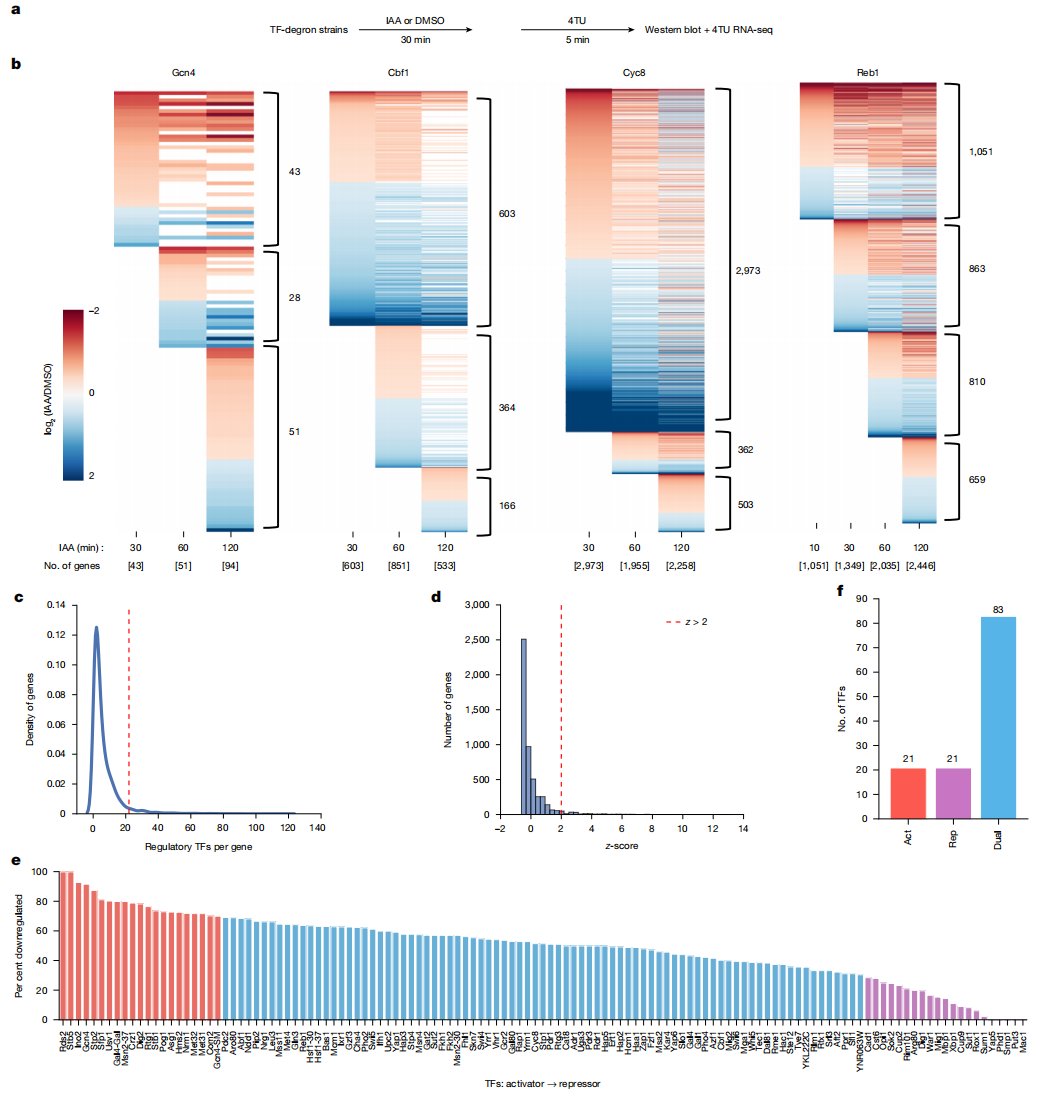
图二 TF快速耗竭以鉴定调控靶基因[9]
随后,研究者整合TF的DNA结合数据及其调控靶基因的表达数据,发现只有少数TF例如Ifh1、Cyc8、Rap1、Abf1和Reb1有中度重叠(>40%)。相比之下,大多数TF的重叠要小得多,即明显的无功能结合比例很高(图三a)。对于许多TF,大多数受调控的靶基因并没有检测到TF在启动子的结合位点(图三b)。例如,Cyc8结合了777个启动子,但30分钟的快速耗竭影响了2765个基因的表达,其中仅有422个基因检测到Cyc8的结合。同样,Reb1结合851个启动子,但30分钟的快速耗竭了改变1244个基因的表达,其中仅有353个基因检测到Reb1的结合。总体而言,约6%的TF结合靶点检测到功能,以及约23%的表达调控靶点检测到TF的结合(图三a和b)。由于许多基因的表达是可诱导的,例如Gal4调节的基因,因此一些TF结合位点可能仅在特定的生长条件下具有活性,这可能解释了大量非功能位点的原因。为了探索这种可能性,研究者在压力条件下耗尽TF,并比较了在诱导条件下的TF结合以及靶基因的表达变化。在甲嘧磺隆诱导氨基酸饥饿60分钟的处理前后检测Gcn4的功能,在热休克10分钟处理前后检测Hsf1和Msn2的功能,在半乳糖添加前后检测Gal4的功能(图三c和d)。正如预期,诱导条件可增加功能结合位点的数量,但重要的是,许多非功能结合位点仍然存在(图三c)。在诱导条件下,仍有许多表达靶点没有检测到TF的结合(图三d)。
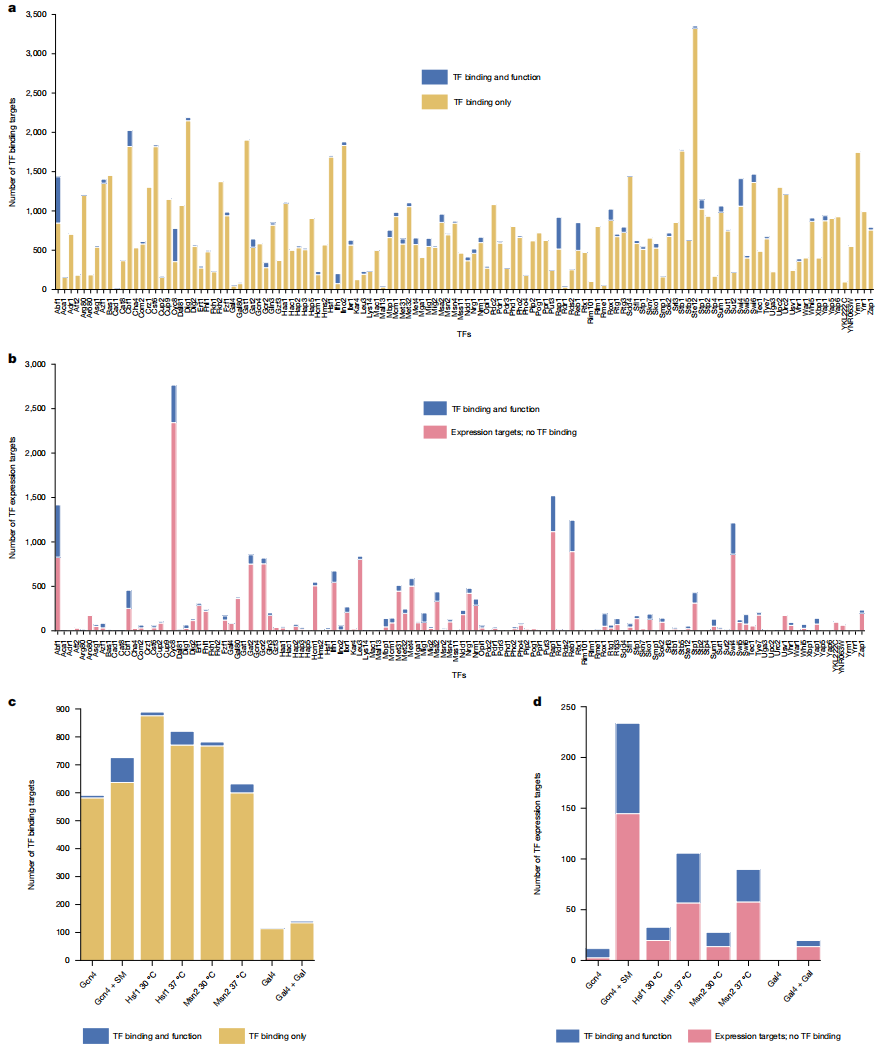
图三 TF结合和靶基因表达之间的重叠性较低[9]
最后,研究者探究在启动子没有检测到TF结合的情况下,TF如何调控靶基因的表达。在不同染色体上构建GFP报告基因,分别由四种TF(Abf1、Cyc8、Stp1和Sum1)强烈调控的结合或未结合的启动子(TSS的-400至+200bp)驱动表达,并测试TF耗竭反应(图四a)。在TF耗竭后,将大于两倍的转录变化定义为TF有效反应(图四b)。没有反应的启动子被列为阴性对照组(灰色),TF结合且做出反应的启动子为阳性对照(蓝色)。结果发现了两类TF未结合的启动子(粉色):保留了反应的启动子和没有反应的启动子。对于Abf1和Cyc8,七分之六的未结合启动子保留了TF依赖性反应(图四b),表明尽管在天然位置上没有检测到TF的结合,但TF是通过-400至+200 bp的启动子序列介导其功能的。相比之下,只有五分之一的Stp1和Sum1靶基因仍然依赖TF(图四b),表明在天然基因的背景下,这些无反应的启动子可能依赖于更远端的调控元件。
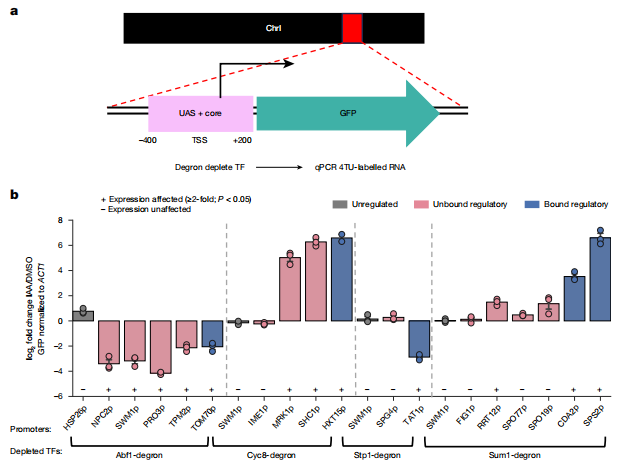
图四 一些TF未结合启动子可以直接响应TF的转录反应[9]
综上所述,该研究绘制了近乎完整的酿酒酵母转录因子结合和调控表达靶点图谱,全面解析了单个细胞类型的几乎所有TF与DNA的相互作用,以及TF对复杂的基因调控程序如何做出贡献。发现很少有TF具有专一的激活作用或抑制作用,大多数TF具有双重功能。尽管几乎所有蛋白质编码基因都受到一个或多个TF的调控,但TF结合和基因调控之间的重叠性有限。该研究为探究TF的作用及其全基因组调控转录的机制提供了重要且意想不到的见解。

GTP研发中心拥有自主知识产权的类精子干细胞介导的半克隆技术,可在体外实现类精子干细胞的多位点改造并进行功能元件测试,最后将类精子干细胞通过卵母细胞注射可以一步法获得基因改造小鼠。基于类精子干细胞单倍体打靶高效率的优势,可快速构建定制化小鼠,如基因组安全位点大片段敲入、条件性基因敲除/敲入和人源化基因改造等。多个复杂调控元件可以通过多次体外细胞打靶构建在同一株类精子干细胞中,经细胞水平质控后,一步法获得基因改造小鼠。

如有需要欢迎联系我们,GTP研发中心将竭诚为您设计最佳方案,制定专属于您的基因改造小鼠。
网址:http://www.sibcb.ac.cn/gtp/
地址:上海市岳阳路320号
责任编辑:多一百
新媒体运营:suway
参考文献
1. Lee TI, Young RA: Transcriptional regulation and its misregulation in disease. Cell 2013, 152(6):1237-1251.
2. Alerasool N, Leng H, Lin ZY, Gingras AC, Taipale M: Identification and functional characterization of transcriptional activators in human cells. Molecular cell 2022, 82(3):677-695.e677.
3. Sahu B, Hartonen T, Pihlajamaa P, Wei B, Dave K, Zhu F, Kaasinen E, Lidschreiber K, Lidschreiber M, Daub CO et al: Sequence determinants of human gene regulatory elements. Nature genetics 2022, 54(3):283-294.
4. Cusanovich DA, Pavlovic B, Pritchard JK, Gilad Y: The functional consequences of variation in transcription factor binding. PLoS genetics 2014, 10(3):e1004226.
5. Hu Z, Killion PJ, Iyer VR: Genetic reconstruction of a functional transcriptional regulatory network. Nature genetics 2007, 39(5):683-687.
6. Rossi MJ, Kuntala PK, Lai WKM, Yamada N, Badjatia N, Mittal C, Kuzu G, Bocklund K, Farrell NP, Blanda TR et al: A high-resolution protein architecture of the budding yeast genome. Nature 2021, 592(7853):309-314.
7. Hackett SR, Baltz EA, Coram M, Wranik BJ, Kim G, Baker A, Fan M, Hendrickson DG, Berndl M, McIsaac RS: Learning causal networks using inducible transcription factors and transcriptome-wide time series. Molecular systems biology 2020, 16(3):e9174.
8. Hahn S, Young ET: Transcriptional regulation in Saccharomyces cerevisiae: transcription factor regulation and function, mechanisms of initiation, and roles of activators and coactivators. Genetics 2011, 189(3):705-736.
9. Mahendrawada L, Warfield L, Donczew R, Hahn S: Low overlap of transcription factor DNA binding and regulatory targets. Nature 2025, 642(8068):796-804.
10. Nishimura K, Fukagawa T, Takisawa H, Kakimoto T, Kanemaki M: An auxin-based degron system for the rapid depletion of proteins in nonplant cells. Nature methods 2009, 6(12):917-922.



