技术分享:快速小鼠全身成像技术描绘亚细胞分辨率的周围神经图谱
神经系统是一个贯穿全身的复杂神经元互连网络,由中枢神经系统(CNS)和周围神经系统(PNS)组成,负责协调生理和心理活动[1-4]。可视化绘制这一复杂神经系统的解剖结构,对于理解其功能和相关疾病的机制至关重要。过去十年得益于三维光学显微镜技术的进步,亚细胞分辨率的全脑中尺度连接组图谱取得了显著进展[5-10]。然而,周围神经系统的类似分析仍面临挑战。与相对紧凑且均质的大脑不同,哺乳动物躯体的体积更大且高度异质,包含了多种组织类型和不规则结构。目前对于PNS结构的认知仍主要基于解剖学观察,仅能提供其路径、分支和连接模式的概貌。因此,在全身尺度上解析长距离、复杂且相互交织的PNS神经回路仍极为困难。
近期技术的发展尝试通过改进溶剂型和水性透明化方法实现沿着身体结构可视化PNS,如DISCO系列的wildDISCO实现了免疫标记小鼠的全身可视化,HYBRID和TESOS实现转基因荧光蛋白小鼠的全身成像。结合传统光片显微镜,这些透明化方法可实现三维重建PNS整体神经结构及其主要分支[11-15]。然而,这些方法无法在全鼠范围内均匀解析稀疏神经分支和单轴突纤维等精细结构。因此,亟需一种快速实现成年小鼠均匀高分辨率的全身成像方法,以支持CNS和PNS的中尺度连接组图谱研究。
2025年7月,Cell期刊发表了一套小鼠全身亚细胞分辨率三维成像技术,与荧光标记、免疫染色、AAV病毒神经示踪三种标记方法一起联用,可在40小时内完成微米级分辨率的成年小鼠全身成像。该技术包括三个关键组成部分:1)优化的全身均匀透明化和多功能标记流程,2)连续切片伴随切面超高速3D成像的blockface-VISoR系统,以及3)从连续3D图像集自动重建体积的算法和软件(图一A)。具体来说,通过对Thy1-EGFP小鼠中单个脊髓纤维的三维重建,揭示了腹侧支和背侧支感觉与运动投射的独特形态特征。通过免疫染色实现了交感神经及其分支的全身测绘,凸显了其在肢体肌肉、骨骼和多数内脏器官的血管伴行模式。AAV病毒神经示踪阐明了迷走神经的精细结构及单个迷走神经纤维的投射路径,意外发现了通向不同器官的投射路线。该研究揭示了周围神经系统的复杂结构,包括脊髓躯体运动和感觉神经、内脏交感神经和迷走神经,以及它们与全身各种非神经组织和器官的相互作用,为理解机体生理和疾病中不同系统的细胞间相互作用提供了整体性研究工具[16]。
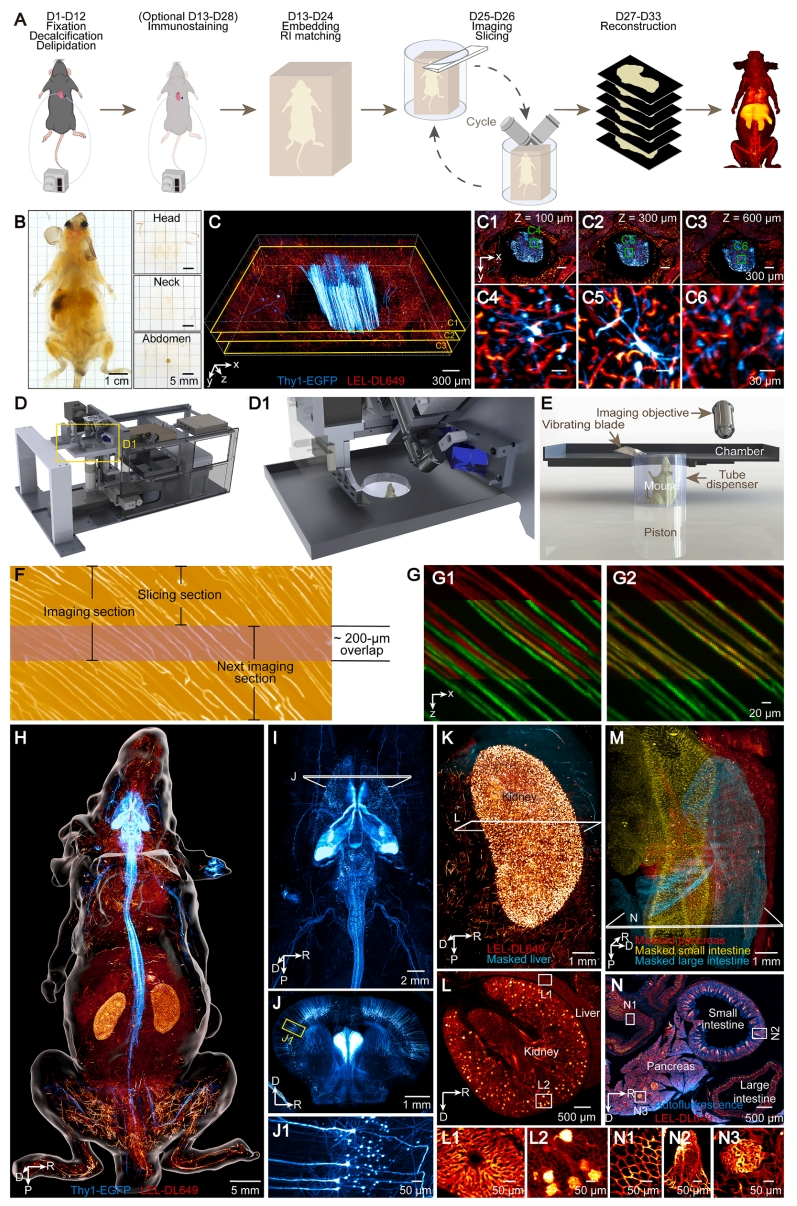
图一 ARCHmap-blockface-VISoR技术方法[16]
高质量样本制备对大体积成像至关重要。研究者建立了一套高效的水溶液透明化流程称为ARCHmap,包含CUBIC-LH透明化、水凝胶包埋和PuClear折射率匹配液。首先,评估多种现有技术对离体器官的透明化效果,选择基于水溶液的透明化方案CUBIC-L进行改良。改良方案CUBIC-LH在实现优异组织透明度的同时,还更好地保留内源性荧光蛋白信号,且组织体积变化最小。为解决切片过程中的样本变形问题,研究者开发了含羟甲基丙烯酰胺单体溶液(HMS)、牛血清白蛋白(BSA)和碘海醇的优化水凝胶包埋溶液,大幅增强了透明化小鼠躯体的机械强度。最后使用PuClear折射率匹配液[15, 17-19],实现全身组织的光学均匀透明化,可清晰观察皮下至少600 μm深度的结构(图一A-C)。
然后,为实现透明化小鼠全身均匀高分辨率成像,研究者开发了一套blockface-VISoR系统(图一D),该系统结合成像与精密切片两个模块。成像模块采用自主开发的超高速VISoR成像技术,精密切片模块配备了振动切片机,可实现微米级精度进样,两个模块相邻安装,样品管装载于平移台上(图一D和E)。成像步骤简要如下:将透明化小鼠推向切刀,被振动切片机平整切割,然后成像模块以200帧/秒速率采集倾斜视角图像,切割面成像后样本返回切片区推进400 μm,进行下一轮循环。平移台以0.7 mm/s匀速移动,相邻成像区域200 μm的重叠设计保障连续性并辅助重建对齐(图一E-G)。将神经元Thy1-EGFP转基因小鼠采用LEL-DL649标记血管,3D重建了小鼠全身性的血管-神经网络(图一H),包括大脑中单个神经元(图一I和J),肝脏肝窦的放射状脉管系统(图一K、L和L1)、肾脏肾小体的肾小球(图一K,L和L2)、肠粘膜和粘膜下层的毛细血管网络(图一M、N、N1和N2)以及胰腺内的朗格尔汉斯岛(图一M,N和N3),所有这些都来自同一个3D数据集。
为验证ARCHmap透明化技术与blockface-VISoR成像系统在PNS研究中的有效性,研究者对Thy1-EGFP小鼠头部(LEL-DL649血管标记)进行三维重建分析(图二A-D)。结果显示,该系统高分辨率清晰呈现了小鼠头部颅神经的微观构造,包括三叉神经(CN V,图二C1和C2)、视觉相关的滑车神经(CN IV,图二C3)、面神经(CN VII,图二C4)、牙周组织感觉神经元(图二D1和D2)、咬肌三叉神经运动末梢(图二D3和D4)。利用数据集的均质高分辨率,研究者对表达EGFP的三叉神经进行了全面详细分析。三叉神经的运动部分显示出许多厚束和密集的树枝状结构,这些结构通过广泛的爪状神经肌肉接头支配头面部肌肉(图二D和E)。值得注意的是,研究者观察到一个三叉神经感觉轴突束支配着切牙和磨牙的牙周组织(图二F和G),三叉神经对牙周组织的感觉传入神经(图二F)及神经末梢(图二F1)也清晰可见。对支配牙周组织的三叉神经运动神经元和感觉神经元单纤维追踪,可区分运动和感觉成分的不同路线(图二G)。以上结果表明,该技术实现了对传统技术难以解析的颅神经中尺度结构的高分辨率可视化。
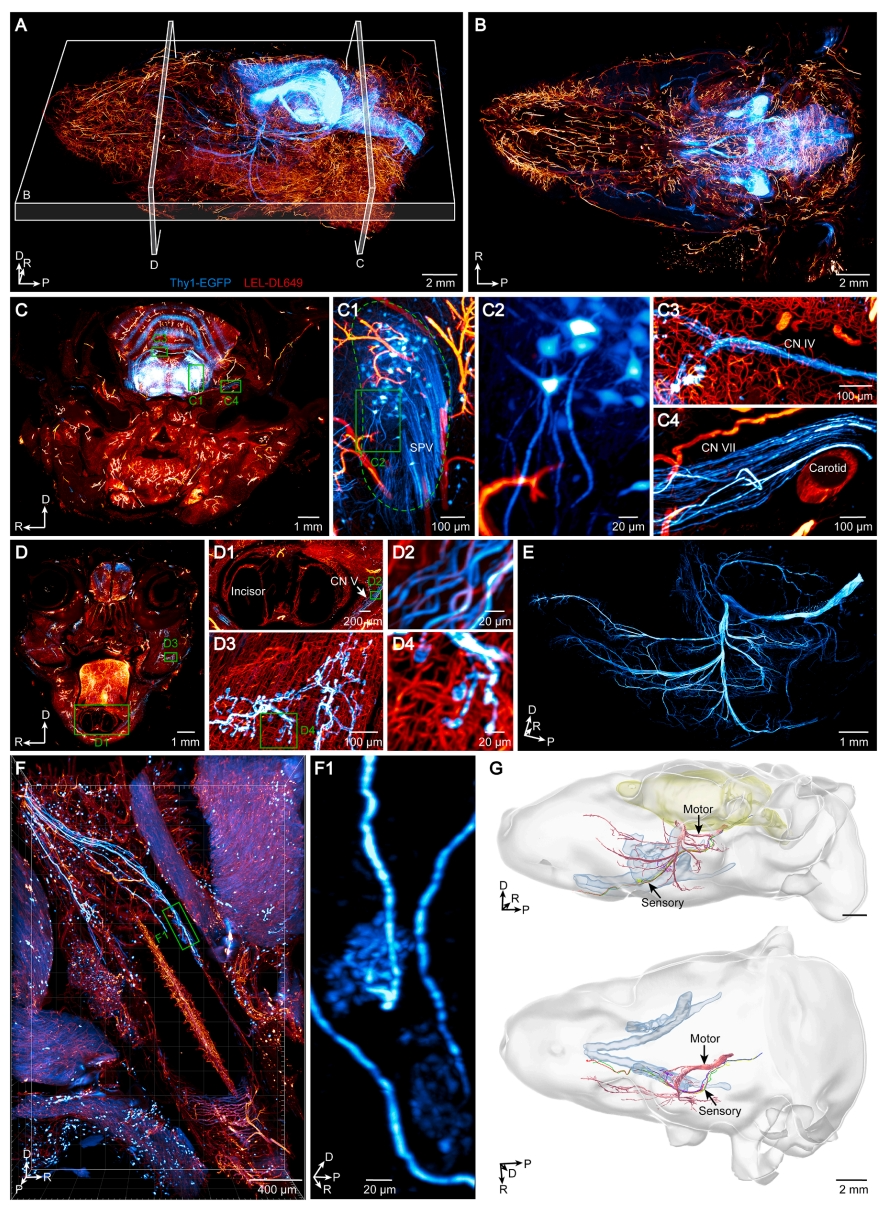
图二 小鼠头部颅神经的可视化[16]
在Thy1-EGFP小鼠中,利用EGFP在脊髓神经元中的稀疏表达特性,研究者通过ARCHmap-blockface-VISoR技术获取的全身图像中能够清晰区分单个神经纤维(图三A-C)。该技术成功追踪了胸段运动神经元(脊髓内)和感觉神经元(背根神经节内)长达8厘米的投射路径,还完整呈现了神经肌肉接头、游离神经末梢、矛状末梢、肌梭等远端结构的精细形态(图三D-I)。该技术能够识别单个运动和感觉神经元的完整结构,包括腹侧支神经元(图三D和E)和背侧支神经元(图三F-I)。腹侧支与背侧支神经元有着显著的形态学差异,在腹侧支中,所有运动纤维和一些感觉纤维沿着肋间向胸部和腹壁投射(图三D),放大区域显示的神经末梢为自由神经末梢(图三D1和E2)和披针形末端(图三D2)、背根神经节(DRG)中的假单极神经元(图三E1)。腹支中超过一半的感觉神经出现了大的V形转弯(图三E2)。在背侧支中,感觉神经和运动神经之间的形态差异变得更加明显,所有感觉神经都显示出具有多个锯齿状转弯的长轴索纤维(图三F),而胸椎运动纤维要短得多,并且表现出密集的树枝状(图三H和I),放大区域显示的是放大的神经肌肉接头区域(图三H1和I1)。
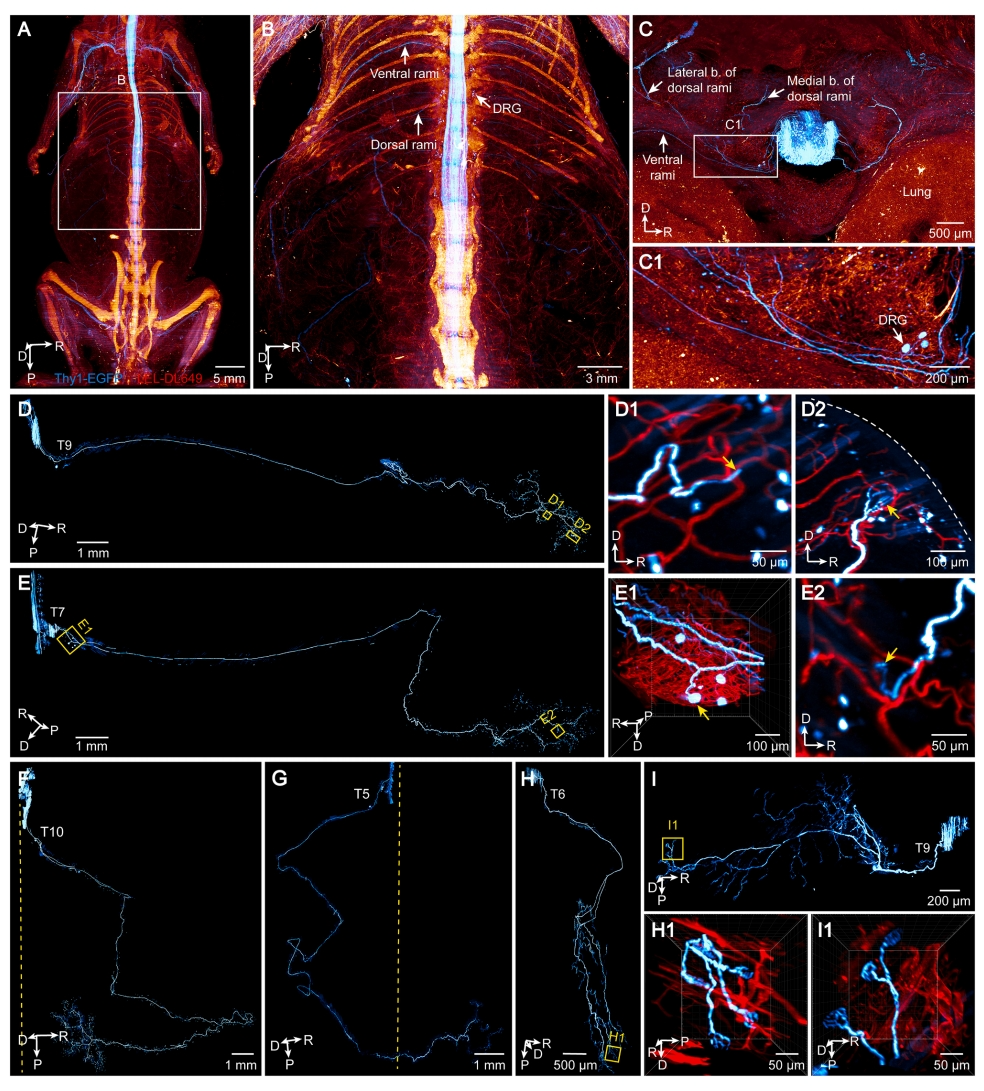
图三 小鼠胸段感觉和运动神经元的可视化[16]
然后,研究者追踪了从T2-T13段的191个表达EGFP的脊髓神经元的外周投射,包括来自腹侧支的66个运动神经元和66个感觉神经元,以及来自背侧支的30个运动神经和29个感觉神经元(图四A和B)。编译的投影图显示了不同脊髓神经元类型及其神经支配模式(图四A-D)。定量分析进一步表明,腹支和背支中单个脊髓运动和感觉神经元的长度和分支范围存在显著差异(图四E-J)。例如腹侧支运动纤维主长度从T2到T13呈递增趋势,而感觉纤维主长度分布相对较为均匀(图四F)。值得注意的是,背侧支运动纤维在T2-T10节段的分支数量和总长度随节段增加(图四G-J)。
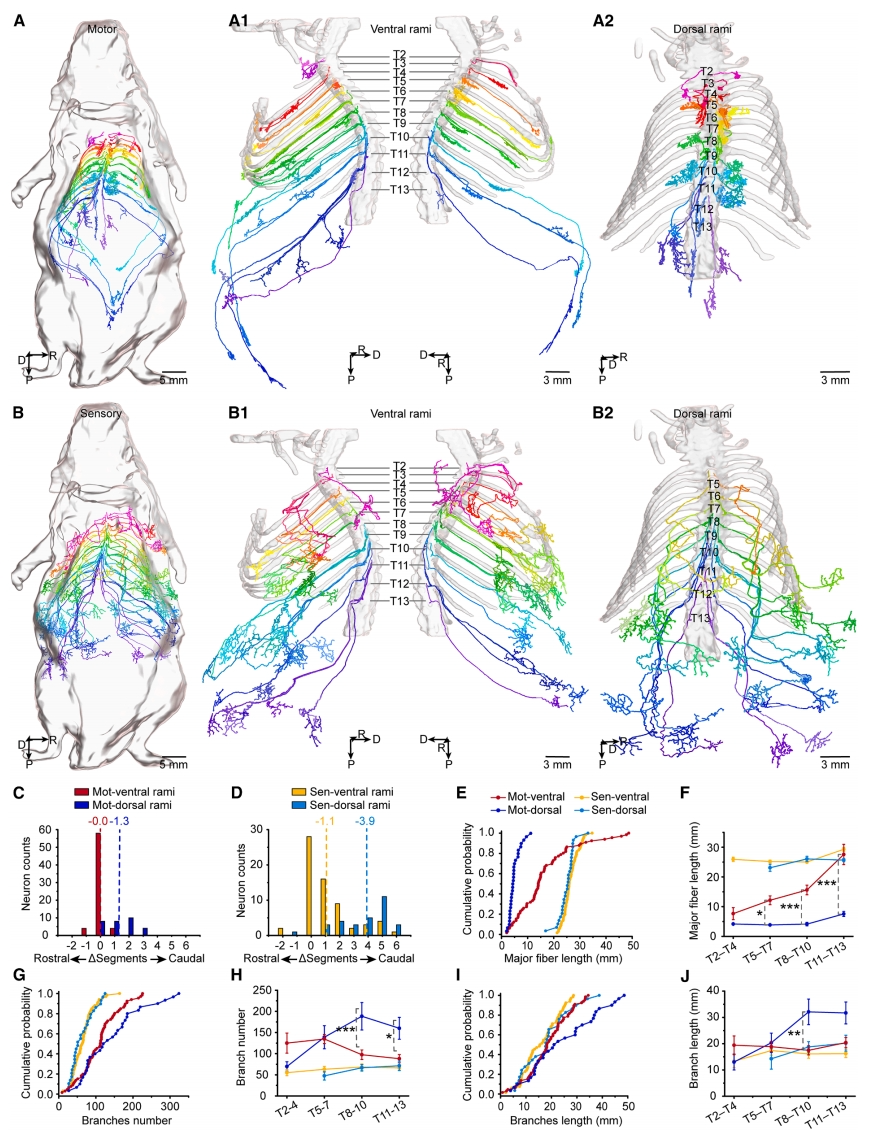
图四 胸段感觉和运动神经元的解剖学比较和分析[16]
交感神经系统广泛分布在全身,调节器官和脉管系统的生理活动[20-24]。尽管既往研究曾对啮齿类动物离体器官或灵长类局部器官的交感神经精细结构进行过观察,但迄今仍缺乏全身交感神经投射的高分辨率图谱[25-28]。由此,为了评估该技术与免疫标记的兼容性,研究者将酪氨酸羟化酶(TH)抗体与该技术整合到一起,实现了交感神经系统的高分辨率成像,可以清晰观察到交感神经链、神经丛及其连接相邻的椎旁神经节如颈上神经节(SCG)和椎前神经节(PVG)的连接分支(图五A-C)。通过DBH-Cre;Ai6小鼠进行验证,TH免疫标记与内源性ZsGreen荧光蛋白标记的交感神经节后神经元及纤维高度共定位(图五C),证实了标记的特异性。脑内TH+儿茶酚胺神经元及纤维呈现强标记信号(图五D和E)。通过将交感神经系统的均匀高分辨率可视化与LEL-DL649血管标记相结合,研究者进一步评估了交感神经如何与整个小鼠体内的血管相互作用。例如,腰骶骨区域交感神经与血管伴行,形成骶骨及骶髂关节周围的网状结构(图五F)。此外,下肢骨骼肌中交感神经沿血管形成密集的外周血管丛(图五G)。这种交感神经投射的血管周围模式在躯干和四肢的各个区域广泛存在。
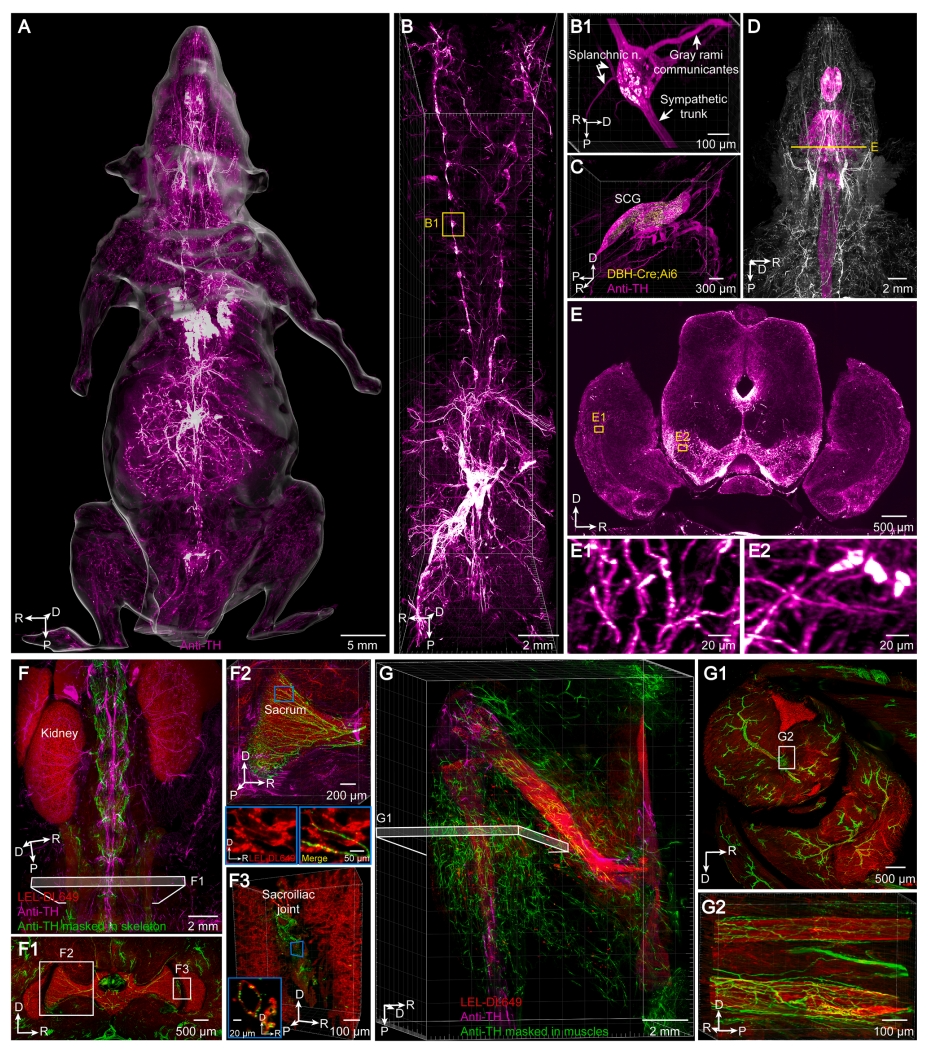
图五 小鼠免疫染色交感神经的可视化[16]
交感神经及其精细分支的全身可视化可以系统地绘制其结构,揭示不同内脏器官的不同投射路线和神经支配模式。在颅骨区域,有一条交感神经投射到耳蜗的骨螺旋板和软轴,形成风车状树枝状结构(图六A和B)。值得注意的是,TH抗体还能标记多巴胺能纤维,例如已知支配位于风车外缘的内毛细胞的外侧橄榄耳蜗神经元的多巴胺能纤维(图六A1)。在腹部,从PVG投射到肾脏的交感神经穿透肾实质,并缠绕在血管周围(图六C和D),神经节包裹肾小球清晰可见(图六C1)。此外,脾脏、肝脏和胰腺的交感神经支配表现出明显的血管伴行模式(图六E)。追踪腹腔肠系膜上神经节(CSMG)可通往不同内脏器官进行投射,包括脾脏、胰腺、肝脏和胃(图六F)。另外,横膈膜肌部分接收来自CSMG和膈神经节的交感输入,而肋膈肌区由胸段椎旁神经节支配(图六G-I)。总的来说,该研究通过高分辨率成像技术,首次在全身尺度揭示了交感神经系统的精细解剖特征及其器官特异性分布规律。
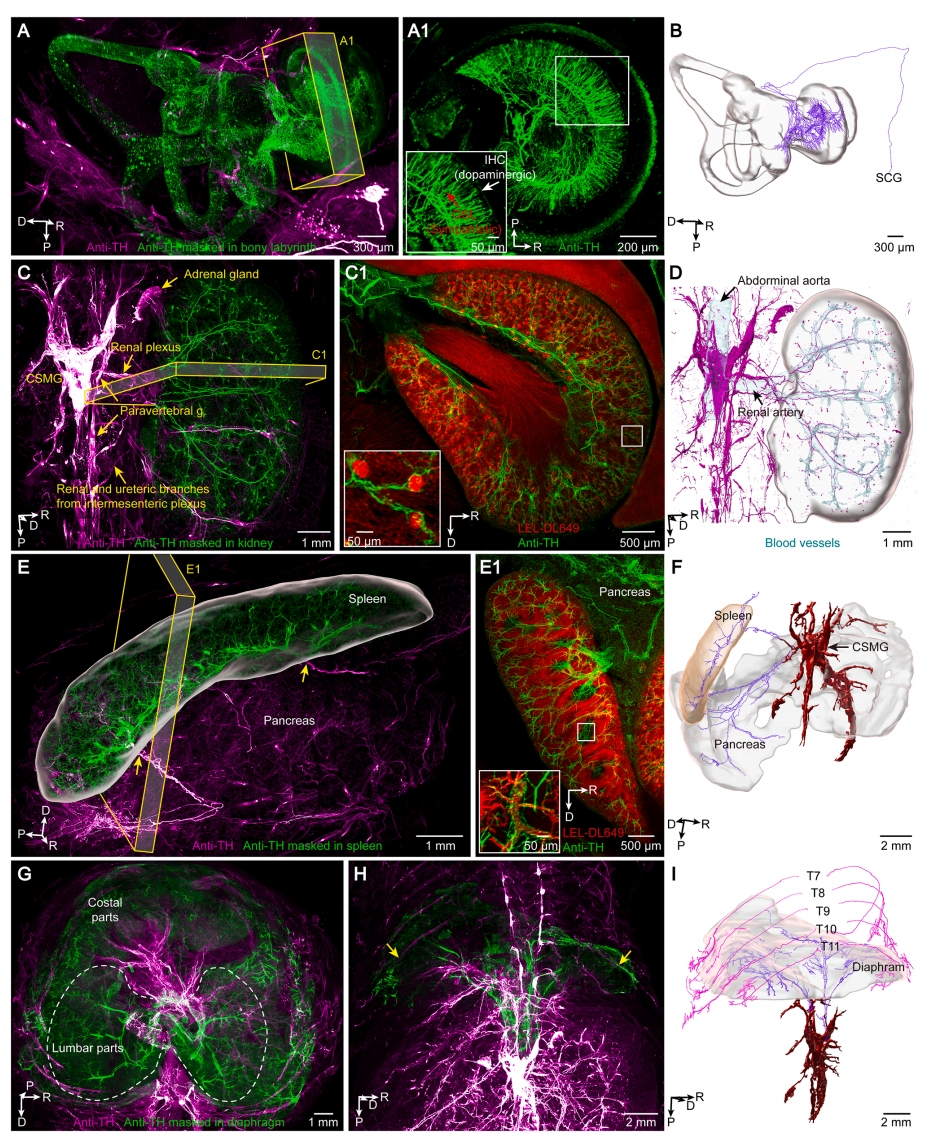
图六 器官交感神经支配的可视化[16]
最后,研究者评估了该技术在病毒神经示踪标记周围神经中的适用性。向小鼠左右两侧迷走神经背核(DMV)注射AAV2/9-EF1α-tdTomato,并向左侧迷走神经节(NG,包括结状节NG与颈静脉节JG)注射AAV2/9-CAG-EYFP。全鼠成像成功追踪了迷走神经运动与感觉成分及其向各器官的投射路径(图七A)。在颈部水平,迷走神经干的感觉与运动纤维保持空间分离,但在远端节段逐渐混合(图七B)。胸段水平还接收对侧神经束,呈现更复杂的分布模式(图七C4),这为感觉与运动纤维束的解剖组织提供了高分辨率视图,弥补了既往显微CT研究的不足[29]。基于这一映射,观察到左右喉返神经(RLN)之间及左右迷走神经干之间的多重交叉连接(图七B)。通过EYFP标记左侧迷走感觉神经元,在三只实验小鼠的两只中观察到左侧RLN纤维在不同位点交叉至右侧支配喉部;而tdTomato标记的RLN运动纤维表现出广泛交叉,在喉部肌肉内形成密集的树枝状结构(图七D和E)。在三只病毒示踪标记的实验小鼠中,标记的迷走运动与感觉纤维专一性支配咽、喉、食管、肺、胃及肠道(图七A、E和F)。腹部三维重建显示,左右迷走神经干的胃支分别密集支配胃前壁与后壁(图七F和F1),放大视图显示了束状轴突形成的肌间神经丛网状结构(图七F2-F4)。为进一步评估该技术的单纤维追踪方法的能力,研究者在胃部注射AAV9-retro-CAG-Cre,在DMV注射AAV2/9-EF1α-DIO-tdTomato,对胃投射迷走运动纤维进行稀疏标记(图七G)。结果显示,DMV中三个迷走神经元及其完整纤维清晰可见(图七H),其中两条纤维沿同侧迷走神经干投射至胃前/后壁,第三条纤维从左DMV发出后在膈上位置交叉至对侧,然后沿着右迷走纤维旁向头侧移动,随后急转弯向尾侧移动,最终终止于胃后壁(图七H1、I和J)。
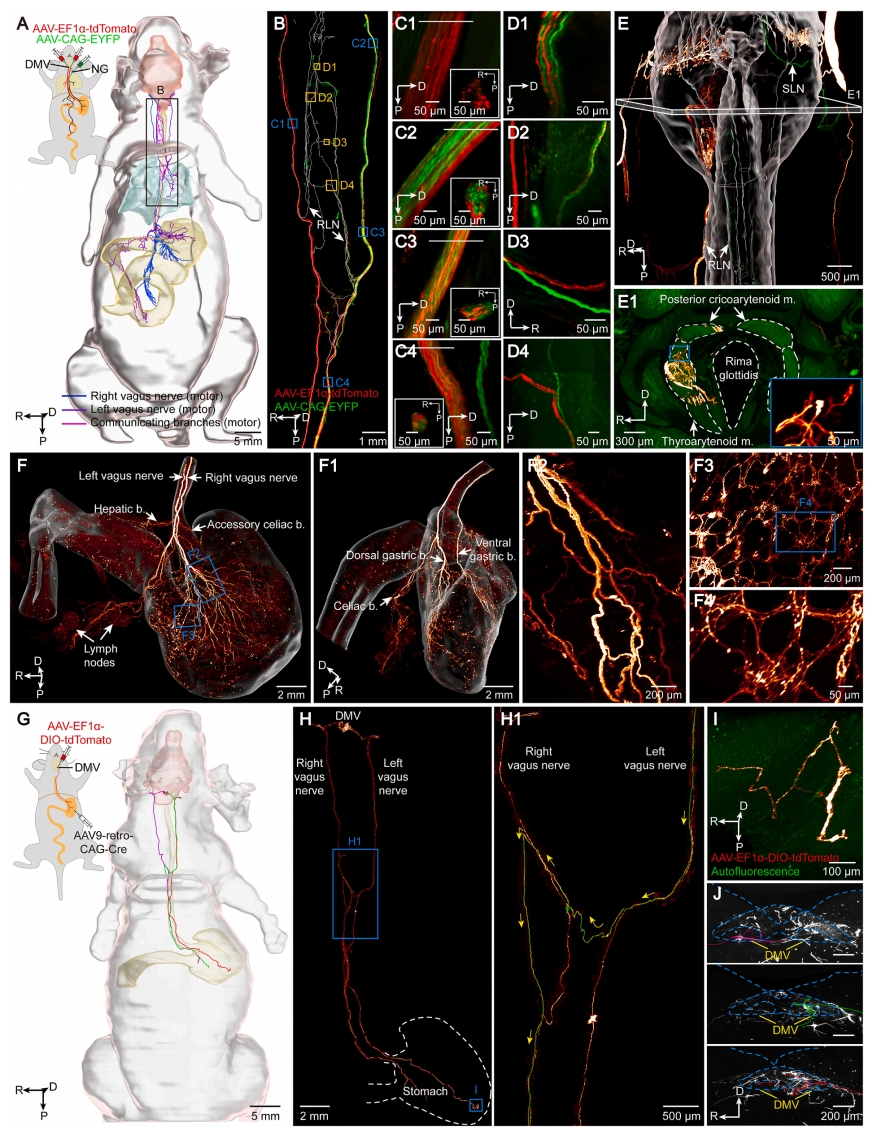
图七 病毒示踪标记的迷走神经可视化和单个神经元追踪[16]
综上所述,该研究开发的ARCHmap透明化技术与blockface-VISoR成像系统,实现了在40小时内以均匀的亚细胞分辨率对整个成年小鼠进行成像,在宏观和微观尺度上有效可视化周围神经系统(PNS),重建了Thy1-EGFP脊髓神经元和跨段投射,绘制了免疫标记的交感神经投射及其血管周围模式,解析了病毒示踪标记的迷走神经结构和复杂的单纤维通路。该研究通过技术创新实现了周围神经系统的"全器官-单细胞"尺度解析,不仅修正了传统解剖学认知,更是首次建立了成年小鼠全身神经-器官互作图谱,为生理机制研究和疾病治疗策略开发奠定了方法论基础。

GTP研发中心拥有自主知识产权的类精子干细胞介导的半克隆技术,可在体外实现类精子干细胞的多位点改造并进行功能元件测试,最后将类精子干细胞通过卵母细胞注射可以一步法获得基因改造小鼠。基于类精子干细胞单倍体打靶高效率的优势,可快速构建定制化小鼠,如基因组安全位点大片段敲入、条件性基因敲除/敲入和人源化基因改造等。多个复杂调控元件可以通过多次体外细胞打靶构建在同一株类精子干细胞中,经细胞水平质控后,一步法获得基因改造小鼠。

如有需要欢迎联系我们,GTP研发中心将竭诚为您设计最佳方案,制定专属于您的基因改造小鼠。
网址:http://www.sibcb.ac.cn/gtp/
地址:上海市岳阳路320号
责任编辑:甜点
新媒体运营:suway
参考文献
1. Dinan TG, Cryan JF: The Microbiome-Gut-Brain Axis in Health and Disease. Gastroenterol Clin North Am 2017, 46(1):77-89.
2. Matsubara Y, Kiyohara H, Teratani T, Mikami Y, Kanai T: Organ and brain crosstalk: The liver-brain axis in gastrointestinal, liver, and pancreatic diseases. Neuropharmacology 2022, 205:108915.
3. Kleinfeld D, Bharioke A, Blinder P, Bock DD, Briggman KL, Chklovskii DB, Denk W, Helmstaedter M, Kaufhold JP, Lee WC et al: Large-scale automated histology in the pursuit of connectomes. J Neurosci 2011, 31(45):16125-16138.
4. Simon SA, de Araujo IE, Gutierrez R, Nicolelis MA: The neural mechanisms of gustation: a distributed processing code. Nat Rev Neurosci 2006, 7(11):890-901.
5. Ueda HR, Dodt HU, Osten P, Economo MN, Chandrashekar J, Keller PJ: Whole-Brain Profiling of Cells and Circuits in Mammals by Tissue Clearing and Light-Sheet Microscopy. Neuron 2020, 106(3):369-387.
6. Brunner C, Grillet M, Sans-Dublanc A, Farrow K, Lambert T, Mace E, Montaldo G, Urban A: A Platform for Brain-wide Volumetric Functional Ultrasound Imaging and Analysis of Circuit Dynamics in Awake Mice. Neuron 2020, 108(5):861-875 e867.
7. Ragan T, Kadiri LR, Venkataraju KU, Bahlmann K, Sutin J, Taranda J, Arganda-Carreras I, Kim Y, Seung HS, Osten P: Serial two-photon tomography for automated ex vivo mouse brain imaging. Nat Methods 2012, 9(3):255-258.
8. Qiu S, Hu Y, Huang Y, Gao T, Wang X, Wang D, Ren B, Shi X, Chen Y, Wang X et al: Whole-brain spatial organization of hippocampal single-neuron projectomes. Science 2024, 383(6682):eadj9198.
9. Peng H, Xie P, Liu L, Kuang X, Wang Y, Qu L, Gong H, Jiang S, Li A, Ruan Z et al: Morphological diversity of single neurons in molecularly defined cell types. Nature 2021, 598(7879):174-181.
10. Hintiryan H, Bowman I, Johnson DL, Korobkova L, Zhu M, Khanjani N, Gou L, Gao L, Yamashita S, Bienkowski MS et al: Connectivity characterization of the mouse basolateral amygdalar complex. Nat Commun 2021, 12(1):2859.
11. Mai H, Luo J, Hoeher L, Al-Maskari R, Horvath I, Chen Y, Kofler F, Piraud M, Paetzold JC, Modamio J et al: Whole-body cellular mapping in mouse using standard IgG antibodies. Nat Biotechnol 2024, 42(4):617-627.
12. Yi Y, Li Y, Zhang S, Men Y, Wang Y, Jing D, Ding J, Zhu Q, Chen Z, Chen X et al: Mapping of individual sensory nerve axons from digits to spinal cord with the transparent embedding solvent system. Cell Res 2024, 34(2):124-139.
13. Cai R, Pan C, Ghasemigharagoz A, Todorov MI, Forstera B, Zhao S, Bhatia HS, Parra-Damas A, Mrowka L, Theodorou D et al: Panoptic imaging of transparent mice reveals whole-body neuronal projections and skull-meninges connections. Nat Neurosci 2019, 22(2):317-327.
14. Nudell V, Wang Y, Pang Z, Lal NK, Huang M, Shaabani N, Kanim W, Teijaro J, Maximov A, Ye L: HYBRiD: hydrogel-reinforced DISCO for clearing mammalian bodies. Nat Methods 2022, 19(4):479-485.
15. Susaki EA, Ueda HR: Whole-body and Whole-Organ Clearing and Imaging Techniques with Single-Cell Resolution: Toward Organism-Level Systems Biology in Mammals. Cell Chem Biol 2016, 23(1):137-157.
16. Shi MY, Yao Y, Wang M, Yang Q, Ding L, Li R, Li Y, Huang H, Yang CY, Zhou Z et al: High-speed mapping of whole-mouse peripheral nerves at subcellular resolution. Cell 2025, 188(14):3897-3915 e3820.
17. Tainaka K, Kuno A, Kubota SI, Murakami T, Ueda HR: Chemical Principles in Tissue Clearing and Staining Protocols for Whole-Body Cell Profiling. Annu Rev Cell Dev Biol 2016, 32:713-741.
18. Ueda HR, Erturk A, Chung K, Gradinaru V, Chedotal A, Tomancak P, Keller PJ: Tissue clearing and its applications in neuroscience. Nat Rev Neurosci 2020, 21(2):61-79.
19. Richardson DS, Lichtman JW: Clarifying Tissue Clearing. Cell 2015, 162(2):246-257.
20. Triposkiadis F, Karayannis G, Giamouzis G, Skoularigis J, Louridas G, Butler J: The sympathetic nervous system in heart failure physiology, pathophysiology, and clinical implications. J Am Coll Cardiol 2009, 54(19):1747-1762.
21. Cole SW, Nagaraja AS, Lutgendorf SK, Green PA, Sood AK: Sympathetic nervous system regulation of the tumour microenvironment. Nat Rev Cancer 2015, 15(9):563-572.
22. Scott-Solomon E, Boehm E, Kuruvilla R: The sympathetic nervous system in development and disease. Nat Rev Neurosci 2021, 22(11):685-702.
23. Muller PA, Schneeberger M, Matheis F, Wang P, Kerner Z, Ilanges A, Pellegrino K, Del Marmol J, Castro TBR, Furuichi M et al: Microbiota modulate sympathetic neurons via a gut-brain circuit. Nature 2020, 583(7816):441-446.
24. Wang T, Teng B, Yao DR, Gao W, Oka Y: Organ-specific sympathetic innervation defines visceral functions. Nature 2025, 637(8047):895-902.
25. Messal HA, Almagro J, Zaw Thin M, Tedeschi A, Ciccarelli A, Blackie L, Anderson KI, Miguel-Aliaga I, van Rheenen J, Behrens A: Antigen retrieval and clearing for whole-organ immunofluorescence by FLASH. Nat Protoc 2021, 16(1):239-262.
26. Adori C, Daraio T, Kuiper R, Barde S, Horvathova L, Yoshitake T, Ihnatko R, Valladolid-Acebes I, Vercruysse P, Wellendorf AM et al: Disorganization and degeneration of liver sympathetic innervations in nonalcoholic fatty liver disease revealed by 3D imaging. Sci Adv 2021, 7(30).
27. Hasegawa S, Nangaku M: Whole-Kidney Three-Dimensional Staining with CUBIC. J Vis Exp 2022(185).
28. Guillot J, Dominici C, Lucchesi A, Nguyen HTT, Puget A, Hocine M, Rangel-Sosa MM, Simic M, Nigri J, Guillaumond F et al: Sympathetic axonal sprouting induces changes in macrophage populations and protects against pancreatic cancer. Nat Commun 2022, 13(1):1985.
29. Jayaprakash N, Song W, Toth V, Vardhan A, Levy T, Tomaio J, Qanud K, Mughrabi I, Chang YC, Rob M et al: Organ- and function-specific anatomical organization of vagal fibers supports fascicular vagus nerve stimulation. Brain Stimul 2023, 16(2):484-506.



