技术分享:绘制人类组织特异性蛋白质关联图谱
蛋白质-蛋白质相互作用介导了细胞的生物物理结构和功能,这种相互作用的破坏可能导致疾病。解析人类蛋白质的相互作用组至今仍是一个挑战[1]。尽管已将质谱(MS)结合高通量筛选方法如蛋白质片段互补、酵母双杂交、亲和纯化(AP)和共分馏,使得在蛋白质组范围内发现新的相互作用[2, 3]。这些实验数据集通过基于机器学习的现代计算方法进行补充,共同构成了众多公开可用的高质量蛋白质相互作用组数据库。但是,这些汇聚全局互作信息的数据库,缺少了组织或细胞特异性的互作信息,且不到半数的蛋白质组可在所有组织中检测到。
阐明蛋白质相互作用的组织特异性对于理解细胞类型特异性功能、寻找药物靶点以及系统性理解人类细胞非常重要。可是,确定相互作用组的组织特异性是困难的。最初尝试利用基因表达数据来鉴定出共表达基因,或者根据给定组织的缺乏表达来排除蛋白质[4]。然而,这种通过mRNA共表达来预测蛋白质关联性的准确度有限,目前尚不清楚基因表达水平在多大程度上驱动蛋白质相互作用的变化。一些科学家尝试直接建立组织特异性的相互作用组[5],但即便是单个组织,也需要大量的组织资源;或者使用永生化细胞系以及其他模型,但获得的结果可能无法充分代表人类组织。
蛋白质复合物往往是由特定化学计量组装的共表达亚基所组成,而孤儿亚基通常会被降解[6]。蛋白质组学测量的复合物亚基组分比例的变化可用于确定疾病状态下的蛋白质相互作用的改变[7]。因此,可利用蛋白质的共丰度(coabundance)来建立蛋白质的关联性[8]。共丰度是指在样本中的蛋白质表现出共同的丰度变化,已被证明可用于准确预测蛋白质-蛋白质关联性。基于人类癌症大规模蛋白质组学研究的不断增加,以及遗传异质性介导了不同的细胞变化,使得系统性建立人类组织特异的蛋白质关联性成为可能。
2025年5月,Nature Biotechnology期刊报道了一项研究,绘制了一张人类组织特异性的蛋白质关联性图谱,可用于候选致病基因的优先排序。利用7811个人类组织样本的蛋白质丰度数据,对11种组织中1.16亿对蛋白质的关联可能性进行评分。发现超过25%的蛋白质关联性具有组织特异性,其中仅7%归因于基因的差异表达。稳定蛋白质复合物的相互作用在各组织中高度保守,而细胞类型特异性结构如突触成分是组织间差异的重要驱动因素[9]。
研究者首先从一个癌症队列的蛋白质组学研究中收集蛋白质丰度数据,收集了一个包含14种人体组织50项研究的数据集,包括5726个肿瘤样本和2085个癌旁健康组织样本(图一a)。同时纳入2930个肿瘤和722个健康样本的配对mRNA表达数据。由于蛋白质复合物成员在转录和转录后高度共调节,因此可根据丰度数据计算蛋白质-蛋白质关联性概率(图一b)。简单来说,通过计算队列中所有可能蛋白质对之间的Pearson相关性组成的相关矩阵来计算共丰度估计值。每对蛋白质必须在至少30个相同样本中进行定量,用于计算样本间配对丰度。将蛋白质复合物的成对亚基作为基准真实值(CORUM),使用逻辑模型将丰度估计值转换为蛋白质-蛋白质关联性概率。为测试获得的关联性概率复现已知蛋白质复合物成员的能力,计算从蛋白质共丰度、mRNA共表达和蛋白质共分馏中得出概率的受试者工作特征曲线(ROC,图一c)。发现蛋白质共丰度(曲线下面积AUC = 0.80 ± 0.01)的表现优于蛋白质共分馏(AUC = 0.69 ± 0.01) 和mRNA共表达(AUC = 0.70 ± 0.01,图一d)。此外,mRNA和蛋白质共丰度数据的结合并没有显著提高已知蛋白质复合物成员的复现率(图一e),表明转录后过程的调节驱动了蛋白质关联性的大部分预测能力,而不是基因表达驱动。因此,选择只使用蛋白质共丰度来计算关联性概率。使用所有研究量化了共计1115405个关联性概率,发现来自同一组织的重复队列通常聚类在一起,例如血液、大脑、肝脏和肺(图一f)。接着,筛选出组织特异性的关联,即关联的平均概率超过给定组织的第95个百分位,并且在所有的其他组织中平均概率低于0.5。结果显示,与来自不同组织的队列相比,组织特异性关联主要在同一来源组织的队列中复现(图一g)。以上结果表明,来源的组织是队列之间差异的主要驱动因素。

图一 计算蛋白质共丰度优于mRNA共表达和蛋白质共分馏,可复现蛋白质相互作用组[9]
利用代表来源组织的重复队列,将同一组织队列的关联概率汇总为单一关联得分(图二a),发现肿瘤来源的11种组织的评分均优于健康组织的评分(图二b)。于是,根据健康组织的评分计算出组织特异性关联,然后用肿瘤组织进行复现,发现肿瘤组织的评分主要复现了同一健康组织的组织特异性关联(图二c),表明共丰度计算获得的组织特异性关联评分可复现已知的蛋白质相互作用,具有可重复性和代表性。通过平均每个组织队列的关联概率,定义了一个蛋白质关联性图谱,包含了所有量化的蛋白质对关联得分,由此构建了一张包含11种人体组织1.16亿对蛋白质的关联性图谱(图二d)。平均而言,每个组织包含56 ± 6.2百万对蛋白质的关联性得分,其中10 ± 1.0 百万对蛋白质可能相关。
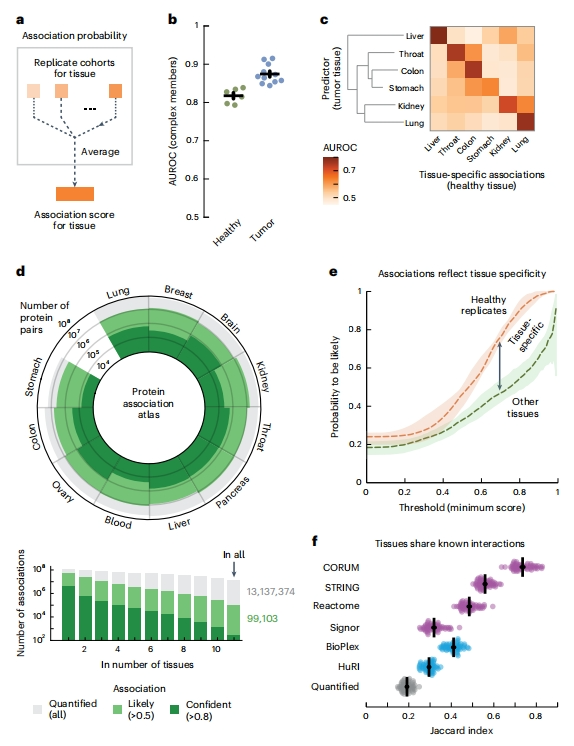
图二 人体组织中蛋白质相互作用可能性评分的关联性图谱构建[9]
众所周知,驱动组织间蛋白质相互作用差异的一个因素是基因表达。在给定组织中定量的蛋白质通常富含该组织表达升高的基因。然而,计算的组织之间关联性差异中,只有7%可以通过基因表达的差异来解释,进一步支持之前的推测,即蛋白质共丰度主要是由转录后过程所驱动的。将评分大于0.5定义为可能性关联,使用阈值关联评分来量化重复组织中的可能性关联(图二e,橙色曲线)。正如预期,可能性关联百分比随着阈值评分的增加而增加,46.3%的可能性关联和90.2%的高置信关联(评分 > 0.8)来自重复组织。当仅比较关联性图谱中的组织对时,这些百分比分别降至32.9%和54.6%(图二e,绿色曲线)。根据阈值评分,发现18.8%至34.0%的可能性关联是组织特异性的,因而预计超过25.8%(18.8%+7%)的蛋白质关联性具有组织特异性。当可能性关联限制在通过高通量筛选如酵母双杂交或亲和纯化(AP)实验鉴定出的相互作用上时,组织对之间的相似性增加了(图二f)。同样,限制在信号传导、生物途径、物理关联或人类蛋白质复合物鉴定的相互作用上时,组织对之间的相似性也会增加(图二f)。
接下来,研究者探究关联图谱中的细胞类型特异性关联,以AP2接头复合物为例。AP2复合物除了具有所有细胞通用的功能外,还有神经元特异性功能[10]。事实上,AP2复合物的亚基在所有组织中都是共存的,亚基之间的平均关联得分为0.80。91种蛋白质与所有组织中的所有AP2亚基有关联性评分,并且与AP2相关。与其他组织相比,51种突触蛋白与大脑中AP2复合物的关联得分较高(平均得分0.54) ,相反,非突触相互作用蛋白与大脑中AP2复合物的关联得分较低(平均得分0.33,图三a)。进一步举例显示疾病条件下的细胞类型特异性关联,发现血红蛋白的蛋白质互作组与贫血相关,且仅存在于血液中;乳糜微粒的亚基(从肠道运输乳脂质)与克罗恩病相关蛋白质具有相关性,且仅限于结肠;肝脏合成的纤维蛋白原亚基与肝脏疾病相关,且仅限于肝脏(图三b),表明关联性图谱可用于研究蛋白质复合物的组织特异性功能和疾病基因的关联。
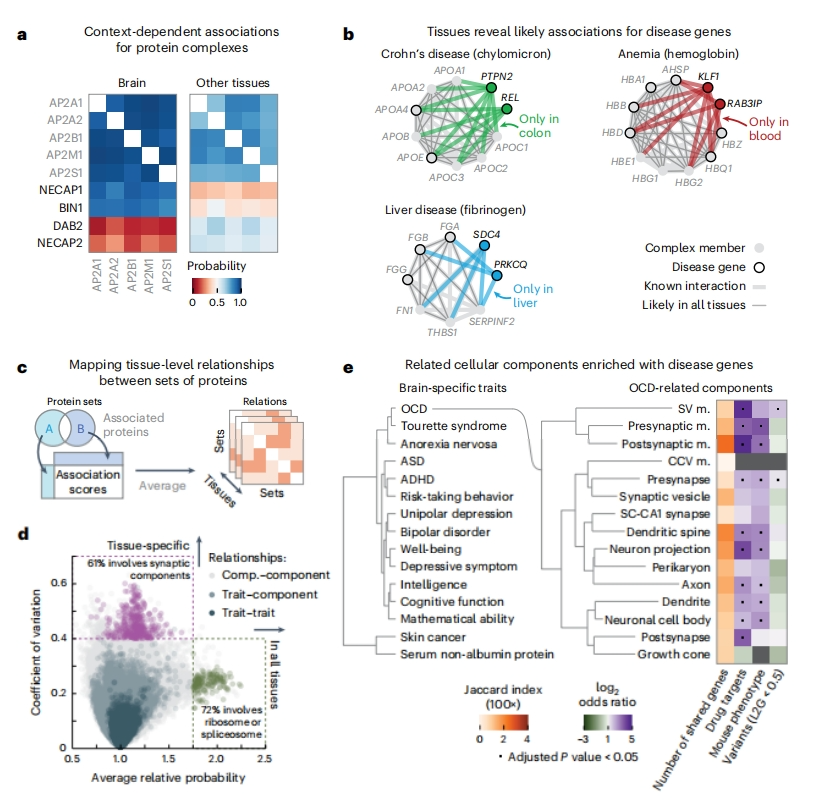
图三 关联性评分可定义蛋白质集之间的关系[9]
研究者试图概括这些蛋白质复合物的上下游特异性关联。通过基因本体论(GO)定义了细胞成分,并根据全基因组关联研究(GWAS)开放靶点(OTAR)位点与基因(L2G)评分定义其人类特征[11]。然后用组间所有可能蛋白质对的中值关联得分对其关系进行评分(图三c)。在所有组织中,关系得分较高的主要是核心细胞成分,如核糖体和剪接体,而跨组织变化最大的关系得分通常涉及组织特异性结构,如突触成分(图三d),表明关系评分以组织特异性的方式概括了蛋白质集的相关性,特别是大脑。研究者选择了15个特定于大脑的性状,其中13个确实与大脑有关。根据大脑的性状-性状关系得分进行聚类,揭示了大脑性状的层次结构,发现神经性厌食症、强迫症(OCD)和图雷特综合征等共同发生的疾病紧密地聚类在一起(图三e,左树状图)。进一步确定与强迫症(OCD)最强脑特异性关系的15种细胞成分,除了一种外,所有成分都与神经元有关(图三e,右侧树状图)。并且,大多数的细胞成分与强迫症高置信相关基因几乎没有共同点(图三e,橙色热图)。然而,这15个细胞成分比其他含有强迫症相关基因的细胞成分(药物靶点、小鼠强迫症相关基因和通过GWAS确定的与强迫症关系不太可靠的基因)富集了更多的强迫症相关基因(图三e,紫绿色热图),表明组织特异性关联可以通过功能关联促进疾病相关基因的优先排序。
于是,研究者构建了一个精神分裂症(SCZ)相关基因的大脑相互作用网络。首先通过GWAS研究获取了369个SCZ相关基因,计算每个组织中与SCZ最具组织特异性关系的前25个性状和细胞成分(图四a),进而提供了一组SCZ相关基因。然后,对于每个组织,筛选出一个SCZ基因和一个SCZ相关基因的蛋白质对,限定其关联得分超过组织得分的第97百分位,从而形成SCZ相关基因的组织特异性关联网络。从这一大脑网络中删除SCZ基因后,剩余的基因仍然富含小鼠SCZ相关基因、SCZ药物靶点和其他SCZ相关变体,并且这种富集是大脑特有的(图四b)。为验证这一SCZ相关基因的预测关联网络,通过使用人脑细胞下拉实验建立了一个相互作用数据集,包含了30种诱饵蛋白的7887个人脑相互作用,并已纳入IntAct数据库[12]。结果显示,计算的SCZ相关基因关联性在SCZ相关诱饵蛋白的相互作用组中强烈富集(图四c)。获得了205个已验证的SCZ相关基因的大脑相互作用组成网络,仅展示了具有先前证据的突触基因(图四d)。这一可视化网络包含了56个蛋白质,通过66个已验证的大脑相互作用与三种诱饵蛋白相连。这些连接的蛋白质包括了SCZ药物靶点,小鼠SCZ相关蛋白质以及先前证据较弱的SCZ相关蛋白质(图四d)。使用AlphaFold2预测了这205个蛋白质相互作用的结构,与CORUM中已知复合物成员的模型相比,预测的模型具有更高置信的相互作用。总共确定了15个中等置信度的交互作用,包括所有三种14-3-3蛋白(YWHAG、YWHAH和YWHAZ)与HCN1的特异性结合(图四e)。这三个模型的界面重叠,位于HCN1的C端无序区(残基775-802),其中一个预测的14-4-3结合位点已通过下拉实验得到验证。有研究表明,14-3-3蛋白与HCN1的结合依赖于S789磷酸化,这一相互作用可能抑制了HCN1的降解[13]。
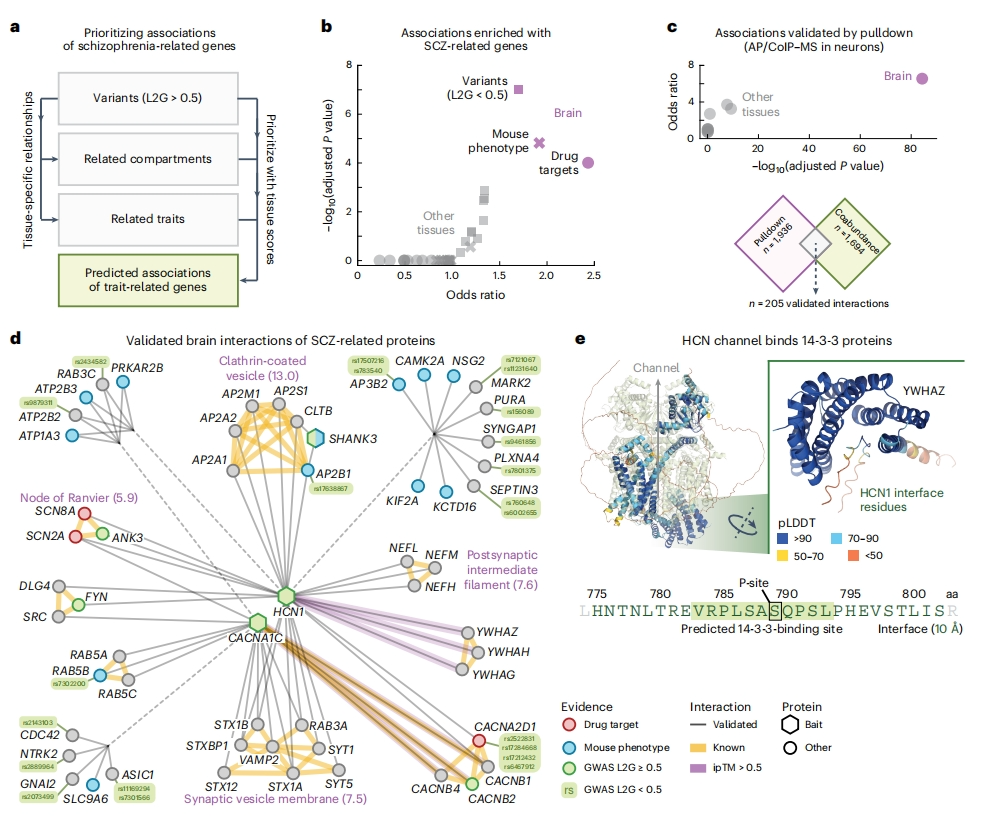
图四 精神分裂症(SCZ)相关蛋白的已验证大脑互作网络[9]
最后,研究者利用这一计算方法来解析突触的相互作用组。制备并纯化了大鼠大脑的突触体,用尺寸排阻色谱法(SEC)将突触体分为75个组分,进行液相色谱LC-MS/MS。共检测到3409个独特的蛋白质,包括已知蛋白质复合物CCT复合物亚基,其特征在各组分之间相关(图五a)。对突触体的分馏曲线进行预处理,计算了各组分之间的蛋白质共丰度,对大鼠突触中4276350对蛋白质的共分馏进行评分。与单独的共分馏研究相比,构建的突触相互作用组量化了1619个蛋白质的1309771对相互作用的概率,并改善了已知相互作用的复现(图五b)。在相互作用组的1619个蛋白质中,24%在SynGO数据库中被注释为突触蛋白,49%被报告为富含小鼠大脑的突触,56%之前已通过小鼠突触体的交联MS(XL-MS)鉴定。与非突触蛋白的关联相比,突触富集蛋白之间的相互作用形成了更可能的关联,特别是与关联性图谱的其他组织相比,大脑中的关联更为明显(图五c)。通过小鼠表型分析(IMPC)或已知药物靶点(ChEMBL)筛选出突触蛋白相关基因的GWAS特征,在13个特征中,有10个是与大脑明显相关的疾病,并选择了727个高置信突触相互作用,使用AlphaFold2预测这727个相互作用的结构。与CORUM或HuMAP中已知相互作用的模型相比,预测模型具有更可靠的相互作用。总共确定了105个中等置信度的相互作用,可视化了大脑疾病特征相关基因之间已验证的突触相互作用网络(图五d)。

图五 共分馏蛋白质的突触相互作用网络[9]
综上所述,该研究分析了11种人体组织7811个蛋白质组样本的蛋白质丰度数据,绘制了一张人类组织特异性的蛋白质关联性图谱,能够以组织特异性的方式对候选致病基因进行优先排序。超过25%的关联性是具有组织特异性的,仅有<7%归因于基因的表达差异。细胞组分是组织特异关联性的关键驱动力。该研究详细展示了一个精神分裂症相关基因的大脑相互作用网络,并通过突触体的共分馏实验、下拉数据和AlphaFold2建模验证了大脑突触蛋白质关联性。该图谱助力更深入地理解不同组织中细胞结构的独特蛋白质互作组,增强对疾病机制的理解,还提出了一种基因优选策略,有助于候选药物靶点的筛选排序。

GTP研发中心拥有自主知识产权的类精子干细胞介导的半克隆技术,可在体外实现类精子干细胞的多位点改造并进行功能元件测试,最后将类精子干细胞通过卵母细胞注射可以一步法获得基因改造小鼠。基于类精子干细胞单倍体打靶高效率的优势,可快速构建定制化小鼠,如基因组安全位点大片段敲入、条件性基因敲除/敲入和人源化基因改造等。多个复杂调控元件可以通过多次体外细胞打靶构建在同一株类精子干细胞中,经细胞水平质控后,一步法获得基因改造小鼠。

如有需要欢迎联系我们,GTP研发中心将竭诚为您设计最佳方案,制定专属于您的基因改造小鼠。
网址:http://www.sibcb.ac.cn/gtp/
地址:上海市岳阳路320号
责任编辑:多一百
新媒体运营:suway
参考文献
1. Drew K, Wallingford JB, Marcotte EM: hu.MAP 2.0: integration of over 15,000 proteomic experiments builds a global compendium of human multiprotein assemblies. Molecular systems biology 2021, 17(5):e10016.
2. Hein MY, Hubner NC, Poser I, Cox J, Nagaraj N, Toyoda Y, Gak IA, Weisswange I, Mansfeld J, Buchholz F et al: A human interactome in three quantitative dimensions organized by stoichiometries and abundances. Cell 2015, 163(3):712-723.
3. Skinnider MA, Scott NE, Prudova A, Kerr CH, Stoynov N, Stacey RG, Chan QWT, Rattray D, Gsponer J, Foster LJ: An atlas of protein-protein interactions across mouse tissues. Cell 2021, 184(15):4073-4089.e4017.
4. Pierson E, Koller D, Battle A, Mostafavi S, Ardlie KG, Getz G, Wright FA, Kellis M, Volpi S, Dermitzakis ET: Sharing and Specificity of Co-expression Networks across 35 Human Tissues. Plos Comput Biol 2015, 11(5):e1004220.
5. Huttlin EL, Bruckner RJ, Navarrete-Perea J, Cannon JR, Baltier K, Gebreab F, Gygi MP, Thornock A, Zarraga G, Tam S et al: Dual proteome-scale networks reveal cell-specific remodeling of the human interactome. Cell 2021, 184(11):3022-3040 e3028.
6. McShane E, Sin C, Zauber H, Wells JN, Donnelly N, Wang X, Hou J, Chen W, Storchova Z, Marsh JA et al: Kinetic Analysis of Protein Stability Reveals Age-Dependent Degradation. Cell 2016, 167(3):803-815.e821.
7. Buljan M, Banaei-Esfahani A, Blattmann P, Meier-Abt F, Shao W, Vitek O, Tang H, Aebersold R: A computational framework for the inference of protein complex remodeling from whole-proteome measurements. Nature methods 2023, 20(10):1523-1529.
8. Roumeliotis TI, Williams SP, Gonçalves E, Alsinet C, Del Castillo Velasco-Herrera M, Aben N, Ghavidel FZ, Michaut M, Schubert M, Price S et al: Genomic Determinants of Protein Abundance Variation in Colorectal Cancer Cells. Cell Rep 2017, 20(9):2201-2214.
9. Laman Trip DS, van Oostrum M, Memon D, Frommelt F, Baptista D, Panneerselvam K, Bradley G, Licata L, Hermjakob H, Orchard S et al: A tissue-specific atlas of protein-protein associations enables prioritization of candidate disease genes. Nature biotechnology 2025.
10. Guardia CM, De Pace R, Mattera R, Bonifacino JS: Neuronal functions of adaptor complexes involved in protein sorting. Curr Opin Neurobiol 2018, 51:103-110.
11. Ashburner M, Ball CA, Blake JA, Botstein D, Butler H, Cherry JM, Davis AP, Dolinski K, Dwight SS, Eppig JT et al: Gene ontology: tool for the unification of biology. The Gene Ontology Consortium. Nature genetics 2000, 25(1):25-29.
12. Orchard S, Ammari M, Aranda B, Breuza L, Briganti L, Broackes-Carter F, Campbell NH, Chavali G, Chen C, del-Toro N et al: The MIntAct project--IntAct as a common curation platform for 11 molecular interaction databases. Nucleic acids research 2014, 42(Database issue):D358-363.
13. Lankford C, Houtman J, Baker SA: Identification of HCN1 as a 14-3-3 client. Plos One 2022, 17(6):e0268335.



