技术分享:DNA介导的转录因子间协同作用拓展了人类基因调控密码
物种的遗传信息都蕴藏在基因组中。转录因子(TF)可通过识别短DNA序列基序来调控基因表达,进而TF的DNA结合特异性组成了基因调控密码的分子基础[1]。在简单的低等生物体中,单个TF结合到特定靶基因上来调控转录[2, 3]。相比之下,具有多种不同器官的高等生物需要在发育过程中确保所有器官都处于正确的位置,还需要在三维时空上整合位置信息[3]。这就要求具备高特异性和复杂信息的组合处理能力,可通过TF之间的协同作用来实现。TF-TF相互作用是确定细胞命运和执行细胞类型特异性转录程序所必需的。因此,破译TF之间协同作用的基因调控密码,能帮助人们更好地解读遗传天书。
已知一些TF家族参与调控胚胎轴形成。例如,同源结构域蛋白家族的成员参与建立胚胎前后(A-P)轴,并沿着前后轴差异化表达。可是,将这种TF家族的差异表达转化为不同发育结果的确切机制尚不清楚。此外,研究表明通过协同作用形成的TF-TF-DNA复合物,可以增加TF特异性识别基序的数量[4, 5]。因为单个TF的识别位点可以以不同的方向和/或间距出现,促使一对TF可以结合更多不同的DNA基序。其中,一些TF以预先形成的多聚体蛋白复合物的形式结合到DNA上,此类复合物通常是二聚体或三聚体,由同一TF家族的成员组成,例如bHLH、bZIP和BTB结构域锌指蛋白[6]。然而,对DNA上的TF复合物分析表明,许多TF也能以DNA依赖或DNA促进的方式跨越蛋白家族的界限而结合在一起,如OCT4-SOX2对[7]。这种DNA促进的相互作用数量预计大大超过单个TF的DNA结合数量,从而将基因调控密码扩展到远远超出简单的蛋白质-蛋白质相互作用所能实现的范围。因此,人类基因调控密码比遗传密码复杂得多,特别是包括有多于1600个TF的相互作用,但TF-TF-DNA的相互作用图谱仍不清楚。
2025年4月,Nature期刊在线报道了一项研究,拓展了人类基因调控密码。使用CAP-SELEX绘制了DNA结合TF之间的生化相互作用,同时识别单个TF的结合偏好、TF-TF相互作用及其互作结合的DNA序列。筛选了超过58000对TF-TF,鉴定出2198对相互作用的TF对,其中1329个优先结合到以不同间距和/或方向排列的基序上。研究还发现了1131个TF-TF复合基序,这些基序与单个TF的基序明显不同。该研究预计筛选鉴定出了所有人类TF-TF基序的18%~47%。新发现的复合基序富集在细胞类型特异性元件,在体内具有活性,倾向于在发育过程中共表达的TF之间形成。此外,参与建立胚胎轴的TF通常与不同的TF相互作用,结合到复合基序,从而解释了具有相似特异性的同一家族TF可以沿着发育轴定义不同的细胞类型[8]。
研究者之前已开发CAP-SELEX,从随机序列库中筛选出同时与两个不同TF结合的特定DNA序列,用于分析DNA存在下的TF-TF相互作用[4]。为了提高CAP-SELEX程序的通量,研究者将其调整为384孔板筛选(图一a)。在大肠杆菌中表达了一组哺乳动物保守的人类TF的拓展DNA结合域(eDBD),这些表达的TF代表了所有主要的TF家族(图一b)。使用N端6×His标记TF1 eDBD(prey),C端SBP标记TF2 eDBD(bait)。共计表达376个His标记的prey TF1和158个SBP标记的bait TF2,将它们组合成共计58754对TF1-TF2。筛选序列包含40 bp随机序列和两端的测序接头序列。将1 μg随机序列库与100 ng TF1-TF2一起孵育,然后连续的标签蛋白亲和纯化TF1-TF2,洗板,PCR扩增结合的DNA并测序。重复三次实验,从富集的序列中检测出TF1-TF2的结合基序。每个384孔板上包含8对已知相互作用的TF1-TF2作为阳性对照(CEBPD-ETV5、CEBPD-ATF4、FOXO1-ETV5、FOXO1-GCM1、TEAD4-ONECT2、TEAD4-COLCK、HES7-TFAP2C和HES7-ETV5)。共计筛选出2198对TF1-TF2 显示出特定的相互作用,包括1329个间距和方向偏好的基序以及1131个复合基序(图一a)。

图一 绘制DNA介导的转录因子间相互作用图谱[8]
为确定单个TF结合位点是否在TF对中显示出间距和方向的偏好性,分析相互作用的TF1-TF2各自基序中包含的特征k-mer。分析表明,大多数TF-TF都是以短间距结合,两个TF特征性8-mer序列的间隔很少超过5 bp(图一c)。对间距的偏好性通常是特定的,同一家族的不同成员与相同或相关TF之间倾向于保持不同的间距(图一c)。对所有类型的相互作用分析表明,TF-TF相互作用通常跨越了TF家族的界限(图一d)。与之前的研究相一致,一些TF家族如TEA更频繁地与其他TF家族相互作用,相比之下,ZNF C2H2与其他TF家族的相互作用则很少(图一d)。
研究者将这些基序与之前发表的CAP-SELEX基序进行比较[4],并绘制相似性图。分析表明,本次研究鉴定出了169个新的复合基序。与间距和/或方向偏好性相似,复合基序也可分为家族、亚家族和旁系同源特定基序。间距和/或方向偏好性主要倾向于家族或亚家族形式,而复合基序更常见的是旁系同源形式(图二a)。例如,大多数前HOX蛋白与TBX21相互作用。然而,HOX蛋白的PITX亚类特异性与FOXA和FOXD相互作用,HOX2的同源物(HOXA2和HOXB2)特异性与PROX1和PROX2相互作用(图二b)。接着,采用MoSBAT评估复合基序和单个TF基序的相似性,发现几乎所有的复合基序都与单个基序明显不同。计算了每个基序对其共识序列的得分,并将其与相应复合基序的最佳匹配共识序列进行比较,表明单个TF对于复合基序共识序列的亲和力相对较低。例如,尽管ELF1-FOXK2的复合基序与单个TF基序肉眼可见的截然不同,但在ChIP-seq实验的DNA结合峰中,ELF1-FOXK2的复合基序出现在各自ChIP-seq的重叠峰序列中,但没出现在非重叠峰中(图二c),表明即使新的复合基序与单个基序不相似,也能在体内被TF-TF对结合。

图二 复合基序的特异性分析[8]
随后,研究者探究具有特定生物或生化功能的TF是否与其他TF表现出不同类型的相互作用。首先分析可以诱导细胞转分化的已知先锋因子[9],确定它们和其他TF之间的几种特定相互作用(图三a)。其中,一些经典先锋因子如FOXA1和SOX11能和许多TF相结合,但其他先锋因子,包括GATA3和GATA4、CEBPs、PAX6和PAX7、以及除SOX11以外的SOXs,对于结合伙伴更具选择性。表明先锋因子在结合伙伴的选择上各有不同,没有表现出特殊的相互作用方式。其次,分析已知参与背腹(D-V)轴神经管排布的发育重要TF,发现大多数特定的细胞类型都有与复合基序结合的特定TF-TF对(图三b)。
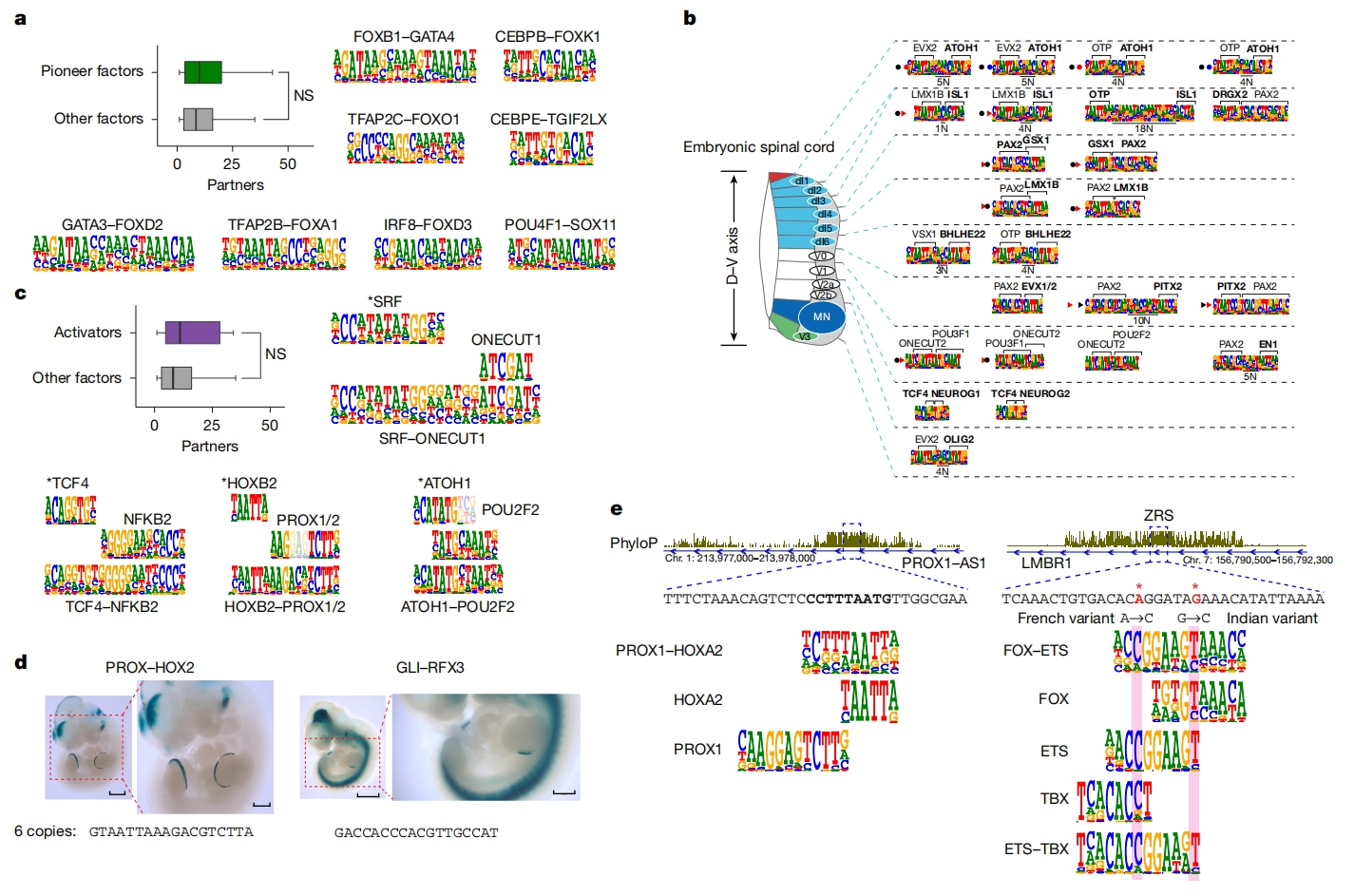
图三 复合基序的生物学作用[8]
研究表明,只有少数人类TF具有很强的转录激活结构域[10]。许多具有强激活结构域的TF,包括TCF4、ATOH1、SRF和HOXB2,都与其他TF特异性相互作用,分别对应结合NFKB2、POU2F2、ONECUT1和PROX1/PROX2(图三c)。该研究仅有16个测试TF具有强激活结构域,然而,与它们相互作用的TF多达171个,提示没有激活结构域的TF通常能与其他TF相互作用,进而募集强转录激活结构域到DNA上。为测试所获得的激活结构域TF的复合基序能否驱动体内基因表达,研究者构建了含有6个拷贝复合基序序列的报告质粒,胞浆注射到受精卵中构建整合在H11位点的转基因增强子报告小鼠。当TF1-TF2体内结合到复合基序上,可激活LacZ报告基因的表达。检测E11.5天的F0胚胎,含有HOXA2-PROX2复合基序的LacZ报告基因在顶端外胚层嵴、前脑、中脑、后脑、颌面部间充质和耳泡中高表达(图三d)。该基序也存在于Prox1本身的保守增强子上,该增强子可抑制淋巴管中的造血(图三e)。此外,GLI3-RFX3复合基序能驱动LacZ报告基因在腹侧中脑、神经管、前脑和肢芽极化活动区(ZPA)表达,与该阶段Shh的表达位置相一致(图三d)。但也有测试的FLI1-GLI3复合基序没能驱动报告基因的表达。
接下来,研究者尝试建立计算预测模型,预测所有TF-TF的协同常数。首先使用AlphaFold v.2.0多聚体函数来预测一些指示性TF-TF复合物。AlphaFold可以预测蛋白质复合物的结构,但不能预测DNA结构。将AlphaFold预测的TF-TF对结构,与实验确定的TF-TF-DNA结构的蛋白质组分进行比较。AlphaFold 2正确预测了几种已知的异二聚体TF-TF-DNA复合物的蛋白质组分结构,如bHLH二聚体MYC-MAX这种预先结合的二聚体(图四a)。然而,AlphaFold 2无法预测DNA介导和DNA依赖性的TF-TF复合物,如MEIS1-DLX3(图四a)。使用最近发布的能预测蛋白质-核酸复合物结构的RoseTTAFold(RoseTTAFold2NA v.0.2)和AlphaFold v.3.0版本,它们在预测TF-TF-DNA复合物的整体几何构造方面表现得更好(图四b)。然而,在大多数情况下,他们仍然无法正确预测TF-TF对与DNA结合时的优先间距和方向(图四b)。在预测异二聚体时,AlphaFold 3成功预测了HOXB13-TEAD4、FOXK1-ELF1、MEIS1-HOXB13和DLX3-MEIS1异二聚物的整体几何构造,RoseTTAFold2NA只预测了HOXB13-TEAD4和FOXK1-ELF1。可是,这两个程序都无法正确预测FOXK1-ELF1、MEIS1-HOXB13和DLX3-MEIS1复合物之间的极小接触,这有助于复合基序的形成(图四c和d),体现了该研究的必要性。

图四 结构分析显示极小的TF-TF接触面实现了高水平的协同作用[8]
两个TF在DNA上相互作用的生化证据并不意味着这种相互作用具有重要生理意义。由于存在超过130万种可能的TF相互作用,此预计的相互作用数量已大大超过生物功能所需数量。为了评估所获得的TF-TF对在人类发育中的作用,研究者将TF配对矩阵与人类细胞图谱的共表达矩阵进行比较(https://www.humancellatlas.org/),结果显示共表达的TF形成复合基序的频率明显高于随机配对(图五a)。例如,许多参与肢体发育的共表达TF形成了特定的复合基序(图五b)。进一步分析TF-TF对基序在细胞类型特异性顺式调控元件(CRE)中的富集情况。分析来自顺式调控图谱(CATlas)的细胞类型特异性开放染色质区域中富集的基序,也在体内观察到了间距偏好性。TF-TF对的基序通常富集在细胞类型特异性候选CRE集,简称为cCRE集。347个代表性复合基序中有211个,以及112个代表性间隔基序中有74个至少富集在一个cCRE集。具有特定间距的基序也富集在特定的cCRE集,如基序间隔4 bp的bHLH-同源结构域TF对高度富集在基质细胞和成纤维细胞的cCRE集,而基序间隔3、5和6 bp分别富集在胰腺β细胞、星形胶质细胞和谷氨酸能神经元的cCRE集(图五c)。基于逻辑回归分析,复合基序也可用于预测哪些元件对特定细胞类型具有特异性(图五d),表明获得的TF-TF对基序具有生物学相关性,并且通常富集在细胞类型特异性的调控元件里。

图五 共表达TF间形成的复合基序[8]
生理意义上,在基因表达的发育调控过程中,使用复合基序而不是单个低亲和力基序有如下四个优点:(1)通过两个TF的共表达可以整合位置信息;(2)单个TF对复合基序的低亲和力允许其具有双重功能:TF可以作为一个结合伙伴,发挥独立于自身基序的其他发育功能;(3)高亲和的复合基序可在TF低蛋白浓度下提高基因表达的特异性;(4)复合基序可显著增加两种TF的协同系数,使得基因调控能够对TF的浓度变化做出非常敏锐的反应(图五e)。
综上所述,该研究借助CAP-SELEX,对58000多种人类转录因子组合能否协同结合DNA进行了系统性评估,鉴定出2198对(3.7%)协同作用的转录因子,并解析了TF-TF协同识别的DNA基序。该研究提供了全面的TF-TF-DNA协同作用数据库,拓展了人类基因调控密码,为深入探索转录因子的各种生物学功能提供了重要的数据支撑和理论依据,也为发育异常和癌症等各类疾病的个性化治疗提供了一种新的思路。

GTP研发中心拥有自主知识产权的“人造精子细胞”介导的半克隆技术,可在体外实现“人造精子细胞”的多位点改造并进行功能元件测试,最后将“人造精子细胞”通过卵母细胞注射可以一步法获得基因改造小鼠。基于“人造精子细胞”单倍体打靶高效率的优势,可快速构建定制化小鼠,如基因组安全位点大片段敲入、条件性基因敲除/敲入和人源化基因改造等。多个复杂调控元件可以通过多次体外细胞打靶构建在同一株“人造精子细胞”中,经细胞水平质控后,一步法获得基因改造小鼠。

如有需要欢迎联系我们,GTP研发中心将竭诚为您设计最佳方案,制定专属于您的基因改造小鼠。
网址:http://www.sibcb.ac.cn/gtp/
地址:上海市岳阳路320号
责任编辑:多一百
新媒体运营:suway
参考文献
1. Kim S, Wysocka J: Deciphering the multi-scale, quantitative cis-regulatory code. Molecular cell 2023, 83(3):373-392.
2. Lambert SA, Jolma A, Campitelli LF, Das PK, Yin Y, Albu M, Chen X, Taipale J, Hughes TR, Weirauch MT: The Human Transcription Factors. Cell 2018, 172(4):650-665.
3. Wunderlich Z, Mirny LA: Different gene regulation strategies revealed by analysis of binding motifs. Trends in genetics : TIG 2009, 25(10):434-440.
4. Jolma A, Yin Y, Nitta KR, Dave K, Popov A, Taipale M, Enge M, Kivioja T, Morgunova E, Taipale J: DNA-dependent formation of transcription factor pairs alters their binding specificity. Nature 2015, 527(7578):384-388.
5. Wolberger C: How structural biology transformed studies of transcription regulation. The Journal of biological chemistry 2021, 296:100741.
6. Reinke AW, Baek J, Ashenberg O, Keating AE: Networks of bZIP protein-protein interactions diversified over a billion years of evolution. Science 2013, 340(6133):730-734.
7. Morgunova E, Taipale J: Structural perspective of cooperative transcription factor binding. Current opinion in structural biology 2017, 47:1-8.
8. Xie Z, Sokolov I, Osmala M, Yue X, Bower G, Pett JP, Chen Y, Wang K, Cavga AD, Popov A et al: DNA-guided transcription factor interactions extend human gene regulatory code. Nature 2025.
9. Zaret KS: Pioneer Transcription Factors Initiating Gene Network Changes. Annual review of genetics 2020, 54:367-385.
10. Tycko J, DelRosso N, Hess GT, Aradhana, Banerjee A, Mukund A, Van MV, Ego BK, Yao D, Spees K et al: High-Throughput Discovery and Characterization of Human Transcriptional Effectors. Cell 2020, 183(7):2020-2035.e2016.



