技术分享:RBM43负调控PGC1α的翻译以及PGC1α-STING信号轴
肥胖与系统性炎症相关,这种炎症会损害线粒体的功能。肥胖引起的全身性炎症特征是脂肪组织的免疫细胞增加,特别是促炎巨噬细胞和T细胞,它们可以通过释放细胞因子改变脂肪细胞的线粒体功能[1]。例如,肿瘤坏死因子α(TNF-α)、白细胞介素-1β(IL-1β)和干扰素(IFN)都与抑制线粒体功能以及产热相关[2, 3]。脂肪在化学能的转化和储存中发挥重要作用,会对所有疾病都产生影响[4]。人类和小鼠的肥胖都伴随着脂肪线粒体含量和氧化代谢的减少[5]。这促使人们研究线粒体对脂肪功能的作用,包括在脂质代谢、胰岛素信号传导和能量消耗中的贡献。与储存化学能的白色脂肪细胞相反,产热脂肪细胞含有大量线粒体,这些线粒体以极快的速度氧化燃料,从而驱动化学循环,将化学能转化为热量。产热脂肪是一种在刺激时由白色脂肪产生的独特细胞类型,包括棕色和米色脂肪细胞。它们的活性对于哺乳动物的体温防御至关重要,能带来代谢益处,包括增加能量消耗、改善葡萄糖和脂质稳态以及提高胰岛素敏感性。
过氧化物酶体增殖物激活受体γ共激活因子1α(PGC1α)可调节棕色脂肪中产热基因的表达,是大多数组织中线粒体生物发生和氧化代谢的主要调节因子[6]。PGC1α协调线粒体生物发生和产热相关无效循环的基因表达。因此,PGC1α是脂肪氧化代谢的核心,其遗传缺陷会减少产热并导致胰岛素抵抗[7]。尽管β肾上腺素能信号传导可诱导其转录,但最近的研究强调Ppargc1a mRNA自身是一个重要的调节节点[8, 9]。例如,发现了一个上游开放阅读框(uORF)可调控Ppargc1a翻译,进而调控多个组织中的氧化代谢[10]。然而,是何种因子调控Ppargc1a翻译及下游的氧化代谢尚未可知。
2025年3月,Cell Metabolism期刊报道了一项研究,发现线粒体生物发生的关键调控因子PGC1α的mRNA翻译受到RNA结合蛋白RBM43的负调控。炎症信号诱导RBM43,抑制PGC1α翻译以限制脂肪细胞线粒体生物发生。选择性破坏小鼠脂肪细胞的Rbm43可提高PGC1α翻译和氧化代谢,改善肥胖诱导的葡萄糖耐受不良,减少脂肪炎症,并抑制脂肪细胞中先天免疫传感器cGAS-STING的激活。该研究定义了一种RBM43-PGC1α的翻译调节轴,将炎症信号与细胞能量代谢联系起来,为代谢性疾病的发病机制提供了新的研究方向[11]。
首先,研究者鉴定氧化代谢的转录后调控因子。分析公开数据库,寻找与脂肪细胞产热相关的RNA结合蛋白,获得25个候选基因。进而,在原代白色脂肪细胞中使用靶向单个候选基因的siRNA进行敲降实验,寻找对PGC1α 蛋白与mRNA的比率造成影响的基因,最终筛选到Rbm43。验证实验发现,靶向Rbm43的两个不同siRNA同样增加了PGC1α 蛋白与mRNA的比率(图一A和B)。然而,PGC1α蛋白的半衰期没有发生变化(图一C)。在含有Ppargc1a 5′ UTR序列的萤光素酶报告系统的HEK293细胞中,过表达RBM43降低了Ppargc1a 5′UTR-nanoLuc mRNA的翻译(图一D),而该序列不包括Ppargc1a 上游开放阅读框(uORF)的起始密码子,表明RBM43不是通过控制核糖体与uORF的结合起作用的。随后,在脂肪细胞中敲降Rbm43,诱导了参与脂肪酸氧化、产热和线粒体功能的基因表达增加,但对脂肪生成没有明显影响,脂肪生成转录调节因子的表达变化也很小(图一E和F)。这些变化与线粒体总呼吸和非耦合呼吸的增加有关,如耗氧量的增加(图一G)。以上数据表明,筛选到的RNA结合蛋白RBM43是Ppargc1a和脂肪细胞氧化代谢的负调节因子。
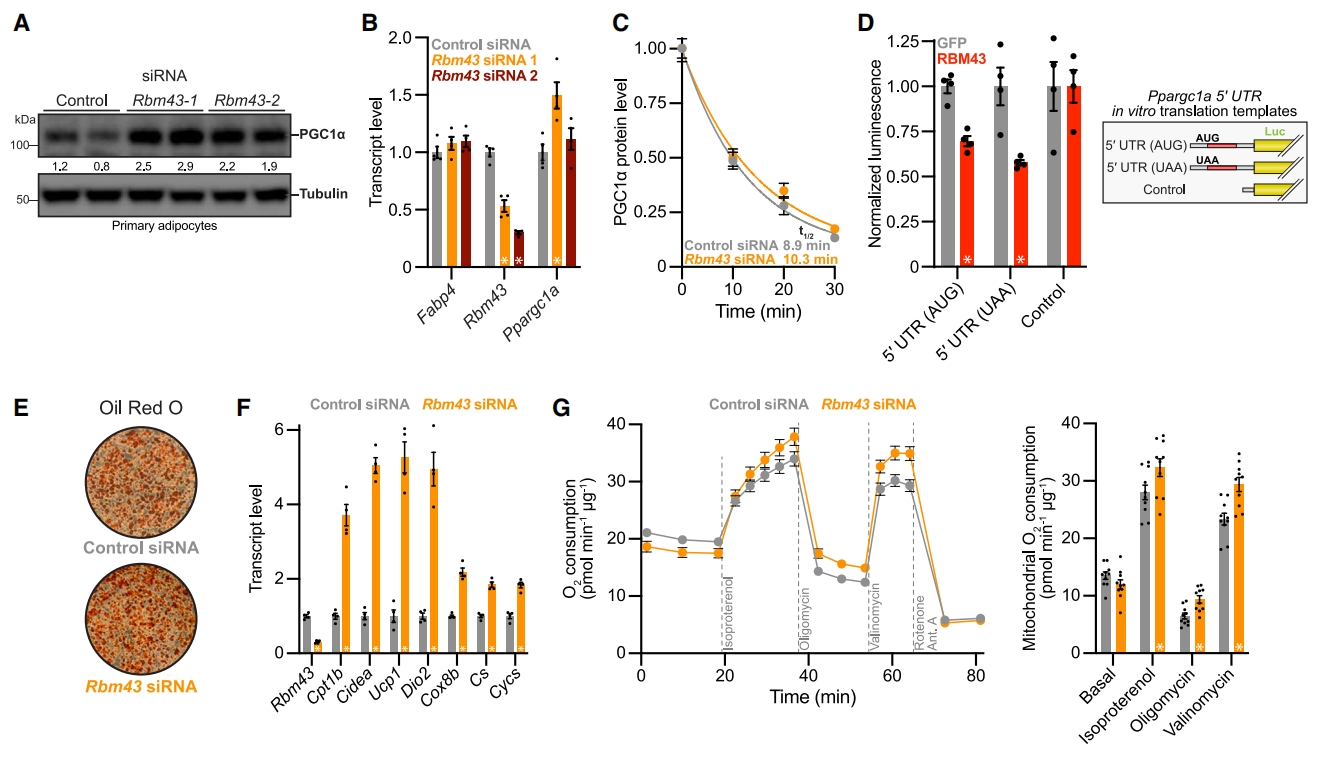
图一 敲降Rbm43可提高原代脂肪细胞的PGC1α蛋白和线粒体呼吸[11]
接着,研究RBM43在小鼠组织的表达谱式。在22°C饲养小鼠的所有检查组织中都检测到了Rbm43,其中白色脂肪组织(WAT)包括腹股沟WAT(iWAT)、内脏脂肪组织(VAT)和脾脏中的表达水平最高;而棕色脂肪组织(BAT)最低,相比白色脂肪低80%(图二A)。使用已发表的翻译核糖体亲和纯化(TRAP)数据评估脂肪细胞中Rbm43的表达情况,证实其选择性表达在白色脂肪,而不是产热脂肪(图二B)。将小鼠低温(4°C)饲养以诱导脂肪产热后,VAT、iWAT和BAT中Rbm43表达都显著降低(图二C和D)。由于肾上腺素能信号驱动产热,研究者评估其对Rbm43的影响。在肾上腺素能激动剂和其他增加cAMP的药物作用下,小鼠原代脂肪细胞的Rbm43表达都显著降低(图二E)。而在用IFN-α、IFN-γ、TNF-α或IL-1β处理小鼠原代脂肪细胞后,都显著增加了Rbm43的表达,其中IFN-α的诱导效果最大(图二F)。IFN反应不仅可以由IFN受体启动,还可以由先天免疫系统的传感器启动,如TLR、RIG-I和cGAS-STING[12]。用脂多糖(LPS,TLR4配体)、聚肌胞苷酸(poly-I:C,TLR3的配体、RNA受体RIG-I与MDA5的激活剂)处理原代脂肪细胞,都显著增加了Rbm43的表达;STING激活剂diABZI或cGAMP具有更大的作用,使Rbm43 mRNA增加了15倍(图二G)。同样地,在人脂肪细胞中也能重复上述实验(图二H-J)。以上结果表明,Rbm43受肾上腺素能信号的负调控,但受炎性细胞因子和先天免疫受体的正调控。
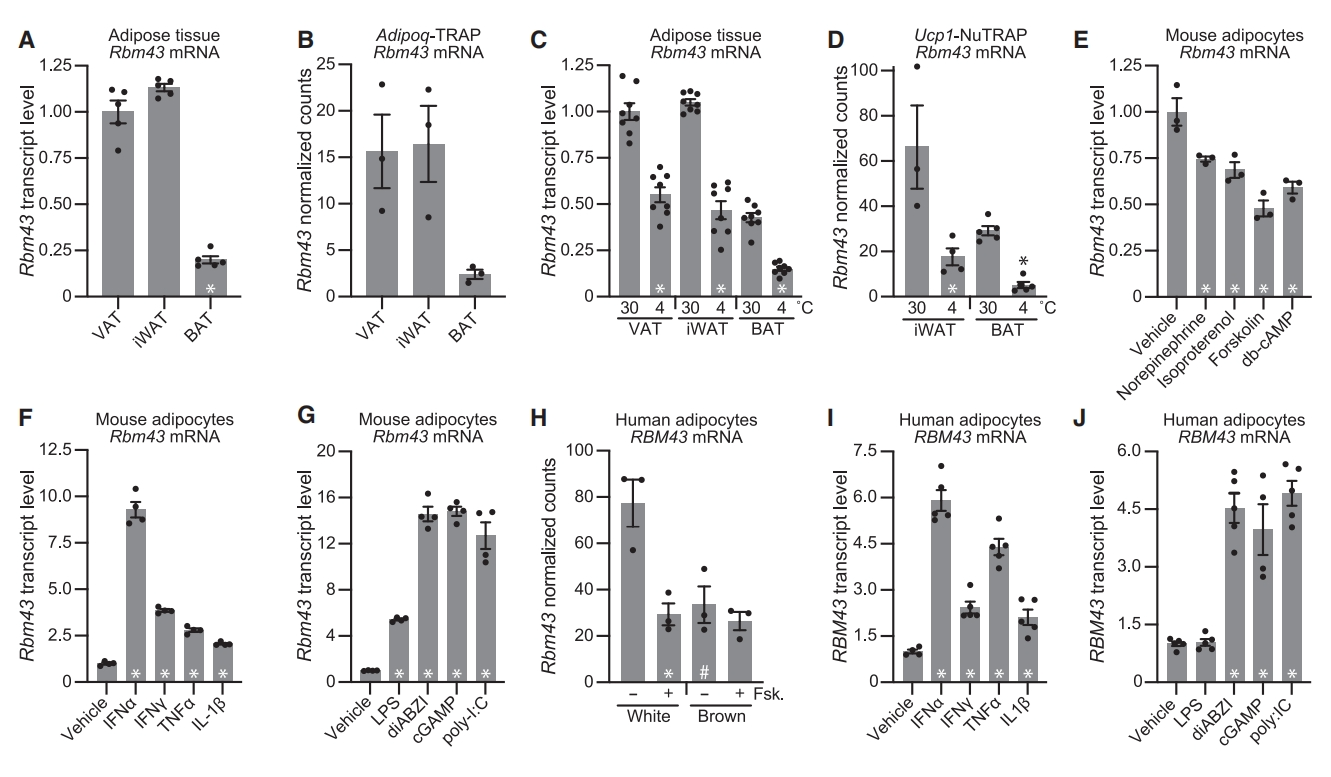
图二 Rbm43受肾上腺素能信号和炎性细胞因子的调控[11]
接下来,研究者探究受RBM43调控的全局基因表达及其对PGC1α的影响。使用表达Cre或GFP(对照)的腺病毒转导原代Ppargc1afl/fl脂肪细胞,同时敲降Rbm43。仅敲降Rbm43时,上调了693个基因的表达,其中氧化磷酸化、脂肪生成和脂肪酸代谢是三个代表性最高的基因集(图三A和B),并且71%的基因与PGC1α的活性升高相关,包含有PGC1α的直接和间接靶标(图三B-D)。仅敲降Rbm43时,下调了253个基因的表达,其中上皮间质转化(EMT)、未折叠蛋白反应和炎症反应是最三个代表性的基因集,仅有26%的基因与PGC1α相关(图三E和F)。以上结果表明,RBM43一方面通过抑制PGC1α来抑制氧化代谢相关基因,另一方面激活细胞应激和炎症相关基因,后者小部分同样依赖于PGC1α。

图三 RBM43通过PGC1α调控氧化代谢基因表达[11]
研究者进一步验证RBM43对PGC1α以及氧化代谢的影响。构建了脂肪细胞特异性敲除Rbm43的小鼠(Rbm43-AKO),小鼠出生符合孟德尔比率,对棕色和白色脂肪的重量没有差异。在Rbm43-AKO 的iWAT中,PGC1α蛋白水平的增加与其mRNA不成比例(图四A和B),再次表明PGC1α的转录后调控发生了改变。用放线菌酮处理分离的脂肪细胞保留核糖体-mRNA相互作用,然后裂解并进行蔗糖密度梯度离心[13],评估Ppargc1a的翻译。与同窝Rbm43fl/fl小鼠相比,Rbm43-AKO提取物中Ppargc1a mRNA的分布向更高密度的组分偏移,而作为对照的Retn mRNA的分布没有显著改变(图四C),表明Ppargc1a的翻译增加。研究者分离了iWAT脂肪细胞,进行定量蛋白质组学分析,发现RBM43缺失在蛋白水平上导致多个通路上调,其中氧化磷酸化、脂肪生成和脂肪酸代谢的上调最多,而下调通路包括EMT和炎症信号传导(图四D)。这些变化同样与PGC1α活性高度相关,包括参与氧化磷酸化和脂肪产热的蛋白(图四E-I)。免疫印迹证实RBM43缺失后,解偶联蛋白1(>10倍)和单个氧化磷酸化复合物组分(1.2至2倍)的升高(图四G和H)。海马生物分析仪测量显示,Rbm43-AKO小鼠的iWAT,而非VAT,具有更高的单位质量呼吸能力(图四J)。用β3-肾上腺素能受体激动剂CL-316243处理小鼠后,置于代谢笼测量能量消耗。结果显示,尽管注射PBS后,在Rbm43-AKO小鼠和同窝Rbm43fl/fl小鼠中都引起了等效应激反应,但CL-316243处理后在Rbm43-AKO小鼠中显著诱导了更高的能量消耗(图四K)。以上结果表明,RBM43抑制了白色脂肪中的线粒体生物合成和氧化代谢。
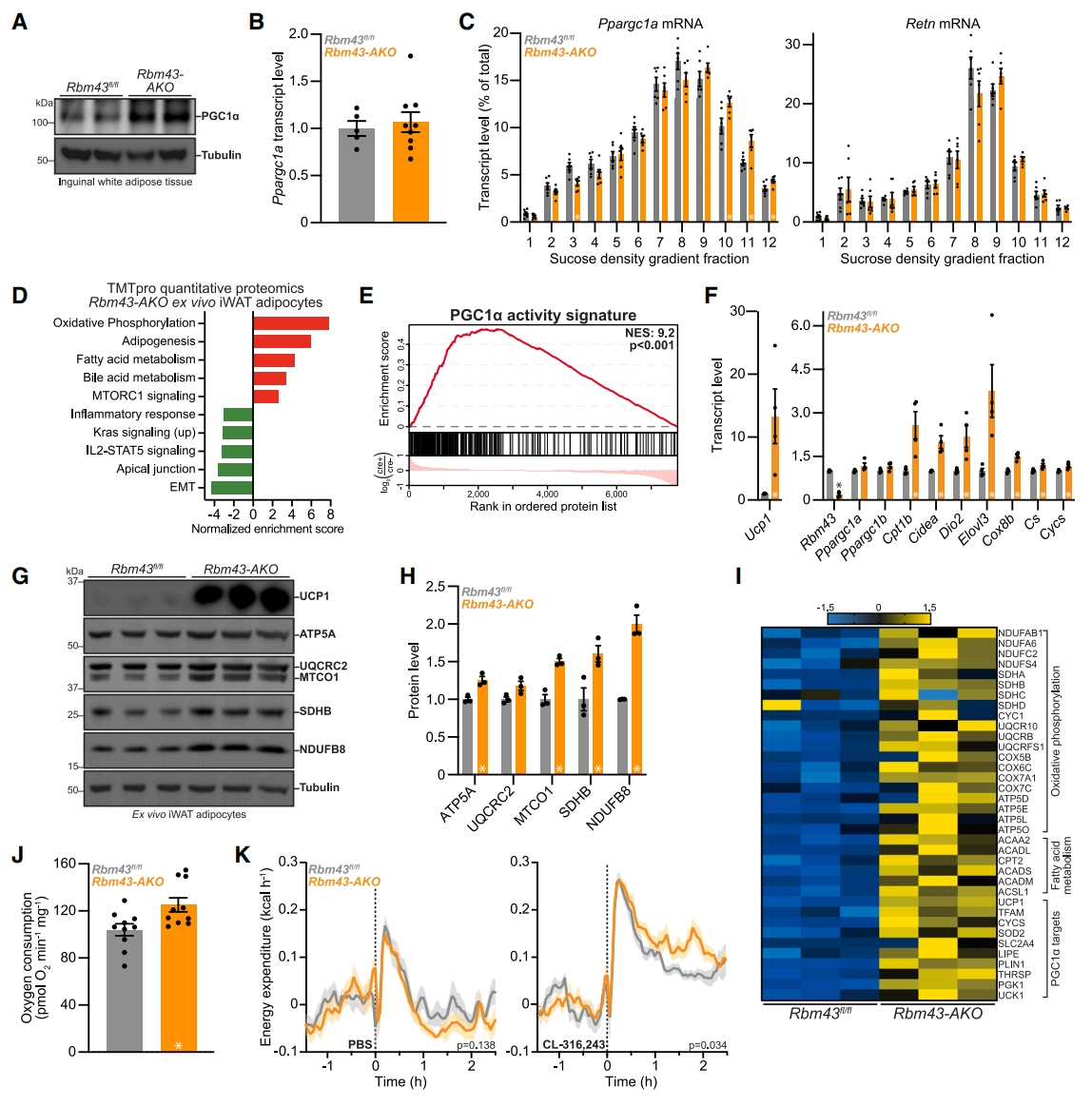
图四 RBM43调控白色脂肪的PGC1α翻译、线粒体生物合成和氧化代谢[11]
研究者推测Rbm43-AKO小鼠可能由于脂肪产热能力的提高,而抵抗高脂饮食(HFD)引起的体重增加。于是,比较正常饮食和高脂饮食喂养的两组小鼠,发现尽管Rbm43-AKO小鼠的体重和体脂比与同窝Rbm43fl/fl对照组没有区别(图五A和B、D和E),但是,Rbm43-AKO小鼠在高脂喂养后明显改善了葡萄糖稳态(图五C和F),表明Rbm43-AKO小鼠不易受到肥胖造成的代谢影响。
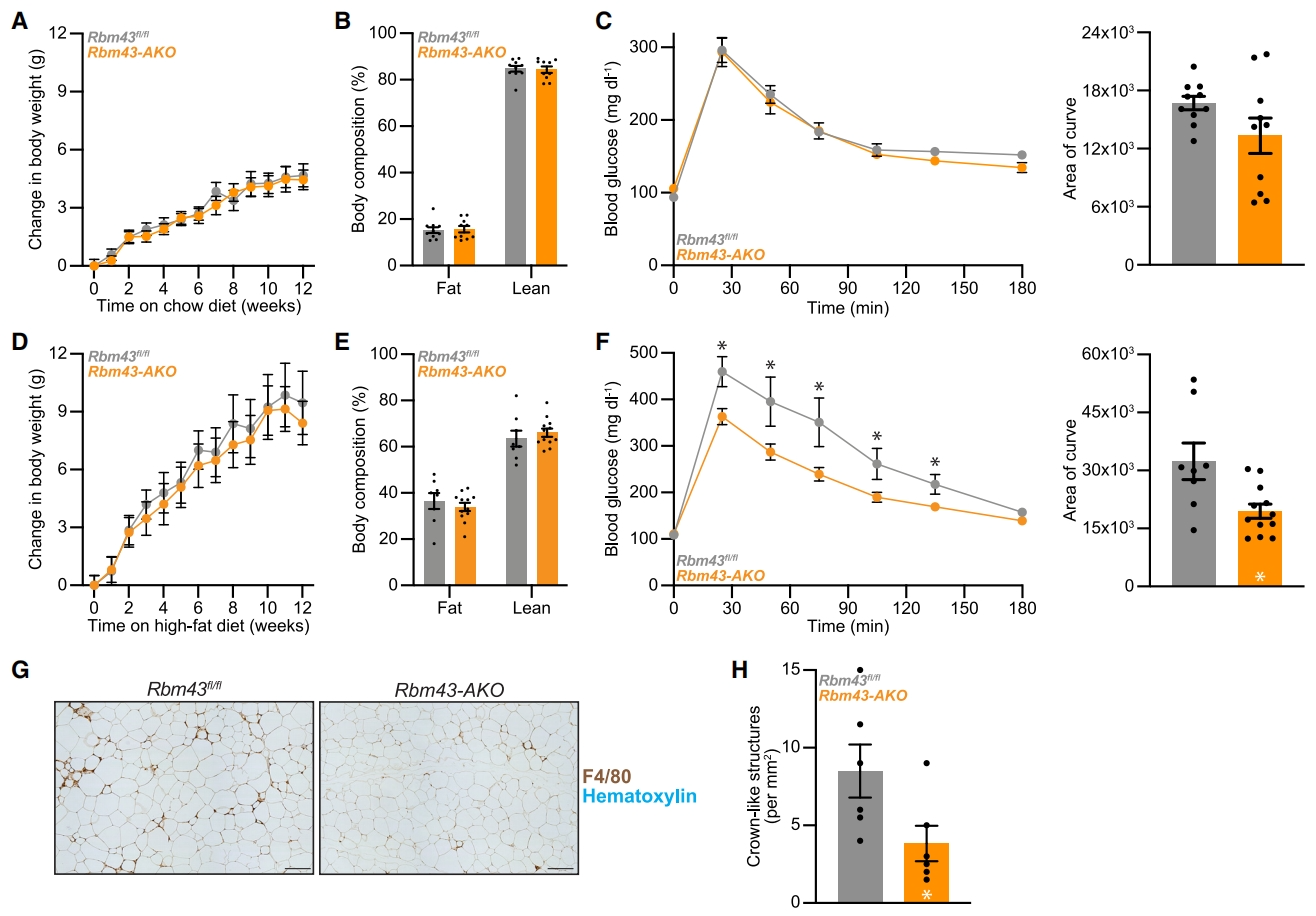
图五 高脂饮食小鼠的脂肪细胞缺失RBM43可独立于体重改善葡萄糖稳态[11]
Rbm43-AKO小鼠在高脂饮食喂养后,VAT巨噬细胞的积聚显著减少(图五G和H)。VAT在小鼠体内的产热能力相对较低,但其细胞因子分泌增加且与人类代谢疾病高度相关,可能在肥胖相关的炎症中发挥作用。于是,研究者探究RBM43对WAT产热功能之外的影响。分离高脂饮食喂养的Rbm43-AKO小鼠VAT脂肪细胞,同样显示PGC1α蛋白水平升高,而mRNA没有变化(图六A和B)。定量蛋白质组富集分析显示,氧化磷酸化是Rbm43-AKO VAT脂肪细胞中上调最显著的基因集(图六C),下调通路包括NF-κB和IFN刺激的炎症信号传导以及EMT,这些通路都与肥胖有关(图六C和D),脂肪细胞响应高脂饮食的全局特征也显著下调(图六E),表明Rbm43的缺失广泛阻止了促炎和细胞外基质重塑程序的激活。Rbm43-AKO VAT脂肪细胞中STING激活的表达特征也显著降低(图六F),表明先天免疫传感器系统cGAS-STING激活减少,该系统检测细胞质DNA,包括病原体来源的DNA以及异常定位的线粒体DNA(mtDNA)[14]。高脂饮食后,Rbm43-AKO小鼠的VAT细胞质mtDNA大幅减少,尽管细胞内总mtDNA水平增加(图六G和H);cGAS产物2′3′-cGAMP也保持在较低水平(图六I),cGAS和STING蛋白以及下游信号TBK1和IRF3的磷酸化也显著减少(图六I和J)。以上数据表明,在肥胖的情况下,RBM43缺失可保护脂肪细胞免受细胞质mtDNA积累和STING激活的影响。

图六 RBM43促进肥胖小鼠白色脂肪细胞的促炎基因表达和STING激活[11]
最后,研究者探究RBM43激活STING是否与PGC1α有关。构建了脂肪细胞选择性敲除Ppargc1a小鼠(Ppargc1a-AKO),高脂饮食喂养后分离VAT脂肪细胞。与同窝Ppargc1afl/fl对照小鼠相比,Ppargc1a-AKO脂肪细胞的细胞质mtDNA增加了2.2至2.5倍,总mtDNA变化不大(图七A和B);cGAS产物2′3′-cGAMP水平增加(图七C);STING下游信号蛋白和基因表达的增加(图七D和E)。研究者使用慢病毒CRISPR-Cas9在原代iWAT脂肪细胞中靶向Ppargc1a,使PGC1α水平降低了90%以上。在这些细胞中敲降Rbm43对氧化代谢基因表达几乎没有显著影响,证实了RBM43对PGC1α的依赖性。尽管细胞总mtDNA减少了33%-39%,但破坏Ppargc1a增加了细胞质mtDNA(图七F和G);同样地,2′3′-cGAMP、STING通路组分蛋白磷酸化以及IFN刺激基因表达都显著增加(图七H-J)。一致地,Ppargc1a−/−脂肪细胞的全转录组分析,确定了IFN反应上调以及STING活性特征的激活(图七K和L)。重要的是,使用STING小分子拮抗剂H-151完全阻止了PGC1α缺失引起的IFN刺激基因的上调(图七J)。相比之下,H-151不影响RBM43介导的PGC1α抑制,证实STING活性不是RBM43功能所必需的。这些结果确定了PGC1α在保护细胞质mtDNA积累和cGAS-STING激活方面的作用,提示RBM43抑制PGC1α导致代谢性疾病的潜在途径。
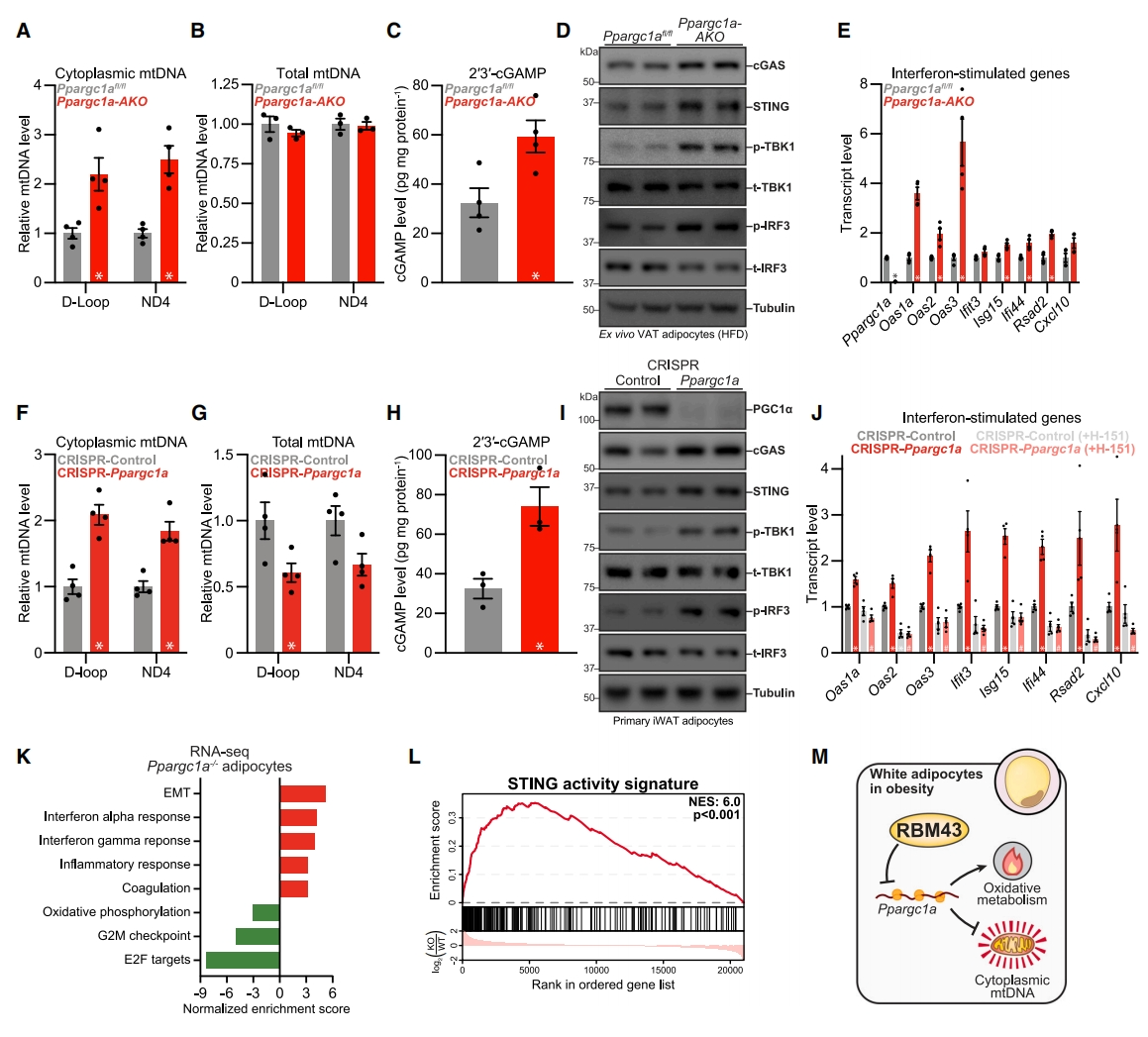
图七 PGC1α防止细胞质mtDNA积累和STING激活[11]
综上所述,该研究证明了RNA结合蛋白RBM43是细胞炎症因子信号传导和氧化代谢之间的潜在调控因子。RBM43由促炎信号诱导,抑制了线粒体生物合成的关键调节因子PGC1α的翻译,进而削弱了脂肪氧化代谢。而PGC1α促进了线粒体生物发生和氧化代谢能力,同时防止细胞质mtDNA的积累以及由此引发的STING激活。小鼠在高脂饮食诱导肥胖后,RBM43激活了cGAS-STING,介导了脂肪细胞炎症反应以及葡萄糖耐受不良,该过程大部分依赖于抑制了PGC1α翻译(图七M)。该研究揭示了一个RBM43-PGC1α-STING轴,提示炎症信号可支配细胞能量代谢,为炎症和线粒体在代谢性疾病机制中的作用提供了新的见解。

GTP研发中心拥有自主知识产权的“人造精子细胞”介导的半克隆技术,可在体外实现“人造精子细胞”的多位点改造并进行功能元件测试,最后将“人造精子细胞”通过卵母细胞注射可以一步法获得基因改造小鼠。基于“人造精子细胞”单倍体打靶高效率的优势,可快速构建定制化小鼠,如基因组安全位点大片段敲入、条件性基因敲除/敲入和人源化基因改造等。多个复杂调控元件可以通过多次体外细胞打靶构建在同一株“人造精子细胞”中,经细胞水平质控后,一步法获得基因改造小鼠。

如有需要欢迎联系我们,GTP研发中心将竭诚为您设计最佳方案,制定专属于您的基因改造小鼠。
网址:http://www.sibcb.ac.cn/gtp/
地址:上海市岳阳路320号
责任编辑:多一百
新媒体运营:suway
参考文献
1. Villarroya F, Cereijo R, Villarroya J, Gavaldà-Navarro A, Giralt M: Toward an Understanding of How Immune Cells Control Brown and Beige Adipobiology. Cell metabolism 2018, 27(5):954-961.
2. Chen XH, Zhao YP, Xue M, Ji CB, Gao CL, Zhu JG, Qin DN, Kou CZ, Qin XH, Tong ML et al: TNF-alpha induces mitochondrial dysfunction in 3T3-L1 adipocytes. Molecular and cellular endocrinology 2010, 328(1-2):63-69.
3. Nisoli E, Briscini L, Giordano A, Tonello C, Wiesbrock SM, Uysal KT, Cinti S, Carruba MO, Hotamisligil GS: Tumor necrosis factor alpha mediates apoptosis of brown adipocytes and defective brown adipocyte function in obesity. Proc Natl Acad Sci U S A 2000, 97(14):8033-8038.
4. Sakers A, De Siqueira MK, Seale P, Villanueva CJ: Adipose-tissue plasticity in health and disease. Cell 2022, 185(3):419-446.
5. Heinonen S, Buzkova J, Muniandy M, Kaksonen R, Ollikainen M, Ismail K, Hakkarainen A, Lundbom J, Lundbom N, Vuolteenaho K et al: Impaired Mitochondrial Biogenesis in Adipose Tissue in Acquired Obesity. Diabetes 2015, 64(9):3135-3145.
6. Puigserver P, Wu Z, Park CW, Graves R, Wright M, Spiegelman BM: A cold-inducible coactivator of nuclear receptors linked to adaptive thermogenesis. Cell 1998, 92(6):829-839.
7. Kleiner S, Mepani RJ, Laznik D, Ye L, Jurczak MJ, Jornayvaz FR, Estall JL, Chatterjee Bhowmick D, Shulman GI, Spiegelman BM: Development of insulin resistance in mice lacking PGC-1α in adipose tissues. Proc Natl Acad Sci U S A 2012, 109(24):9635-9640.
8. Lu H, Ye Z, Zhai Y, Wang L, Liu Y, Wang J, Zhang W, Luo W, Lu Z, Chen J: QKI regulates adipose tissue metabolism by acting as a brake on thermogenesis and promoting obesity. EMBO reports 2020, 21(1):e47929.
9. Fisher FM, Kleiner S, Douris N, Fox EC, Mepani RJ, Verdeguer F, Wu J, Kharitonenkov A, Flier JS, Maratos-Flier E et al: FGF21 regulates PGC-1α and browning of white adipose tissues in adaptive thermogenesis. Genes & development 2012, 26(3):271-281.
10. Dumesic PA, Egan DF, Gut P, Tran MT, Parisi A, Chatterjee N, Jedrychowski M, Paschini M, Kazak L, Wilensky SE et al: An Evolutionarily Conserved uORF Regulates PGC1α and Oxidative Metabolism in Mice, Flies, and Bluefin Tuna. Cell metabolism 2019, 30(1):190-200.e196.
11. Dumesic PA, Wilensky SE, Bose S, Van Vranken JG, Gygi SP, Spiegelman BM: RBM43 controls PGC1α translation and a PGC1α-STING signaling axis. Cell metabolism 2025, 37(3):742-757.e748.
12. Marchi S, Guilbaud E, Tait SWG, Yamazaki T, Galluzzi L: Mitochondrial control of inflammation. Nature reviews Immunology 2023, 23(3):159-173.
13. Pringle ES, McCormick C, Cheng Z: Polysome Profiling Analysis of mRNA and Associated Proteins Engaged in Translation. Current protocols in molecular biology 2019, 125(1):e79.
14. Hopfner KP, Hornung V: Molecular mechanisms and cellular functions of cGAS-STING signalling. Nature reviews Molecular cell biology 2020, 21(9):501-521.



