技术分享:高尔基体存在尺寸过滤器调控顶部膜蛋白的极性定位
膜蛋白在细胞生命活动中发挥着重要功能。极性细胞如上皮细胞,存在着膜蛋白的选择性定向分布,表现为顶部和基侧部膜蛋白的极性排序。这种差异的膜蛋白极性排序主要发生在高尔基体,确保了细胞不同区域的特定功能,而极性排序出错会引发多种人类疾病[1]。基侧部膜蛋白序列中包含了短的“邮政编码”可提供定位分布信息[2]。然而,顶部膜蛋白的分选机制仍不完全清楚。在一些特定情况下,部分顶部膜蛋白通过脂质相分离、N-糖基化或pH依赖性蛋白质-聚糖相互作用以及通过转胞吞作用的间接递送进行聚集,但这些机制并非广谱适用于所有的顶部膜蛋白[3]。
许多顶部膜蛋白具有一个有趣的特征:它们的胞质结构域很短或甚至没有。一些顶部膜蛋白是由糖基磷脂酰肌醇(GPI)连接的,并不会穿过细胞膜到达细胞质[4]。相比之下,基侧部膜蛋白通常具有大的胞质结构域,并且经常与其他组分形成复合物,如受体酪氨酸激酶和钙粘蛋白[5]。顶部膜蛋白与基侧部膜蛋白之间这种结构不对称的原因,及其是否与差异分选有关尚不清楚。
2024年10月,Nature Cell Biology期刊发表一篇研究,揭示了顶部膜蛋白在上皮细胞中的极性建立机制。研究者将Crb3、Ace2和Muc1作为小胞质结构域的代表性顶部膜蛋白,使用合成生物学方法确认高尔基体上存在一个“尺寸过滤器”调节顶部膜蛋白的分选定位,且具有保守性。高尔基体的这种“尺寸过滤器”优先促进了具有小胞质结构域蛋白质的顶部运输,增加胞质结构域的大小会导致蛋白质在基侧部皮层的部分错误定位,并显著延缓蛋白质离开高尔基体的时间。此外,Pals1在高尔基体上与Crb3的解离是Crb3顺向运输和递送到顶部细胞膜的重要调控步骤[6]。
首先,研究者调查PolarProtDB数据库中的所有上皮膜蛋白[7]。按细胞质结构域长度进行注释,对于多次跨膜蛋白,选择最长的胞质片段。与基侧部膜蛋白相比,顶部膜蛋白的胞质结构域明显较小(图一a),而总长度的差异要小得多(图一c)。筛选的膜蛋白以大量的多次跨膜溶质载体为主,这些膜蛋白大致均匀地分布在顶部和基侧部区域。这些溶质载体同样存在顶部和基侧部定位的胞质结构域的大小差异,但总长度也几乎没有差异(图一b和d)。因此,研究者推测高尔基体上存在着“尺寸过滤器”优先使这些短胞质结构域的膜蛋白分配到顶部细胞膜中。
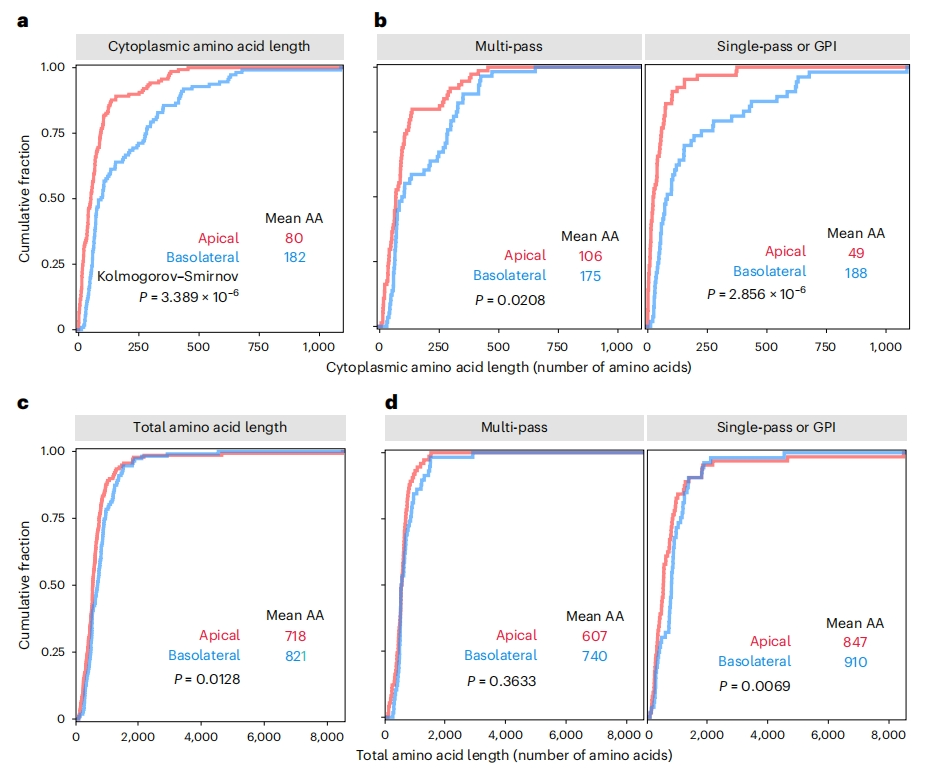
图一 顶部和基侧部膜蛋白的胞质结构域尺寸存在显著差异[6]
接下来,研究者使用RUSH系统探究顶部膜蛋白的顺向运输。将顶部跨膜极性蛋白Crb3[8]与SBP融合,链霉亲和素与内质网(ER)定位的Lys-Asp-Glu-Leu(KDEL)序列融合(StrKDEL),基于SBP与链霉亲和素相互作用使得Crb3蛋白保留在ER上(图二a)。当添加生物素后,SBP-StrKDEL相互作用被破坏,使得SBP融合蛋白从ER同步释放,恢复其正常进程(图二a和b)。于是,研究者在极性Eph4乳腺上皮细胞中过表达SBP-Halo-Crb3和StrKDEL,发现SBP-Halo-Crb3可有效地保留在ER上,不存在于顶部表面和细胞连接处;而添加生物素使SBP-Halo-Crb3从ER释放,同步转移到高尔基体,并在2小时内被输送到顶部细胞膜(图二c)。通过Cas9介导的基因编辑,在Crb3信号肽后敲入SBP-Halo构建内源性表达Crb3-SBP-Halo的Eph4细胞系,在表达StrKDEL和添加生物素后,可见Crb3在高尔基体中积累,后来在顶部细胞膜上积累,其动力学与过表达系统相似(图二d)。在同一细胞内使用顶部膜蛋白Crb3和侧面膜蛋白Ecadherin的RUSH系统进行实验,发现这些蛋白被独立地运送到相应的膜上(图二e)。重要的是,Ecadherin-RUSH的存在并没有阻碍Crb3的顶部运输。以上数据表明,生物素触发的大剂量膜蛋白运输不会使ER-高尔基体系统出现饱和。
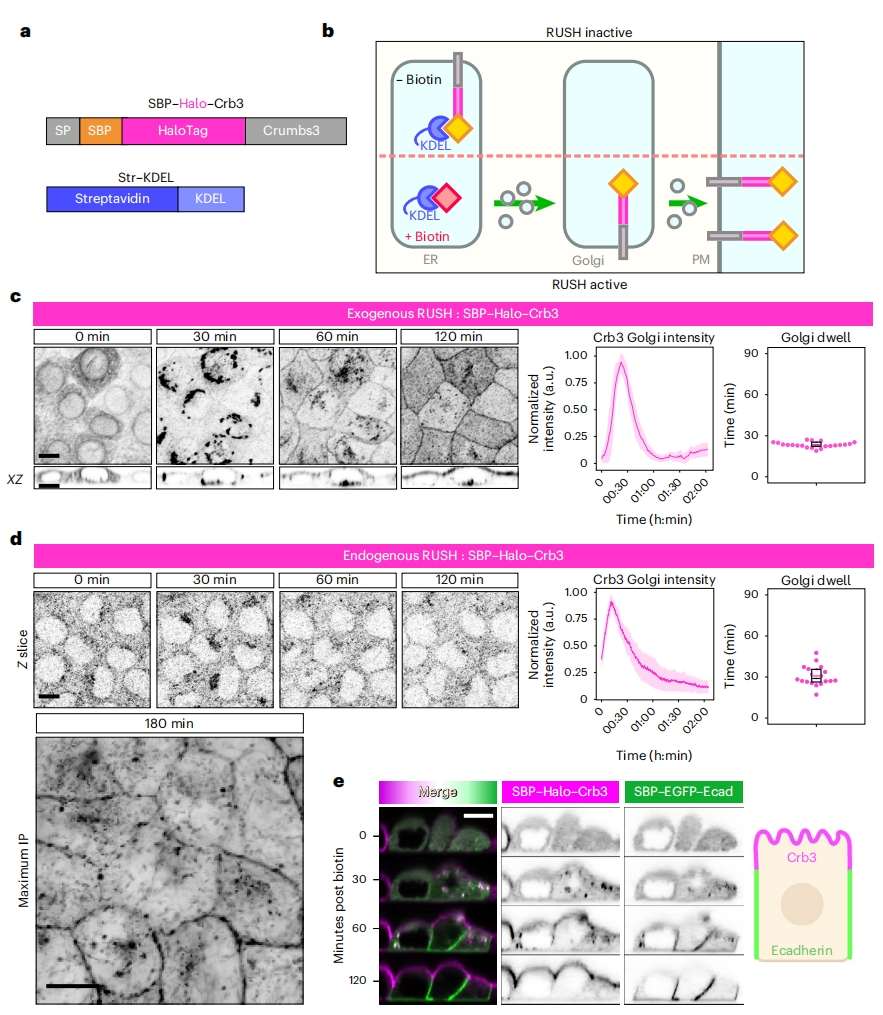
图二 顶部膜蛋白Crb3的同步化顺向运输[6]
随后,研究者评估胞质结构域尾部大小对顶部膜蛋白分选动力学的影响。使用FKBP-FRB化学诱导二聚体策略,通过添加化学诱导(CID),将不同大小标签标记的FRB募集到SBP-Halo-Crb3-FKBP的胞内结构域(图三a和b)。研究者首先比较在稳定状态下,极性Eph4细胞表达SBP-Halo-Crb3-FKBP和对照SBP-EGFP-Crb3,确保FKBP融合到细胞质表面不会损害顶部定位(图三c)。SBP-Halo-Crb3-FKBP可以与胞质中不同大小标签标记的FRB二聚化,产生“大”Crb3;而对照SBP-EGFP-Crb3不能二聚化,为“小”Crb3。另外,选择SNAPtag来标记FRB,因为它是单体,且可被荧光染料标记。当添加CID到这些细胞中时,SNAP-FRB与SBP-Halo-Crb3-FKBP在ER共定位,并在RUSH期间聚集在高尔基体(图三d)。在RUSH前添加CID,导致Halo-Crb3-FKBP在侧面皮质上发生明显的定位错误,而对照组仍保持正常的顶部定位(图三e和f)。启动RUSH后对Crb3组成动力学分析表明,SNAP-FRB蛋白被招募到Crb3-FKBP导致显著延长的高尔基体转运时间(图三g-k),但转运持续时间并没有随着FRB上SNAPtag数量的增加而增加,表明转运延迟受确定的细胞质大小阈值的控制(图三k)。与“小”Crb3相比,附着在ER处的SNAP-FRB-Crb3-FKBP在光漂白(FRAP)后的荧光恢复率没有显著差异(图三l和m)。测试启动RUSH后15分钟高尔基体的FRAP动力学,发现“小”和“大”Crb3之间的迁移率也不存在显著差异(图三n)。以上数据表明,胞质结构域增大会导致Crb3在高尔基体转运延迟和定位错误,这种延迟不是蛋白扩散率降低的结果。
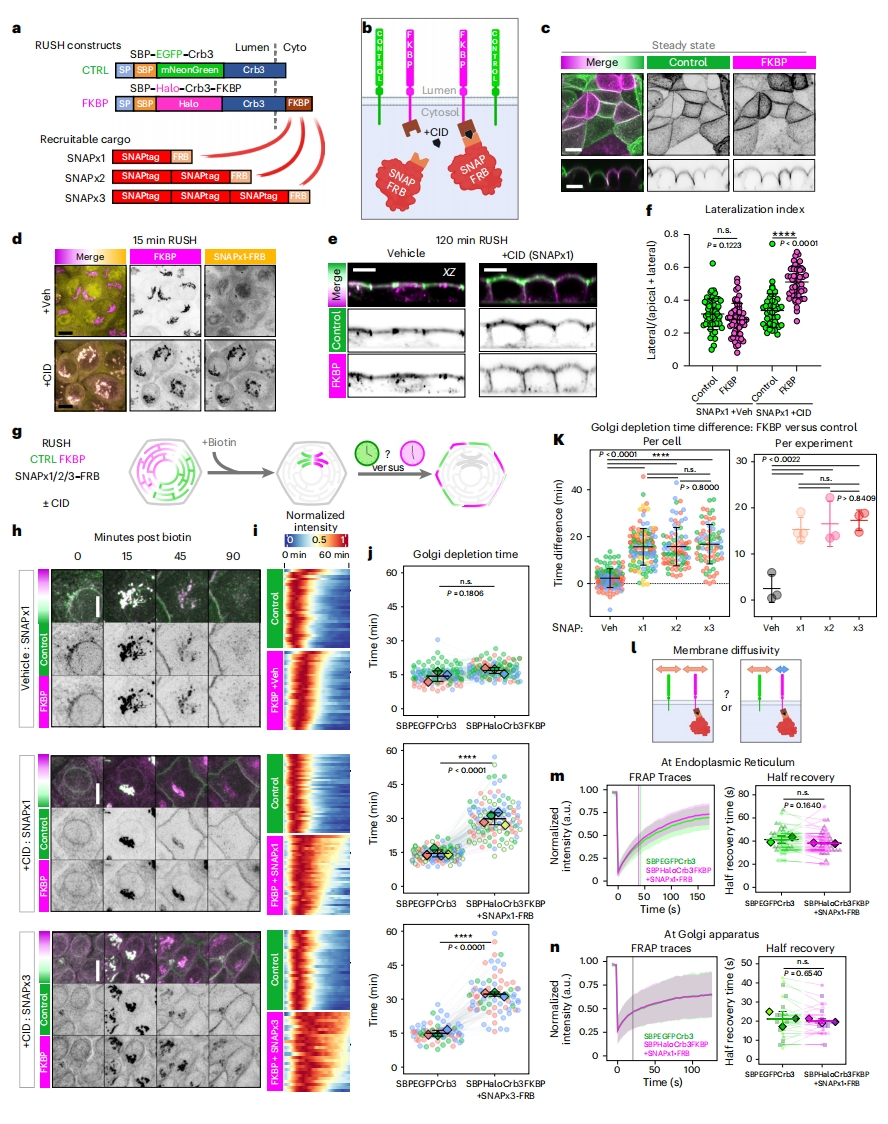
图三 胞质结构域的尺寸调节顶端运输的转运和动态[6]
基于以上数据,研究者推测高尔基体上存在扩散屏障或尺寸过滤器,阻碍具有大胞质结构域的膜蛋白进入顶部分选亚室。与“大”Crb3相比,“小”Crb3将更有效地积聚在这些顶部亚室中。为验证这一推测研究者重复上述实验,使用超分辨率尼康SoRA旋转盘共聚焦显微镜观察活细胞。在CID存在的情况下,高尔基体的不同区域含有“小”GFP-Crb3,但不含有“大”Crb3(图四a和b)。过表达MGAT2-mRuby3或GALT-mRuby3分别标记中间或反式高尔基体[9],观察到与“大”和“小”Crb3有一些重叠,此外,还有与分离的“小”Crb3相对应的离散点,与任一标记都不同(图四c)。以上数据共同表明,高尔基体上存在胞质结构域尺寸依赖的顶部分选机制,该机制可能在反式高尔基体网络(TGN)之前运作。
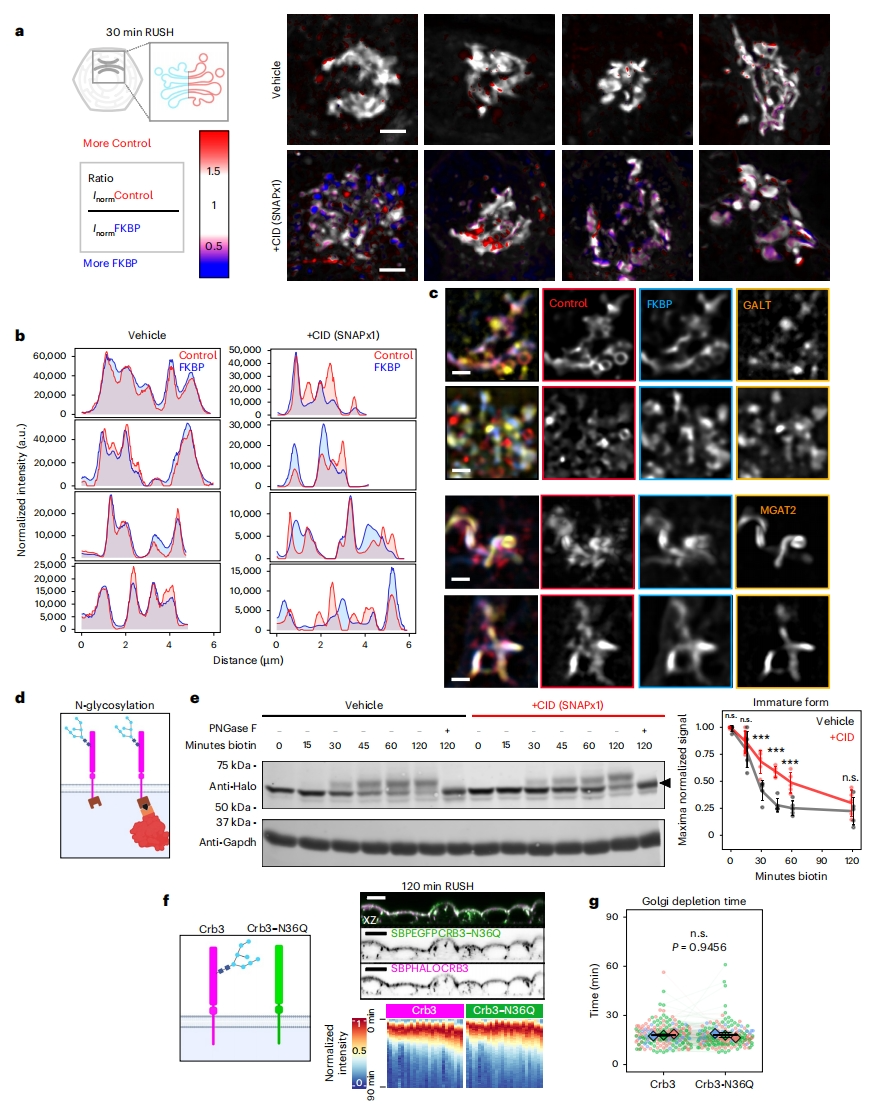
图四 具有小胞质结构域的顶部膜蛋白被优先分选到高尔基体的一个不同区域[6]
Crb3在通过高尔基体的过程中会发生O-和N-糖基化(图四d)。研究者推测在高尔基体中间膜囊之前运作的尺寸过滤器会延迟“大”Crb3的糖基化。于是,在添加生物素后每隔一段时间分离细胞裂解物,并用 SDS-PAGE检测迁移率,N-糖基化会减缓Crb3的电泳迁移率[10]。与“小”Crb3相比,“大”Crb3上面的糖基化条带的形成明显延迟(图四e)。Crb3主要糖基化位点的缺失并不影响高尔基体转运时间或顶部定位(图四f和g)。以上数据表明,阻碍具有大胞质结构域的膜蛋白分选的尺寸过滤器大概位于高尔基体外表面区域,可能位于中间高尔基区域。
许多膜蛋白具有结合伴侣,这种结合作用将大大增加其胞质结构域的大小,如果这种结合发生在内质网或高尔基体,将阻碍顶部膜蛋白的运输。为探究这个问题,研究者在Crb3积累于高尔基体时固定SBP-Halo-Crb3 RUSH细胞,并对一组已知的与Crb3相互作用的蛋白质进行免疫染色。发现Crumbs复合成分Pals1和Patj在高尔基体中与Crb3一起积累,而紧密连接蛋白ZO1和极性蛋白Par6和aPKC均未与Crb3共定位(图五a)。Pals1是一种约74 kDa的细胞质极性蛋白,与Crb3 C末端PDZ配体序列E-R-L-I结合[11]。Pals1的敲除不影响Crb3从内质网到细胞膜的转运动力学,表明这种相互作用对Crb3运输不是必需的。此外,删除Crb3 C末端序列防止Pals1结合也不会阻止Crb3的顶部运输。在Crb3 RUSH Eph4细胞中表达mApple标记的Pals1,并在ER释放Crb3之前和之后跟踪这两种蛋白。通过mApple-Pals1的FRAP确定,当Crb3保留在ER时,其迁移率显著降低,表明形成了Crb3-Pals1复合物(图五b)。此外,mApple-Pals1和SBP-Halo-Crb3的恢复率是相同的,这也表明Pals1与ER处的Crb3有关(图五b)。当激活RUSH时,Crb3和Pals1在高尔基体中同步积累(图五c)。但随着运输的继续,Pals1在Crb3之前从高尔基离开,提前约7分钟(图五c)。Pals1是一种可溶性细胞质蛋白,它以斑点的形式离开高尔基体(图五d),无法确定这些斑点是否为囊泡,或者还有其他蛋白质与Pals1相互作用。为了确认高尔基体的相互作用不是过表达的产物,研究者构建双纯合敲入的Eph4细胞系,给内源Crb3加上SBP-Halo,Pals1加上SNAPtag,确认了内源性Crb3和Pals1之间的共转运(图五e和f)。但由于蛋白质的表达水平低,无法确认Pals1何时离开TGN。以上数据表明,Pals1在高尔基体上与Crb3的解离,而不是复合物的形成,对Crb3转运动力学十分重要。
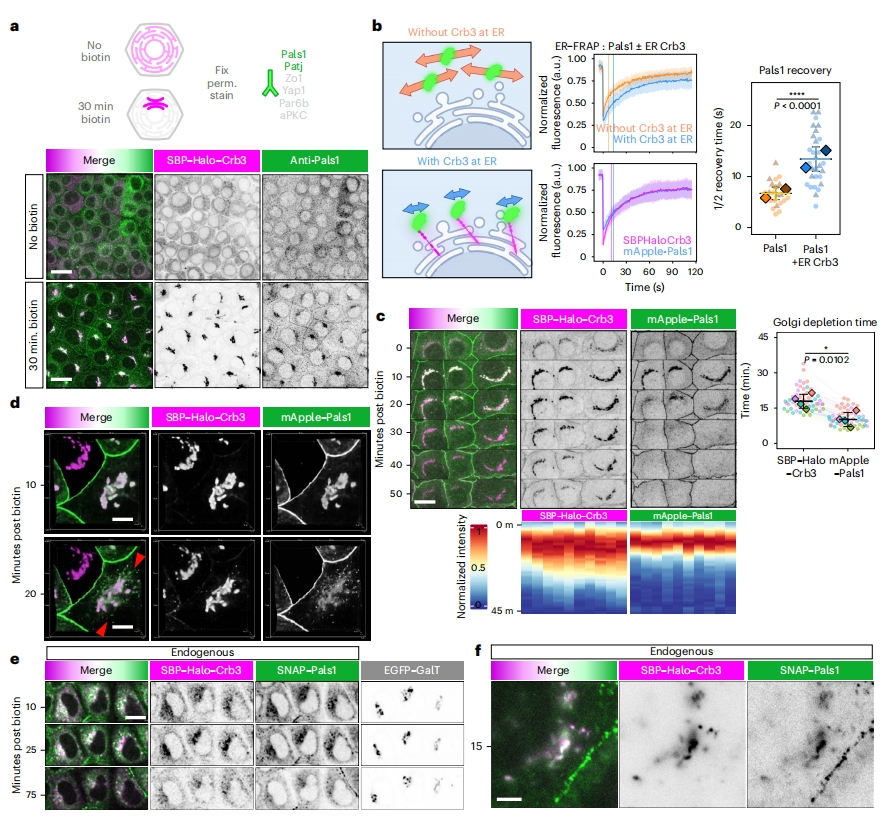
图五 Crb3和Pals1从ER到高尔基体共转运,但在离开高尔基体前分离[6]
最后,研究者探究Pals1的解离是否是Crb3从TGN中快速排出的必要条件。构建Pals1的截短突变体(EGFP-Pals1ΔN),该突变体缺少Par6/Patj/Lin7c相互作用所需的N端部分,保留了与Crb3结合所必需的PDZ-SH3-GUK结构域(图六a)。在Crb3-RUSH实验中,当EGFP-Pals1ΔN表达时,它与Crb3共转运,但不会像全长Pals1那样从高尔基体解离到细胞质中(图六b),Crb3通过高尔基体的时间明显延长(图六c)。以上数据表明,Pals1在高尔基体与Crb3的解离是Crb3顺向运输和递送到顶部细胞膜的重要调控步骤。
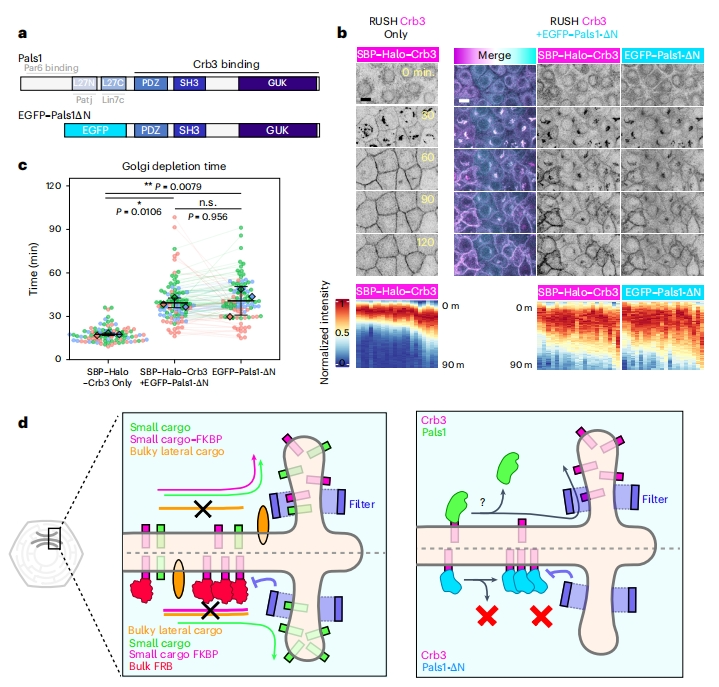
图六 Pals1的解离对于Crb3从高尔基体的离开至关重要[6]
综上所述,该研究结合RUSH和FKBP-FRB系统量化了顶部膜蛋白的分选动力学,并确定了胞质结构域大小对分选动力学的影响。该研究提出在高尔基体存在一种尺寸过滤器,优先允许具有小胞质结构域的膜蛋白进入亚室进行顶部运输,而增加胞质结构域大小会导致部分蛋白错误定位,并减缓高尔基体的转运时间(图六d)。高尔基体的这种定位分选机制在不同的顶部膜蛋白如Crb3、Ace2和Muc1以及不同细胞类型中具有保守性。该研究解析了顶部膜蛋白在上皮细胞的极性建立机制,为膜蛋白和上皮细胞生物学研究提供了新的方向。

GTP研发中心拥有自主知识产权的“人造精子细胞”介导的半克隆技术,可在体外实现“人造精子细胞”的多位点改造并进行功能元件测试,最后将“人造精子细胞”通过卵母细胞注射可以一步法获得基因改造小鼠。基于“人造精子细胞”单倍体打靶高效率的优势,可快速构建定制化小鼠,如基因组安全位点大片段敲入、条件性基因敲除/敲入和人源化基因改造等。多个复杂调控元件可以通过多次体外细胞打靶构建在同一株“人造精子细胞”中,经细胞水平质控后,一步法获得基因改造小鼠。

如有需要欢迎联系我们,GTP研发中心将竭诚为您设计最佳方案,制定专属于您的基因改造小鼠。
网址:http://www.sibcb.ac.cn/gtp/
地址:上海市岳阳路320号
责任编辑:甜点
新媒体运营:suway
参考文献
1. Stoops EH, Caplan MJ: Trafficking to the apical and basolateral membranes in polarized epithelial cells. Journal of the American Society of Nephrology : JASN 2014, 25(7):1375-1386.
2. Legouis R, Jaulin-Bastard F, Schott S, Navarro C, Borg JP, Labouesse M: Basolateral targeting by leucine-rich repeat domains in epithelial cells. EMBO reports 2003, 4(11):1096-1102.
3. Levic DS, Bagnat M: Self-organization of apical membrane protein sorting in epithelial cells. 2022, 289(3):659-670.
4. Paladino S, Pocard T, Catino MA, Zurzolo C: GPI-anchored proteins are directly targeted to the apical surface in fully polarized MDCK cells. The Journal of cell biology 2006, 172(7):1023-1034.
5. Chen YT, Stewart DB, Nelson WJ: Coupling assembly of the E-cadherin/beta-catenin complex to efficient endoplasmic reticulum exit and basal-lateral membrane targeting of E-cadherin in polarized MDCK cells. The Journal of cell biology 1999, 144(4):687-699.
6. de Caestecker C, Macara IG: A size filter at the Golgi regulates apical membrane protein sorting. 2024, 26(10):1678-1690.
7. Zeke A, Dobson L, Szekeres LI, Langó T, Tusnády GE: PolarProtDb: A Database of Transmembrane and Secreted Proteins showing Apical-Basal Polarity. Journal of molecular biology 2021, 433(11):166705.
8. Boncompain G, Divoux S, Gareil N, de Forges H, Lescure A, Latreche L, Mercanti V, Jollivet F, Raposo G, Perez F: Synchronization of secretory protein traffic in populations of cells. Nature methods 2012, 9(5):493-498.
9. Tie HC, Lu L: Studying the Organization of the Golgi by Super-Resolution Microscopy. Methods in molecular biology (Clifton, NJ) 2023, 2557:113-125.
10. Harder JL, Whiteman EL, Pieczynski JN, Liu CJ, Margolis B: Snail destabilizes cell surface Crumbs3a. Traffic (Copenhagen, Denmark) 2012, 13(8):1170-1185.
11. Li Y, Wei Z, Yan Y, Wan Q, Du Q, Zhang M: Structure of Crumbs tail in complex with the PALS1 PDZ-SH3-GK tandem reveals a highly specific assembly mechanism for the apical Crumbs complex. Proceedings of the National Academy of Sciences of the United States of America 2014, 111(49):17444-17449.



