技术分享:乳酸感应器AARS1介导p53乳酰化促进肿瘤发生
新陈代谢异常是癌症的一个重要标志[1]。正常细胞和组织在有氧条件下将葡萄糖代谢成二氧化碳和水,并且在无氧或低氧条件下进行糖酵解将葡萄糖转化为乳酸。然而,大多数癌症细胞即使在有氧的情况下也会发生糖酵解。这种代谢改变被称为“瓦博格效应”,它启动微环境,并导致糖酵解产物如乳酸的升高,以支持癌细胞增殖。乳酸可参与调节各种生物过程,如免疫细胞分化、肿瘤免疫监测、纤维化、缺血性损伤,以及通过一种称为赖氨酸乳酰化的表观遗传修饰调节基因表达[2, 3]。赖氨酸乳酰化是一种连接细胞代谢和蛋白质功能的翻译后修饰,乳酸可以共价修饰蛋白质,但细胞内乳酸监测及其参与乳酰化的机制尚不清楚。
肿瘤抑制因子p53介导多种功能。作为一种转录因子,它通过激活许多控制DNA修复、细胞周期进程和应激下存活的靶点来保护细胞免受恶性转化[4]。在肿瘤发生过程中,p53的活性经常被抑制,超过一半的人类癌症存在p53突变,这损害了p53的肿瘤抑制活性[5]。p53活性也受到翻译后修饰的调控,如磷酸化、乙酰化和泛素化。了解p53调控机制可以拓宽基于提高p53活性的癌症治疗策略。
2024年4月,Cell期刊在线报道了一项研究,发现肿瘤来源的乳酸是p53的天然抑制剂,并筛选发现丙氨酰tRNA合成酶AARS1作为细胞内乳酸感应器,介导了全局性赖氨酸乳酰化。AARS1介导p53的DNA结合域上的K120和K139残基特异性乳酰化,进而破环了p53的液-液相分离和DNA结合,导致降低了p53的体内外肿瘤抑制作用。β-丙氨酸可竞争性拮抗乳酸与AARS1的结合,减少p53乳酰化,减轻肿瘤的发生[6]。
首先,研究者评估TCGA乳腺癌数据库,发现在野生型p53患者中,高血清乳酸水平的患者p53信号通路得分显著降低(图一A),提示肿瘤中的乳酸可能抑制p53的功能。为研究乳酸在体内的作用,研究者在免疫缺陷NSG小鼠接种MMTV-PyMT肿瘤细胞进行乳腺癌造模,并在造模前后每天腹腔注射乳酸钠。结果发现,乳酸钠注射导致提前形成可触摸的肿瘤和更大的肿瘤负荷(图一B),同时显著抑制了p53活性(图一C)。乳酸脱氢酶A(LDHA)可将丙酮酸转化为乳酸,研究者发现LDHA的高表达与乳腺癌患者的低生存率相关。于是,将MMTV驱动Cre重组酶表达的PyMT小鼠与Ldhafl/fl小鼠杂交,获得乳腺上皮Ldha单等位基因缺失小鼠(Ldhafl/+Cre+,图一D),发现该小鼠的肿瘤形成延迟(图一E),肿瘤乳酸和负荷降低(图一F),p53活性增加(图一G)。使用糖酵解抑制剂2-脱氧-D-葡萄糖(2-DG)或LDHA抑制剂草胺酸钠阻断内源性乳酸的产生,发现显著增加了HCT116野生型p53(p53+/+)肿瘤细胞中p53靶基因表达,而在p53-/-肿瘤细胞中没变化(图一H)。在HCT116野生型p53细胞中,MDM2抑制剂Nutlin-3可诱导内源性p53激活,而这一激活作用可被乳酸处理所抑制(图一I)。研究者还使用基于萤光素酶片段互补分析(LCA)的无细胞系统来体外测量p53活性,当萤光素酶活性高时指示p53活性低。结果显示,乳酸或乳酸钠孵育没有改变p53活性,但加入Ldhafl/+ Cre+小鼠的乳腺上皮细胞裂解液可导致p53活性降低,且p53乳酰化程度与其活性的丧失相关(图一J和K)。以上数据表明,肿瘤来源的乳酸是一种p53天然抑制因子,乳酸可以在细胞中被感知,并转化为p53乳酰化,促使p53失活(图一L)。
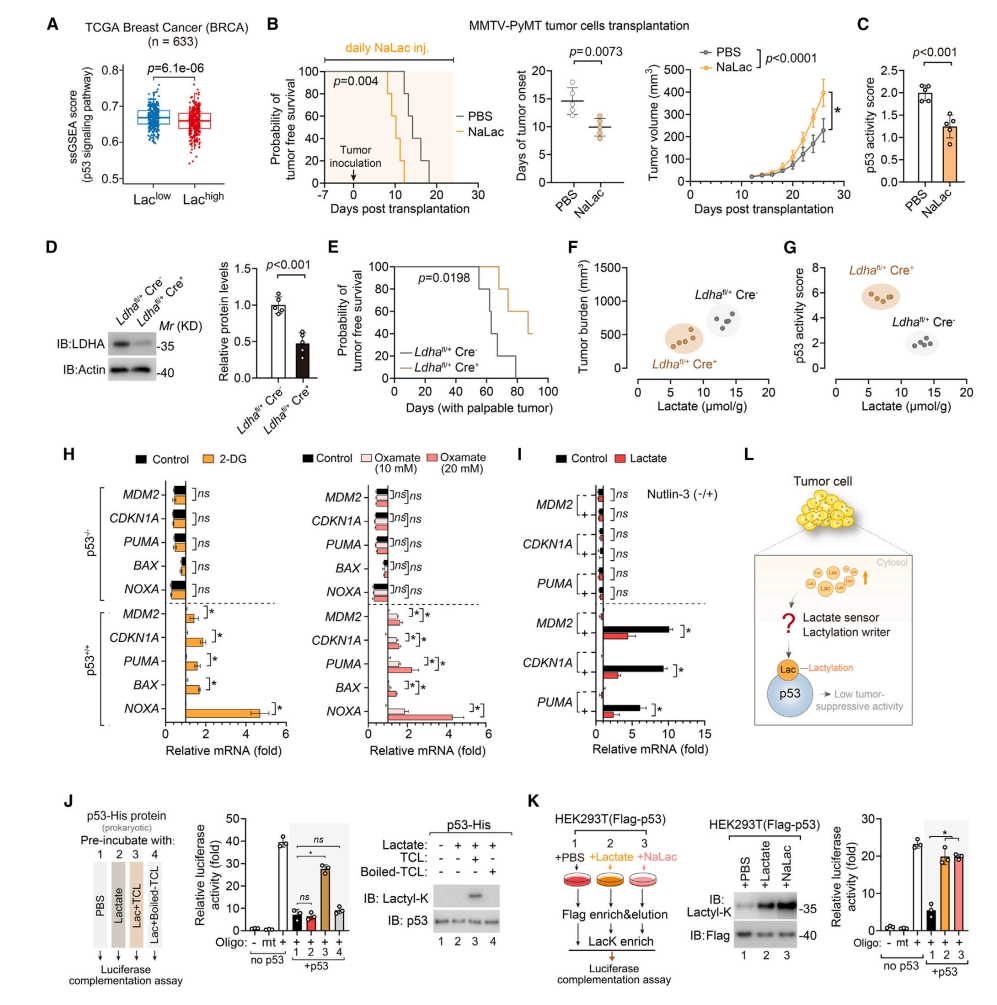
图一 肿瘤来源的乳酸通过p53乳酰化来抑制p53活性[6]
接着,研究者进行全基因组CRISPR筛选寻找负责p53乳酰化的蛋白。构建稳定表达p53诱导报告基因的HeLa细胞,导入全基因组sgRNA文库,在采用乳酸和Nutlin-3处理后,分选富集前10%的GFP阳性细胞进行测序,以鉴定富集的sgRNA(图二A)。发现丙氨酰tRNA合成酶1(AARS1)为首要靶点(图二B)。人类AARS1(简称HsAlaRS)是氨酰基tRNA合成酶(ARS)家族的一员,在蛋白质翻译过程中催化丙氨酸与tRNA(Ala)的连接[7]。使用蛋白质组学方法对全局性赖氨酸乳酰化进行分析(图二C),发现乳酸诱导的乳酰化蛋白质组被AARS1敲降所抑制(图二D)。在AARS1短暂耗竭后,约80%的乳酰化赖氨酸(Klac)肽和蛋白质丰度下降(图二E),其中多达10%的Klac肽和蛋白质减少了10倍以上(图二F)。相反,过表达AARS1增强了全局性赖氨酸乳酰化水平(图二G)。在过表达HsAlaRS的细胞中,90%的Klac肽和蛋白丰度增加,其中近50%的Klac肽和蛋白强度增强了10倍以上(图二H和I),表明AARS1介导了细胞内赖氨酸乳酰化。AARS1在包括大肠杆菌的许多物种中保守(图二J)。大肠杆菌EcAlaRS的过表达也增强了细胞内赖氨酸乳酰化(图二K-M)。对HsAlaRS和EcAlaRS的靶位点和蛋白进行比较,发现了大规模的重叠(图二N),表明AARS1可能在催化赖氨酸乳酰化过程中发挥了古老的作用。β-丙氨酸在结构上类似于乳酸,被广泛用于提高运动表现。有趣的是,用β-丙氨酸预处理细胞后,显示出赖氨酸乳酰化水平的急剧减少(图二O),约90%的Klac肽和蛋白丰度降低,其中10%的丰度降低了10倍以上(图二P和Q)。β-丙氨酸的拮抗作用很好地模拟了AARS1敲降导致的Klac靶位点丰度降低(图二R)。以上数据表明,AARS1可能利用乳酸作为乳酰化的底物,而β-丙氨酸可以有效地拮抗这一过程。
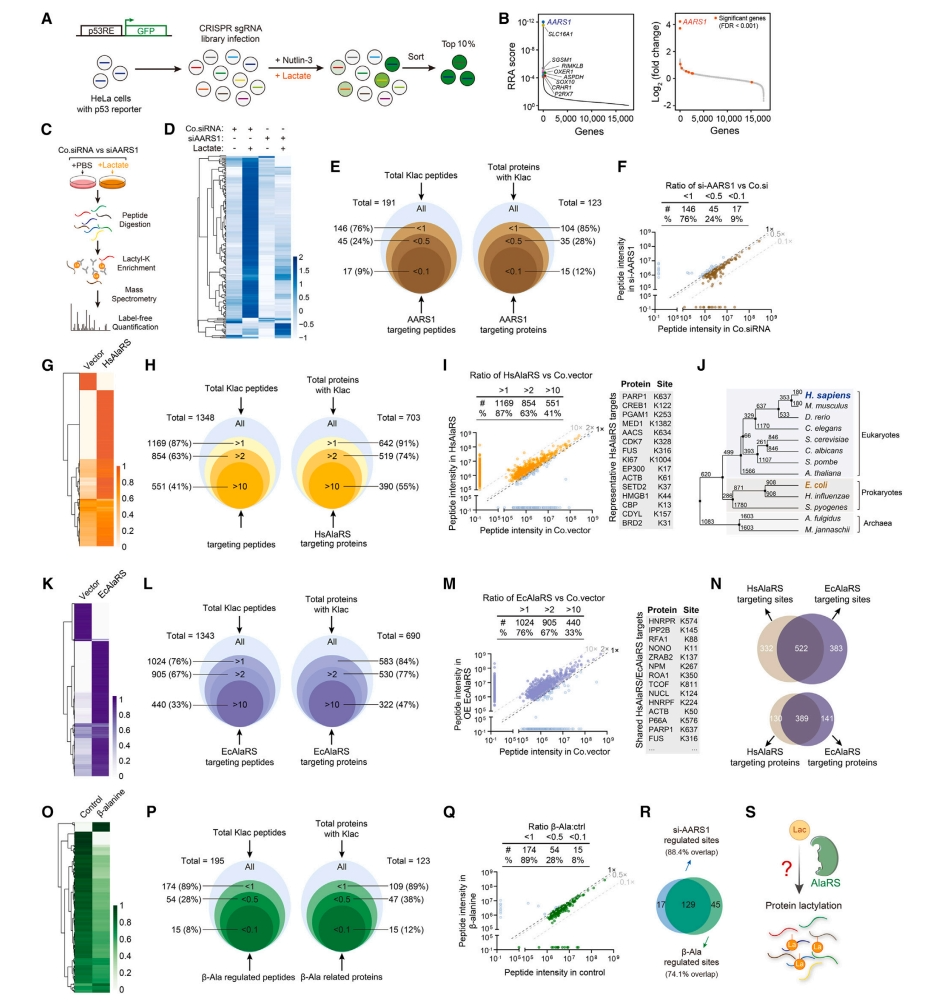
图二 全基因组CRISPR筛选发现AARS1介导了肿瘤细胞的赖氨酸乳酰化[6]
下一步,研究者探索AARS1介导乳酰化的作用机制(图二S)。从细菌中纯化出的EcAlaRS和HsAlaRS可以直接结合乳酸,解离常数分别为13 μM和35 μM(图三A),表明乳酸是其底物。β-丙氨酸对EcAlaRS和HsAlaRS具有更高的亲和性,可以有效地降低乳酸与AARS1之间的结合(图三B和C)。研究者将乳酸分子与HsAlaRS或EcAlaRS的晶体结构进行锚定,以确定结合位点。乳酸与Ala-AMP类似物Ala-SA的丙氨酸部分相重叠,表明乳酸结合位点可能与L-丙氨酸相重叠(图三D)。结果提示HsAlaRS(M46、R77、N216和D239)和EcAlaRS(M43、R69、W170、N212和D235)中的保守残基与乳酸形成了直接接触(图三D)。于是,对这些位点进行突变验证,下拉分析发现将其中一个或两个残基突变为丙氨酸减少了与乳酸的结合,而所有这些保守残基(EcAlaRS-6A和HsAlaRS-5A)的突变直接消除了结合作用(图三E和F)。为了检测AARS1是否直接催化赖氨酸乳酰化,研究者将纯化的EcAlaRS与细胞裂解液孵育。发现只有在反应中同时加入乳酸和ATP后,EcAlaRS-WT才能增强整体赖氨酸乳酰化作用,而结合位点突变体没有作用(图三G和H)。一些合成酶具有编辑活性来避免错误翻译[8]。在该反应中,编辑活性降低的EcAlaRS-C666A突变体在促进乳酰化方面具有与EcAlaRS-WT具有相似的效率(图三H)。类似地,HsAlaRS-WT和HsAlaRS-C723A增加了赖氨酸乳酰化,而结合位点突变体没有作用(图三I-K)。EcAlaRS或HsAlaRS的乳酰化催化活性都可以被β-丙氨酸阻断(图三G和I)。研究者预孵育HsAlaRS、乳酸和ATP以启动反应,随后用旋转脱盐柱去除HsAlaRS后,只有当重新添加HsAlaRS蛋白时,才能观察到细胞裂解液的乳酰化作用(图三L)。使用质谱表征EcAlaRS依赖的赖氨酸乳酰化(图三M),共获得4544个Klac位点,它们分布在1704个Klac蛋白质中(图三N)。其中801个蛋白(47%)仅含有一个Klac位点,而48个蛋白(2.8%)含有超过10个Klac位点(图三O)。分析Klac位点的周围氨基酸序列,发现周围多偏好带正电荷的赖氨酸(图三P和Q)。以上数据表明,AARS1直接与乳酸结合,并以ATP依赖的方式促进赖氨酸乳酰化。
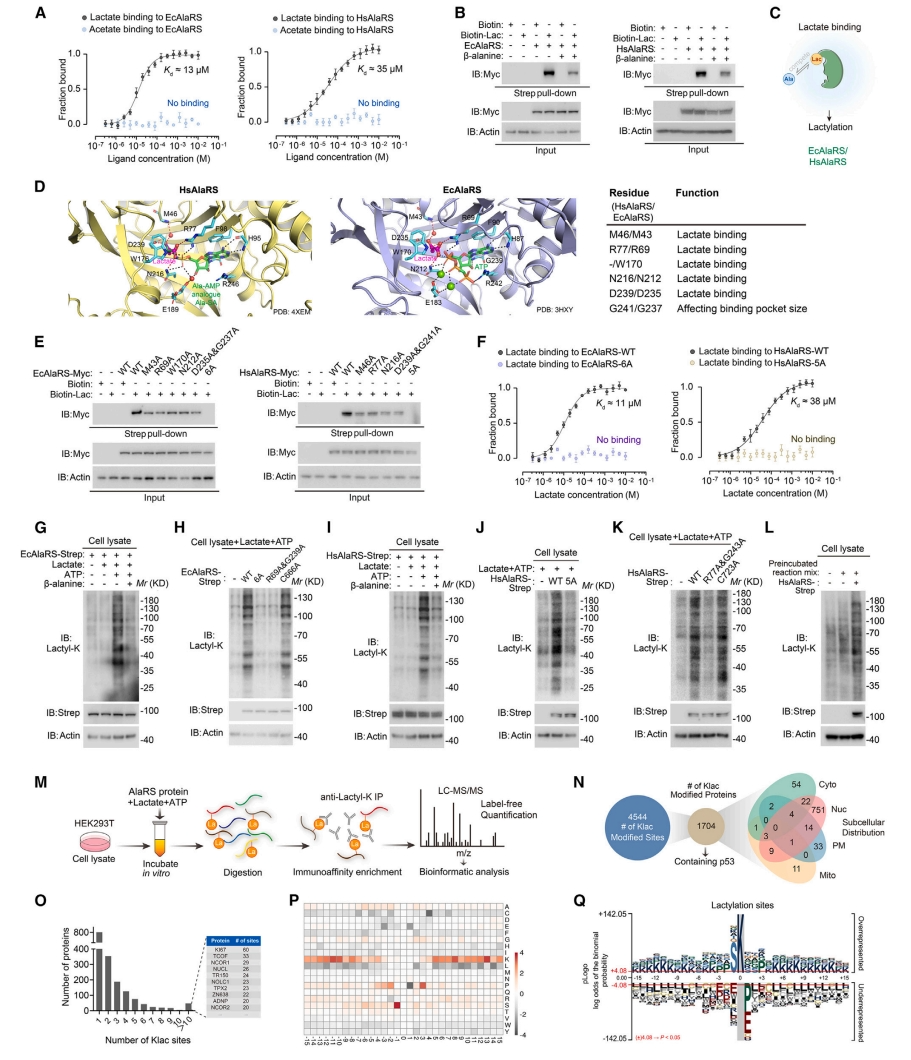
图三 AARS1直接结合乳酸,并以ATP依赖的方式促进赖氨酸乳酰化[6]
来自公共质谱数据的p53相互作用组显示,AARS1和其他蛋白一起作为p53的结合伙伴[9](图四A)。HsAlaRS和EcAlaRS可在体外直接与p53结合(图四B),其解离常数分别达到39 μM和21 μM,接近于它们对乳酸的亲和性(图四C)。在未转染的细胞中,观察到HsAlaRS和p53在多种细胞类型的内源性水平上的相互作用(图四D)。在转染HsAlaRS并用乳酸钠刺激的细胞中,p53在赖氨酸120(K120)和赖氨酸139(K139)位点被乳酰化(图四E和F)。使用特异性识别K120或k139乳酰化p53的抗体,证实了内源性p53中这些残基的乳酰化(图四G)。这些残基乳酰化修饰可被乳酸或乳酸钠激活,并被2-DG和草胺酸钠抑制(图四H和I),并且只有来自AARS1耗尽细胞的裂解液不能对p53进行乳酰化或失活(图四J),表明AARS1是p53的主要乳酸转移酶。在细胞共表达HsAlaRS-WT,而不是HsAlaRS突变体,能有效地乳酰化p53,且可被β-丙氨酸所抵消(图四K)。通过siRNA介导敲低或者单等位基因缺失降低AARS1均可抑制细胞内乳酸诱导的p53乳酰化(图四L)。体外EcAlaRS-WT或HsAlaRS-WT同样可在K120和K139处乳酰化p53,同样被β-丙氨酸所抑制(图四M)。研究者还检测到EcAlaRS-WT或HsAlaRS-WT孵育乳酸和ATP后乳酸-AMP的形成和PPi的释放(图四N)。以上数据表明,AARS1是一种进化保守的酶,通过ATP依赖性催化形成乳酸-AMP,实现将乳酸共价转移到靶蛋白的赖氨酸上(图四O)。
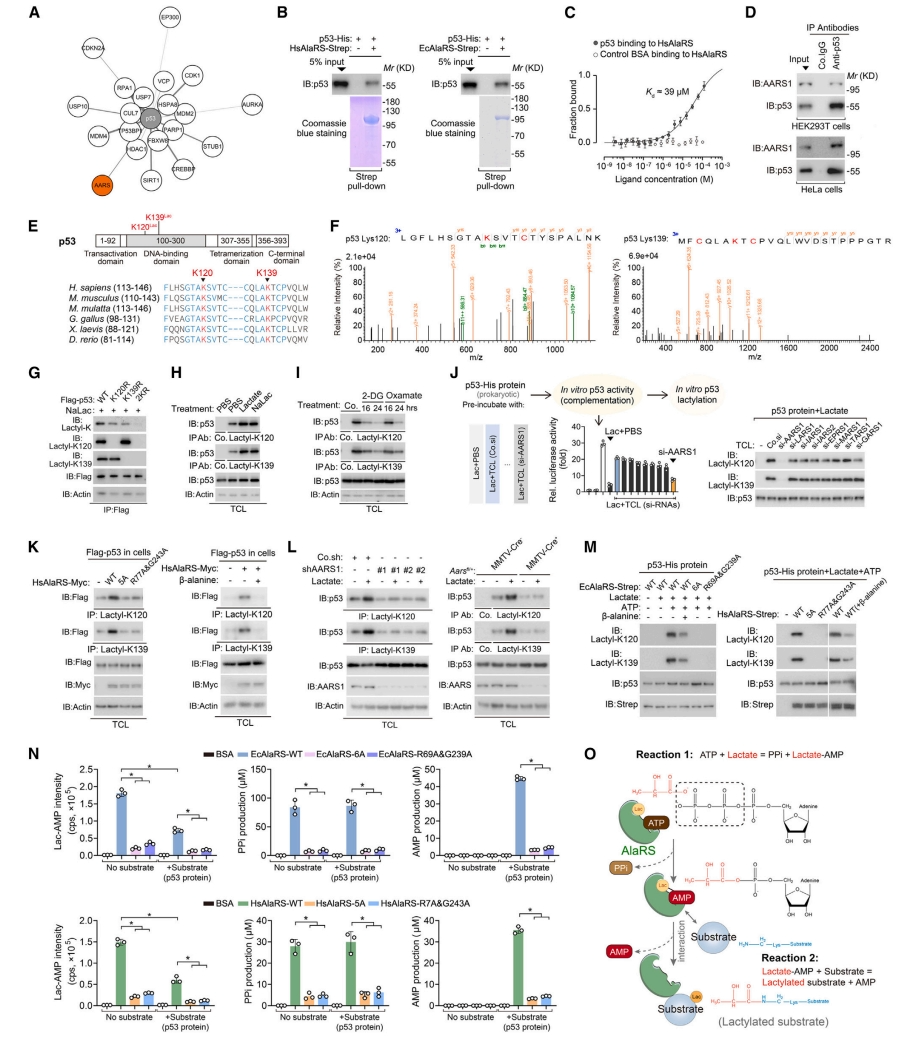
图四 AARS1通过形成乳酸-AMP来催化乳酸与赖氨酸的结合[6]
接下来,研究者探索位于DNA结合域(DBD)的K120和K139乳酰化如何抑制p53活性(图五A)。有研究表明p53可以发生液-液相分离(LLPS)[10]。为了生成位点特异性乳酰化蛋白,研究者开发了一种遗传密码扩展策略来插入N6 -((S)-2-hydroxypropanoyl)-L-lysine(LacK),将LacK掺入p53中,并证实p53K120-Lac、p53K139-Lac和p53Dual-Lac(统称为p53Lac)可分别被对应的特异性抗体所识别(图五B)。与非乳酰化p53(p53Non-Lac)相比,p53Lac对应答元件p53RE-DNA的亲和力分别降低了约100倍、10倍和1000倍(图五C-E)。与p53Lac变体相比,p53Non-Lac在与p53RE-DNA形成液滴方面更为高效(图五F和G)。此外,在LCA测试中,p53Lac的活性低于p53Non-Lac(图五H)。以上结果表明,DBD中的p53乳酰化消除了其LLPS和转录活性。研究者进一步评估在p53-/-细胞中表达FlagLac-p53(p53-WT)和Flag-p53Lac的影响(图五I)。发现Flag-p53Lac蛋白不能结合p53RE-DNA,也不能在刺激反应中凝聚成离散的液滴(图五J和K),并且在驱动p53反应性报告基因和靶基因的表达方面效率低下(图五L)。此外,p53-DBD-WT而不是p53-DBDLac与mCherry-Cry2的融合促使蓝光诱导下的细胞形成液滴(图五M),证实乳酰化损害了细胞内的LLPS。为了检测p53乳酰化的后果,将表达空载体(p53null)、野生型(p53WT)或K120和K139乳酰化p53(p53Dual-Lac)的p53-/-细胞接种到小鼠体内,监测肿瘤生长,发现p53Dual-Lac比p53WT更具致瘤性(图五N)。以上数据表明,p53位点特异性乳酰化减弱了其DNA结合和液-液相分离能力,从而降低了p53的体内外肿瘤抑制作用(图五O)。
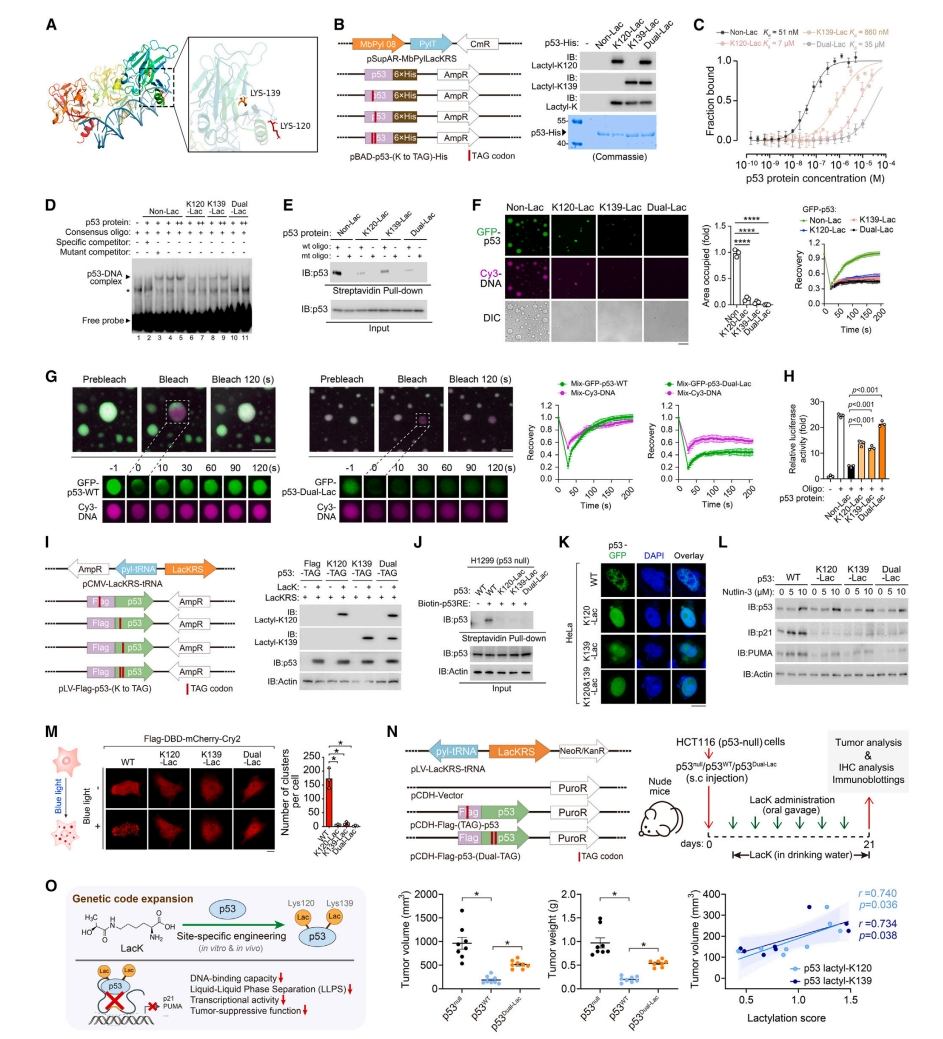
图五 p53位点特异性乳酰化减弱其DNA结合和LLPS,进而降低其肿瘤抑制作用[6]
AARS1在多种肿瘤类型中表达升高,并与WT p53患者的低生存率相关(图六A和B)。最后,研究者评估MMTV-PyMT小鼠中AARS1的作用。Aarsfl/+ Cre+ MMTV-PyMT小鼠的AARS1减少使得发生肿瘤的时间比同窝对照组小鼠更晚,并表现出原发性肿瘤负荷的降低(图六C和D)。对照组小鼠的肿瘤乳酸浓度与肿瘤负荷呈显著正相关,但在AARS1减少小鼠中则没有(图六E)。在AARS1减少小鼠肿瘤中,p53的K120和K139乳酰化减少(图六F),分离细胞对乳酸介导的p53抑制反应较低(图六G),表明AARS1在体内介导p53失活,并有助于肿瘤的发生。研究者对患者来源的肿瘤切片及邻近正常组织进行免疫组化,发现恶性肿瘤的蛋白质组(lactyl-K)和p53(lactyl p53 K120)的乳酰化水平均显著上调(图六H-J)。在TCGA脑肿瘤数据库中,AARS1的表达也与p53活性呈显著负相关(图六K),并在15个新鲜脑肿瘤样本中证实了这一点(图六L),表明人类癌症中包括p53乳酰化在内的全局性赖氨酸乳酰化是受AARS1调控的。为了防止p53乳酰化,研究者体内测试了一种广泛使用的运动补充剂β-丙氨酸的功效[11]。β-丙氨酸处理导致了对细胞增殖、克隆形成和异种移植肿瘤生长的适度抑制,且这些作用被激活p53的基因毒性癌症药物多西环素(Dox)所增强(图六M和N)。β-丙氨酸处理小鼠的肿瘤显示出p53乳酰化程度减少,并且联合Dox处理小鼠的寿命显著增加(图六O)。以上数据表明,β-丙氨酸会破坏乳酸与AARS1的结合,减少p53的乳酰化作用,并减轻肿瘤的发生。
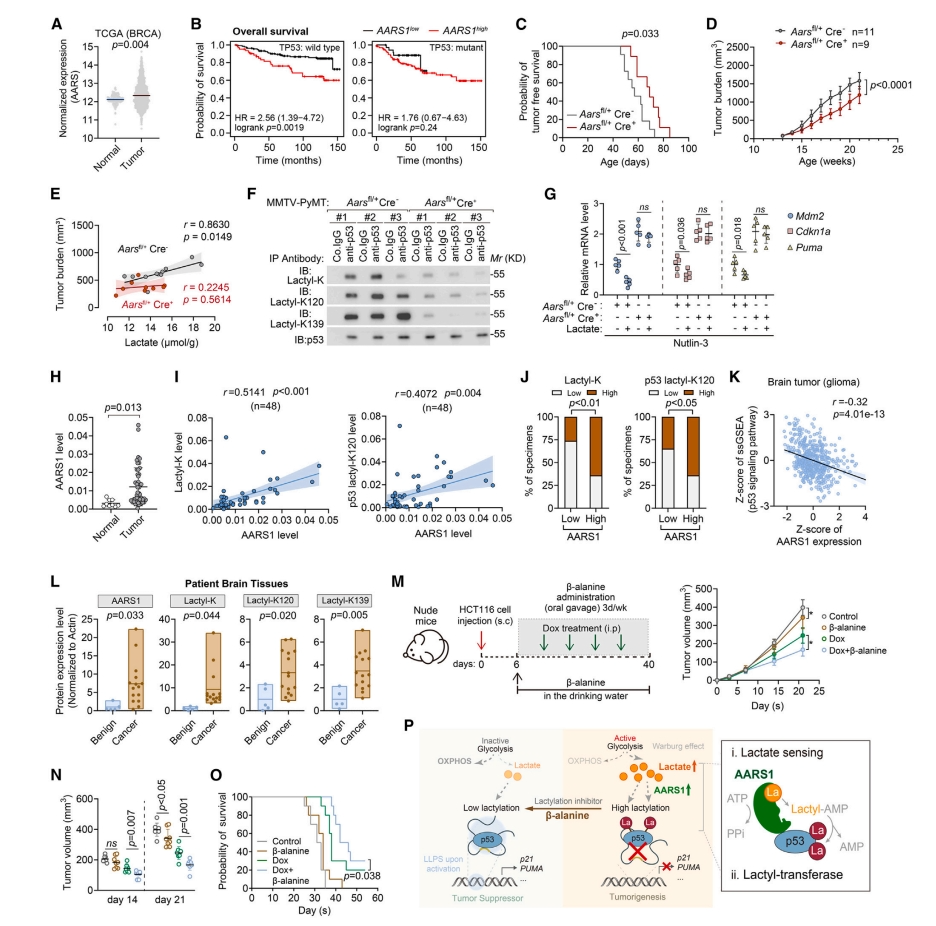
图六 肿瘤生长中AARS1介导的p53乳酰化可被β-丙氨酸拮抗[6]
综上所述,该研究发现AARS1作为一种乳酸感应器和乳酸转移酶,对全局性赖氨酸乳酰化起主导作用。AARS1与乳酸结合并催化乳酸-AMP的形成,然后将乳酸转移到赖氨酸受体残基上。蛋白质组学研究揭示了p53 DNA结合域的两个残基K120和K139,作为AARS1的直接乳酰化靶点,阻断了p53的液-液相分离能力,进而阻断了p53的DNA结合及转录激活,促进了肿瘤的发生(图六P)。在动物模型证实,β-丙氨酸会破坏乳酸与AARS1的结合,减少p53乳酰化作用,并减轻肿瘤的发生。该研究扩展了蛋白质乳酰化的基础知识,揭示了代谢物乳酸与肿瘤发生和发展之间的直接相互作用,为多种人类疾病提供机制上的见解。

GTP研发中心拥有自主知识产权的“人造精子细胞”介导的半克隆技术,可在体外实现“人造精子细胞”的多位点改造并进行功能元件测试,最后将“人造精子细胞”通过卵母细胞注射可以一步法获得基因改造小鼠。基于“人造精子细胞”单倍体打靶高效率的优势,可快速构建定制化小鼠,如基因组安全位点大片段敲入、条件性基因敲除/敲入和人源化基因改造等。多个复杂调控元件可以通过多次体外细胞打靶构建在同一株“人造精子细胞”中,经细胞水平质控后,一步法获得基因改造小鼠。

如有需要欢迎联系我们,GTP研发中心将竭诚为您设计最佳方案,制定专属于您的基因改造小鼠。
网址:http://www.sibcb.ac.cn/gtp/
邮箱:gtp_order@sibcb.ac.cn
地址:上海市岳阳路320号
责任编辑:甜点
新媒体运营:茄子
参考文献
1. Hanahan D, Weinberg RA: Hallmarks of cancer: the next generation. Cell 2011, 144(5):646-674.
2. Zhang D, Tang Z, Huang H, Zhou G, Cui C, Weng Y, Liu W, Kim S, Lee S, Perez-Neut M et al: Metabolic regulation of gene expression by histone lactylation. Nature 2019, 574(7779):575-580.
3. Li X, Yang Y, Zhang B, Lin X, Fu X, An Y, Zou Y, Wang JX, Wang Z, Yu T: Lactate metabolism in human health and disease. Signal transduction and targeted therapy 2022, 7(1):305.
4. Lane DP: Cancer. p53, guardian of the genome. Nature 1992, 358(6381):15-16.
5. Vousden KH, Lu X: Live or let die: the cell's response to p53. Nature reviews Cancer 2002, 2(8):594-604.
6. Zong Z, Xie F, Wang S, Wu X, Zhang Z, Yang B, Zhou F: Alanyl-tRNA synthetase, AARS1, is a lactate sensor and lactyltransferase that lactylates p53 and contributes to tumorigenesis. Cell 2024.
7. Sun L, Gomes AC, He W, Zhou H, Wang X, Pan DW, Schimmel P, Pan T, Yang XL: Evolutionary Gain of Alanine Mischarging to Noncognate tRNAs with a G4:U69 Base Pair. J Am Chem Soc 2016, 138(39):12948-12955.
8. Hilander T, Zhou XL, Konovalova S, Zhang FP, Euro L, Chilov D, Poutanen M, Chihade J, Wang ED, Tyynismaa H: Editing activity for eliminating mischarged tRNAs is essential in mammalian mitochondria. Nucleic acids research 2018, 46(2):849-860.
9. Liao QQ, Shu X, Sun W, Mandapaka H, Xie F, Zhang Z, Dai T, Wang S, Zhao J, Jiang H et al: Capturing Protein-Protein Interactions with Acidic Amino Acids Reactive Cross-Linkers. Small (Weinheim an der Bergstrasse, Germany) 2023:e2308383.
10. Chen C, Fu G, Guo Q, Xue S, Luo SZ: Phase separation of p53 induced by its unstructured basic region and prevented by oncogenic mutations in tetramerization domain. International journal of biological macromolecules 2022, 222(Pt A):207-216.
11. Saunders B, Elliott-Sale K, Artioli GG, Swinton PA, Dolan E, Roschel H, Sale C, Gualano B: β-alanine supplementation to improve exercise capacity and performance: a systematic review and meta-analysis. British journal of sports medicine 2017, 51(8):658-669.



