技术分享:利用天然DNA构建合成染色体
一个细胞存活所必需的最少基因是哪些[1]?为回答这个问题,研究人员采用设计-构建-测试循环策略来合成一条仅包含473个基因的丝状支原体染色体,并且在实验观测周期内可复制产生自由存活的菌体[2]。这个最小的细胞去除了支原体天然存在的428个基因(48%),而在保留的基因中,83%的基因表达与维持遗传信息、细胞膜或胞质代谢功能,余下17%的基因功能未知。这个最小细胞的产生表明可以利用染色体合成来理解定义细胞生命及其多样性机制。
迄今为止,合成染色体的唯一选择是从头合成,通过体内和体外技术相结合,将小的合成DNA片段逐步组装成更大的分子[3]。从头合成的强大优势是允许对染色体的序列和结构进行彻底的重编程。例如,利用从头染色体合成人工合成并替换了全部的4 Mb大肠杆菌基因组,并将其中丝氨酸的密码子TCG和TCA替换为同义密码子AGC、AGT,琥珀密码子TAG替换为TAA,成功构建出一株只含61个密码子的大肠杆菌[4]。Sc2.0项目国际协作组使用从头染色体合成生成的芽殖酿酒酵母,去除所有转座元件,并将LoxP位点插在基因之间,从而能够使用Cre重组酶产生随机的染色体重排[5]。从头染色体合成涉及大量DNA片段的合成和组装,限制了其在生物学研究中的应用。因而,利用天然DNA的克隆片段构建合成染色体可能是一种相对便宜且快速的替代方法。
2023年12月,Nature Communications杂志上报道了一种利用酿酒酵母天然成分构建合成染色体的方法,称为CReATiNG (Cloning, Reprogramming, and Assembling Tiled Natural Genomic DNA)。CReATiNG需要克隆天然染色体片段,然后将它们可编程地组装成合成染色体,以取代受体细胞中的天然染色体。使用CReATiNG可以修改染色体结构,构建多重缺失染色体,以及通过合成染色体与天然染色体之间的重组来修复合成染色体设计中的缺陷[6]。
CReATiNG方法包含两个步骤,第一步是在酵母供体细胞中克隆天然染色体片段,第二步在受体细胞中将这些片段可编程地组装成合成染色体。在第一步中,为克隆靶片段,将三种试剂共转化到由组成型强启动子TDH3驱动表达Cas9的酵母供体细胞中(图一a和b):(1)体外转录gRNA引导Cas9切割靶片段的两端,将其从染色体上切除;(2)构建线性细菌人工染色体/酵母人工染色体(BAC/YAC)克隆载体pASC1,其两侧与靶片段末端同源,能通过同源重组将靶片段整合到克隆载体中;(3)由显性药物标记物KanMX组成的修复模板,其两侧携带的同源臂允许酵母细胞通过同源重组重建断裂的染色体,使得靶片段原位被替换成药筛标记物。靶片段的两侧各设计3个gRNA,同侧gRNA之间的最大距离约为200 bp。pASC1包含BAC/YAC标准组分如URA3标记物等,额外经过工程改造,添加克隆盒,使其可通过定向型限制性酶切和连接进行靶片段的高通量整合,用于克隆特异性靶片段。其中克隆盒约为500 bp,包含两个120 bp的靶片段末端特异性同源臂,由AvrII和XhoI酶切位点分开,两侧是在酵母基因组中不存在的100 bp DNA接头序列和I-SceI位点。设计接头序列用于第二步中不同靶片段的组装,由随机序列生成器生成[7]。确定靶片段后,研究者从头合成克隆盒,通过I-SceI位点整合到pASC1中,再利用AvrII和XhoI酶切线性化pASC1,得到具有不相容的粘性末端且低自连率的线性分子(图一a)。
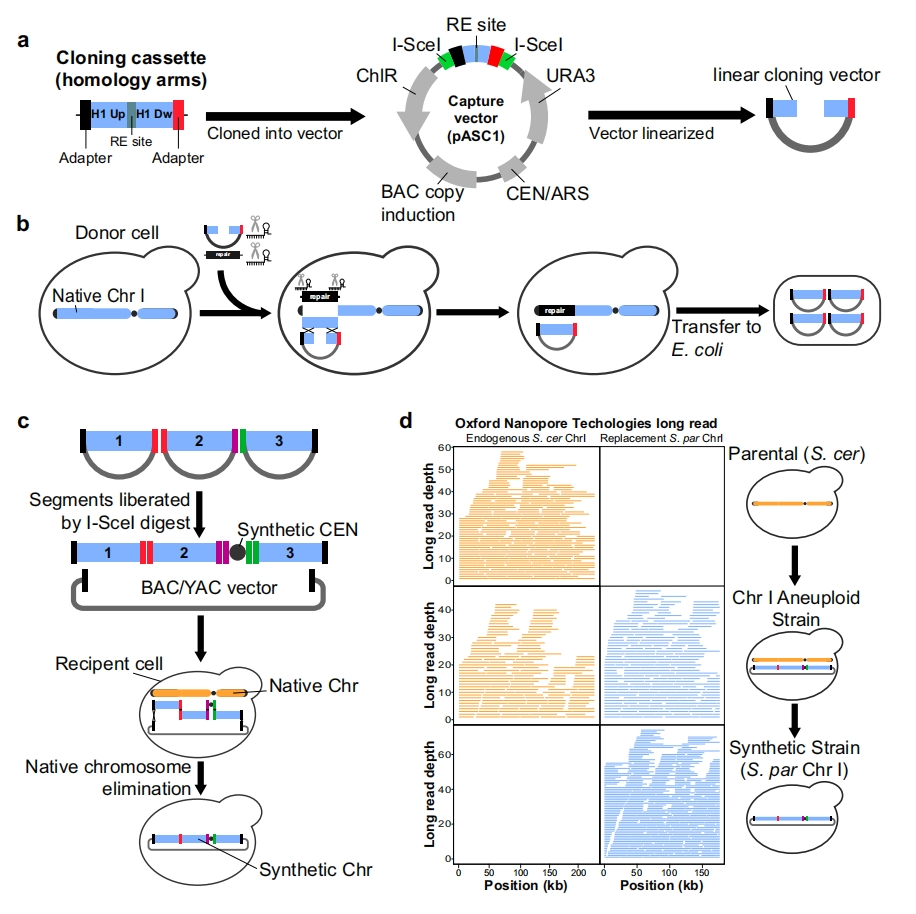
图一 利用CReATiNG合成染色体替代酵母天然染色体[6]
共转化三种试剂后,通过克隆载体上的URA3和修复模板上的KanMX标记物来筛选分离阳性细胞并进行PCR验证(图一b)。阳性克隆的靶片段可以从阳性酵母供体细胞中提取,并转化至EPI300大肠杆菌来高效诱导pASC1拷贝,之后通过用I-SceI酶切将靶片段从载体中分离。共享相同末端接头序列的多个不同片段可以与适当载体一起转化到受体酵母细胞中,从而获得正向排序的合成染色体(图一b和c)。
研究者选择包含117个已知或预测蛋白质编码基因的230 kb的I号染色体(ChrI)[8] 作为范例测试CReATiNG方法。选择奇异酿酒酵母(S. paradoxus)作为初始供体,因其与酿酒酵母约12%的核苷酸差异[9],易于将受体细胞中的合成染色体与天然染色体区分开来。通过分析将供体ChrI分为三个51-64 kb的非重叠片段,包含除着丝粒、亚端粒和端粒外的整个染色体,并避免破坏注释功能元件。随后,按照CReATiNG方法,得到这三个供体ChrI片段。并合成含有CEN6以及酵母(KanMX)和大肠杆菌(ampR)的耐药性标记的合成着丝粒盒。将这三个片段、具有适当接头的着丝粒盒和线性pASC2(一种缺乏着丝粒并含有HIS3作为酵母标记的pASC1)共同转化到酿酒酵母BY4742(BY)菌株中,定向组装这些片段,通过药筛获得阳性单克隆(图一c),进行全基因组Oxford Nanopore Technologies(ONT)测序,确认了两个拷贝ChrI的存在,一个来自BY,另一个来自奇异酵母(图一d)。以上数据表明,CReATiNG方法可成功利用供体天然染色体来合成替换受体细胞。
接下来,研究者探索CReATiNG方法的潜在应用。第一个应用是不同菌株或物种之间的染色体重组,有助于研究遗传表型的遗传基础。研究者另外克隆了酿酒酵母BY和另一种酿酒酵母菌株RM11-1a(RM)的三个ChrI片段。通过接头实现包含3个菌株片段的27种排列组合的新菌株(图二a)。菌落PCR检测这些组合的效率在20-100%之间(图二b),并通过ONT进一步验证组装正确性。随后,研究者在30°C和35°C的完全液体培养基下测量这27个具有重组染色体的菌株的生长速率,发现两种条件下的生长速率都有很大差异,携带完全S.par ChrI的菌株在两种温度下均表现出最慢的生长(图二c和d)。此外,研究者还测量了个体和组合表型效应。结果显示,在30°C时,片段1和片段2对生长有显著影响(图二e);在35°C时,三个片段都对生长有非常显著的影响(图二f)。以上数据表明,三个片段都有助于不同温度下的生长变化,特别是片段3显示出与温度的强烈相互作用。
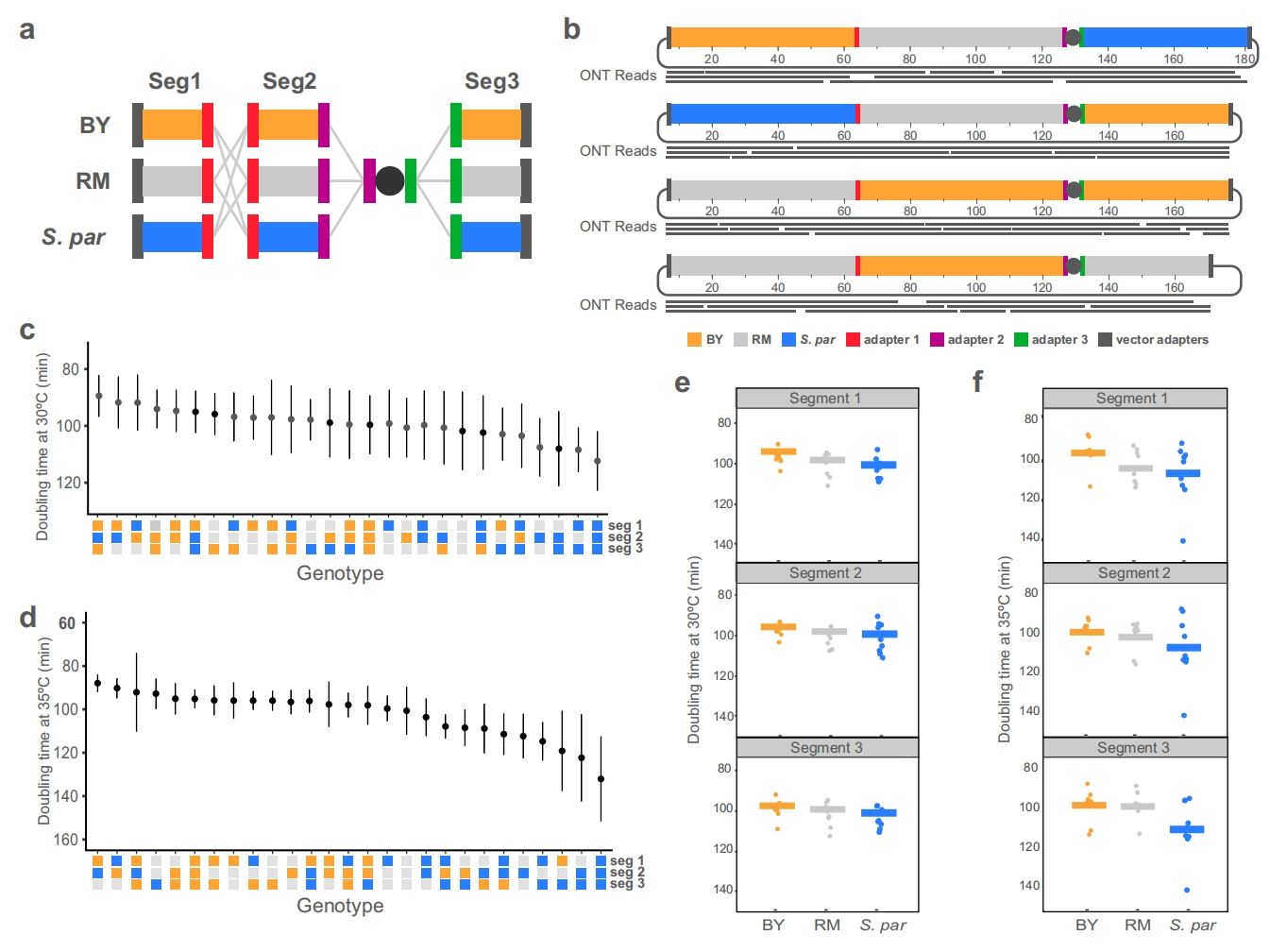
图二使用CReATiNG重组菌株和物种之间的染色体[6]
CReATiNG也可用于合成具有一个或多个倒位、复制、缺失或修改基因顺序的染色体。研究者在BY中克隆了片段1到3,并改变了每个片段两侧的接头序列,使其可以按所有可能的排列组合组装片段,但不倒位任何片段。利用这些重新克隆的片段,设计了5个内容相同但顺序不同的非天然ChrI结构,即1-3-2、2-3-1、2-1-3、3-1-2和3-2-1(图三a),每个组装都通过PCR或ONT测序进行验证。这五种菌株都是可行的,但它们表现出显著的表型变异。相比天然1-2-3构型,2-3-1构型表现出7%的生长改善,3-1-2和1-3-2构型则分别显示出18%和68%的生长减慢(图三b)。由于染色体重组的菌株都具有相同的基因数,对这些实质性生长缺陷的解释是,染色体重组在片段边界上产生的变化可能导致表型重要基因的错误表达[10]。评估所有重组的染色体,在3-1-2和1-3-2菌株中出现了独特的连接,这些连接在其他ChrI构型中都不存在(图三c)。已知在过度表达时会导致生长缺陷的几个非必需基因位于这些独特的连接处:V型马达肌球蛋白(MYO4),囊泡膜SNARE蛋白(SNC1)和核孔蛋白成分(NUP60)[8]。这两种菌株都具有一个连接点,将MYO4置于着丝粒盒KanMX附近。相对于1-2-3野生型菌株,MYO4在1-3-2和3-1-2菌株上的表达分别高出1.5倍和1.2倍(图三d)。以上数据表明,利用CReATiNG编程的重组染色体可以用来识别具有表型效应的非天然染色体构型,以及导致基因过表达的基因邻域的变化是此类实验中生长缺陷的主要原因。
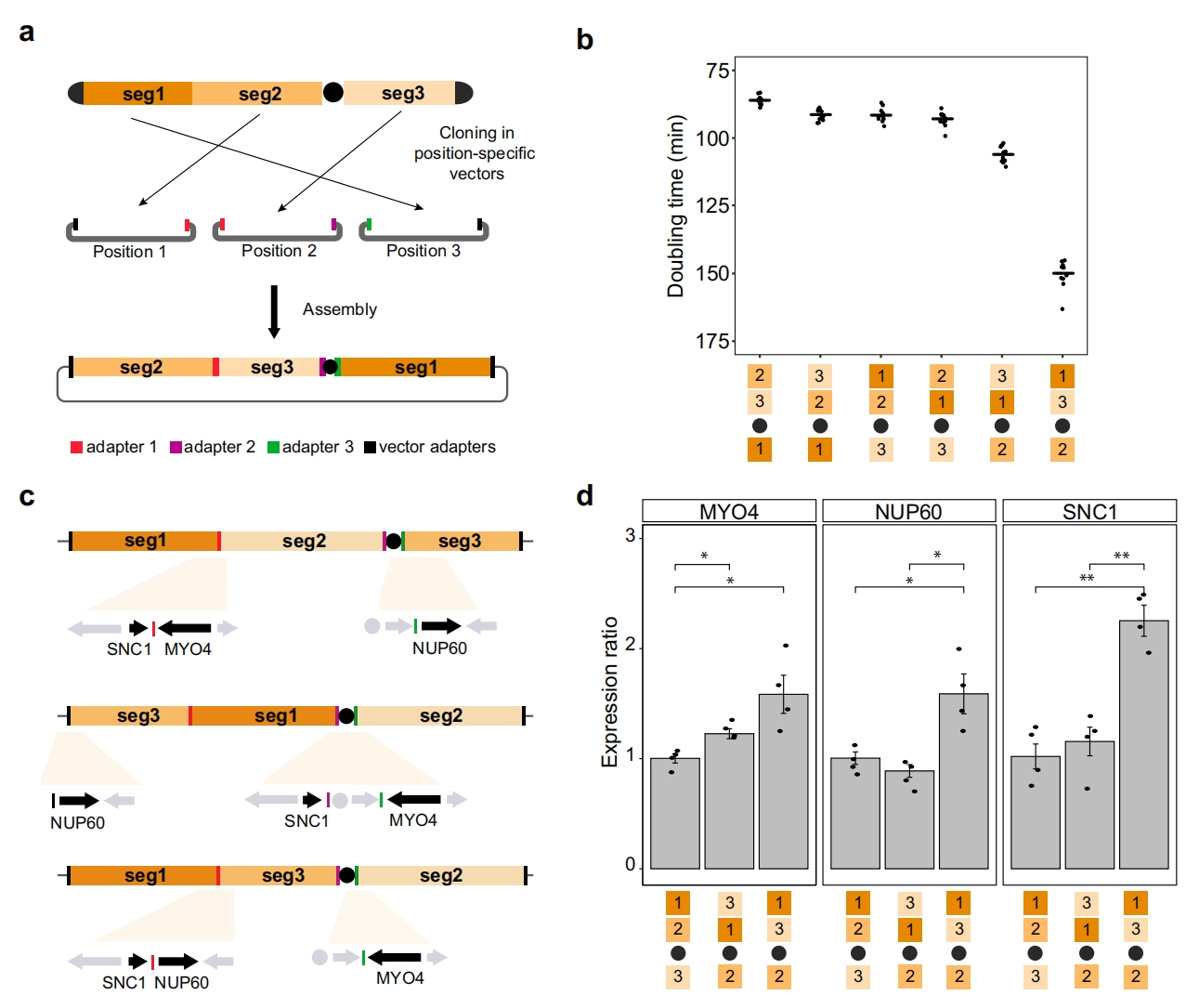
图三 利用CReATiNG重组ChrI[6]
CReATiNG的另一个应用是实现高度多重缺失,这项任务对于传统基因组编辑技术来说仍然具有挑战性[11]。研究者设计了一个工作流程,从BY ChrI中删除10个核心染色体区域,总计30 kb,包含18个不相邻的遗传元件(图四a)。该设计需要克隆11个片段,大小从3.8 kb至21 kb不等,用适当的接头序列对其进行组装。将11个片段与着丝粒盒和线性化pASC2载体一起共转化到BY中。ADE1是一种编码腺嘌呤生物合成酶的基因,其功能丧失会导致菌落积累红色色素,存在于一个目标缺失区域中,为天然ChrI消除提供了额外的表型选择。天然ChrI消除后,挑选10个红色菌落,通过PCR检测每个菌落组装连接处,发现没有一个红色菌落具有所有缺失,但单个红色菌落具有10个缺失中的9个(图四b)。这种具有多重缺失的菌株比具有完整合成BY ChrI的菌株表现出明显较慢的生长(图四c)。在靶向缺失的染色体区域中,只有第9个缺失区域保留在片段6和7段之间,位于该区域的单个基因是SYN8,它编码一个参与囊泡与细胞膜融合的SNARE蛋白。对挑选的10个红色菌落的检查发现都保留了SYN8,这表明它与一个或多个其他缺失元件在基因上相互作用。为了测试这种相互作用,研究者从多重缺失菌株中删除SYN8,发现倍增时间达到394 min,比多重缺失菌株增加了86%,比完整合成BY ChrI菌株增加了319%(图四c)。相反,SYN8缺失对BY没有影响(图四c)。多重缺失揭示了SYN8和ChrI上其他元件之间的未知的遗传相互作用,将SYN8认定为准必需基因[2]。这种未知的相互作用是精简染色体和基因组的主要障碍,它们会导致携带合成染色体的菌株在实验室中表现出生长缓慢和难处理性。然而,SYN8的例子表明,由CReATiNG生成的合成染色体可以通过在天然染色体消除之前与天然染色体重组来克服这种未知的相互作用(图四d)。
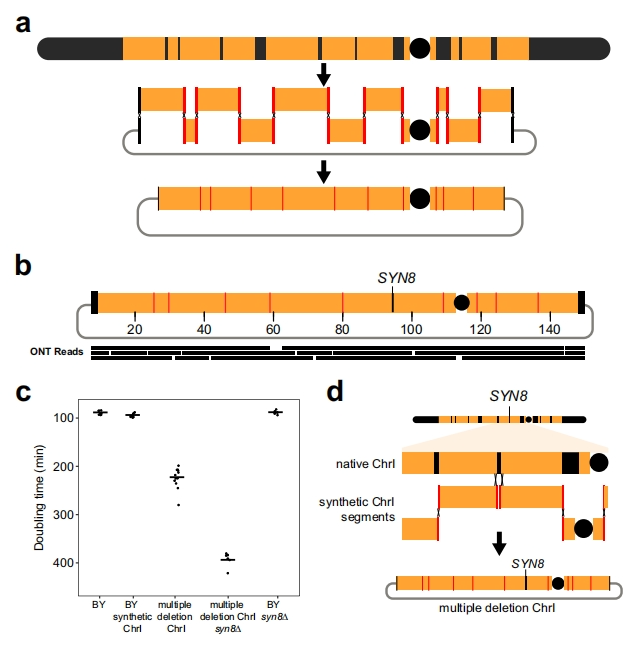
图四使用CReATiNG进行多重基因缺失和染色体精简[6]
综上所述,CReATiNG是一种利用天然DNA成分构建具有多种设计的合成染色体的方法,首先克隆天然染色体片段,然后将它们可编程地组装成合成染色体,以取代细胞中的天然染色体。CReATiNG采用天然染色体的克隆片段,而不是从头合成小DNA片段,比从头合成染色体更具性价比,速度更快。操作过程中还能够通过合成染色体与其天然对应物之间的重组,自主修复合成染色体设计中的缺陷。这些不同的应用凸显出CReATiNG在生物学研究中的作用,可用于研究遗传学、基因组学和进化中的各种基本问题,促进了合成生物学的发展与运用。

GTP研发中心拥有自主知识产权的“人造精子细胞”介导的半克隆技术,可在体外实现“人造精子细胞”的多位点改造并进行功能元件测试,最后将“人造精子细胞”通过卵母细胞注射可以一步法获得基因改造小鼠。基于“人造精子细胞”单倍体打靶高效率的优势,可快速构建定制化小鼠,如基因组安全位点大片段敲入、条件性基因敲除/敲入和人源化基因改造等。多个复杂调控元件可以通过多次体外细胞打靶构建在同一株“人造精子细胞”中,经细胞水平质控后,一步法获得基因改造小鼠。

如有需要欢迎联系我们,GTP研发中心将竭诚为您设计最佳方案,制定专属于您的基因改造小鼠。
网址:http://www.sibcb.ac.cn/gtp/
地址:上海市岳阳路320号
责任编辑:甜点
新媒体运营:suway
参考文献
1. Glass JI, Merryman C, Wise KS, Hutchison CA, 3rd, Smith HO: Minimal Cells-Real and Imagined. Cold Spring Harbor perspectives in biology 2017, 9(12).
2. Hutchison CA, 3rd, Chuang RY, Noskov VN, Assad-Garcia N, Deerinck TJ, Ellisman MH, Gill J, Kannan K, Karas BJ, Ma L et al: Design and synthesis of a minimal bacterial genome. Science 2016, 351(6280):aad6253.
3. Blount BA, Lu X, Driessen MRM, Jovicevic D, Sanchez MI, Ciurkot K, Zhao Y, Lauer S, McKiernan RM, Gowers GF et al: Synthetic yeast chromosome XI design provides a testbed for the study of extrachromosomal circular DNA dynamics. Cell genomics 2023, 3(11):100418.
4. Fredens J, Wang K, de la Torre D, Funke LFH, Robertson WE, Christova Y, Chia T, Schmied WH, Dunkelmann DL, Beránek V et al: Total synthesis of Escherichia coli with a recoded genome. Nature 2019, 569(7757):514-518.
5. Richardson SM, Mitchell LA: Design of a synthetic yeast genome. 2017, 355(6329):1040-1044.
6. Coradini ALV, Ne Ville C, Krieger ZA, Roemer J, Hull C, Yang S, Lusk DT, Ehrenreich IM: Building synthetic chromosomes from natural DNA. Nature communications 2023, 14(1):8337.
7. RANDOM SEQUENCE GENERATOR - random DNA, RNA or protein sequences. https://molbiotools.com/randomsequencegenerator.php.
8. Engel SR, Dietrich FS, Fisk DG, Binkley G, Balakrishnan R, Costanzo MC, Dwight SS, Hitz BC, Karra K, Nash RS et al: The reference genome sequence of Saccharomyces cerevisiae: then and now. G3 (Bethesda, Md) 2014, 4(3):389-398.
9. Ono J, Greig D: A Saccharomyces paradox: chromosomes from different species are incompatible because of anti-recombination, not because of differences in number or arrangement. Current genetics 2020, 66(3):469-474.
10. Brooks AN, Hughes AL: Transcriptional neighborhoods regulate transcript isoform lengths and expression levels. 2022, 375(6584):1000-1005.
11. Utomo JC, Hodgins CL, Ro DK: Multiplex Genome Editing in Yeast by CRISPR/Cas9 - A Potent and Agile Tool to Reconstruct Complex Metabolic Pathways. Frontiers in plant science 2021, 12:719148.



