技术分享:肝脏SAMe调控禁食适应性代谢反应
禁食有助于促进人类健康、延缓衰老和预防肥胖。禁食会触发包括肝脏、大脑和脂肪组织等主要器官系统的适应性代谢反应,该过程受到激素、生长因子和细胞因子组成的复杂网络所调控[1]。大多数哺乳动物禁食12-24小时会导致肝脏肝糖原储备减少,而增加脂肪来源的酮体和游离脂肪酸作为主要能量来源。这种肝脏的适应性代谢反应非常复杂,受到严格调控,包括葡萄糖、脂质和蛋白质代谢的变化。
有研究认为,蛋氨酸(Met)通路中的酶可能在禁食期间受到调节,以维持Met水平稳定[2]。Met在肝脏中通过Met腺苷转移酶(MAT)同工酶I和III代谢合成S-腺苷甲硫氨酸(SAMe)。MAT同工酶主要有3种:MATI、MATII和MATIII。MATI和III的催化亚基(MAT1A)由主要在肝脏中表达的Mat1a基因编码,而MATII的催化亚基(MAT2A)由广泛表达的Mat2a基因编码[3]。SAMe参与肝脏的多胺生物合成、谷胱甘肽(GSH)合成以及甲基转移作用,例如作为磷脂酰乙醇胺(PE)转化为磷脂酰胆碱(PC)的甲基供体。多项研究指出Met循环在维持脂质稳态中发挥重要作用。Met限制饮食(MRD)的啮齿动物表现出肝脏Met和SAMe水平降低、寿命延长、体重增加减缓和胰岛素敏感性增加,并且不会发生肝脏脂肪变性[4]。在人类中,血浆SAMe水平随着体重指数的增加而增加[5]。禁食期间,肝脏SAMe水平下降,而Met水平保持不变,表明在饥饿条件下肝脏SAMe的利用率提高[6]。以上研究提示了一种可能性,SAMe水平的调节,而不是Met本身,可以调节进食/禁食状态下的脂质稳态。然而,这种SAMe响应身体对禁食的代谢反应发生了何种变化,及其发挥何种重要功能仍未可知。
2023年8月,Cell Metabolism期刊上揭示了肝脏SAMe在调节禁食代谢反应中的关键作用。SAMe作为营养的代谢传感器,通过调节肝脏的磷脂酰乙醇胺N-甲基转移酶(PEMT)活性、内质网-线粒体接触、β氧化和ATP产生,以及FGF21介导的脂肪组织脂解和产热,来调节禁食代谢反应。禁食期间,胰高血糖素促进肝脏SAMe合成酶蛋氨酸腺苷转移酶α1(MAT1A)的表达增加,使得SAMe在线粒体相关膜(MAMs)区域被合成,起到刹车的作用,防止过度的β氧化和线粒体ATP合成,最终缓解内质网应激和肝脏损伤[7]。
首先,研究者对小鼠禁食后肝脏Met、SAMe和S-腺苷-L-同型半胱氨酸(SAH)水平进行详细的时间进程研究,发现肝脏SAMe水平早在禁食6小时显著下降,同时甲基转移后形成的副产物SAH水平升高,导致SAMe/SAH比率降低,而Met水平较为稳定(图一A)。当小鼠维持5天的60%热量限制时,也出现类似结果(图一B)。肝脏中的SAMe主要由MAT1A合成,禁食后Mat1a mRNA和MAT1A蛋白表达从12小时到24小时逐渐增加,并在36小时保持增加(图一C和D)。同样,在维持5天60%热量限制的小鼠中,Mat1a mRNA和MAT1A蛋白水平也显著增加(图一E)。糖皮质激素如曲安奈德和地塞米松已被证明可诱导Mat1a的表达[8],研究者测试胰高血糖素是否也有相同作用。实验发现,原代肝细胞培养物经胰高血糖素处理12小时和16小时后,Mat1a mRNA和蛋白质水平也都显著上调(图一F)。禁食过夜的野生型(WT)小鼠经胰高血糖素处理30分钟,肝脏Mat1a mRNA水平就开始有所升高,但这种效果在缺乏胰高血糖素受体的Gcgr-KO小鼠中不发生[9](图一G)。以上数据表明,禁食诱导肝脏SAMe合成酶Mat1a表达上调,且该过程可被胰高血糖素调控;同时SAMe水平减少,且SAH水平升高,提示食物限制时尽管SAMe的合成增加,但其使用显著增强。
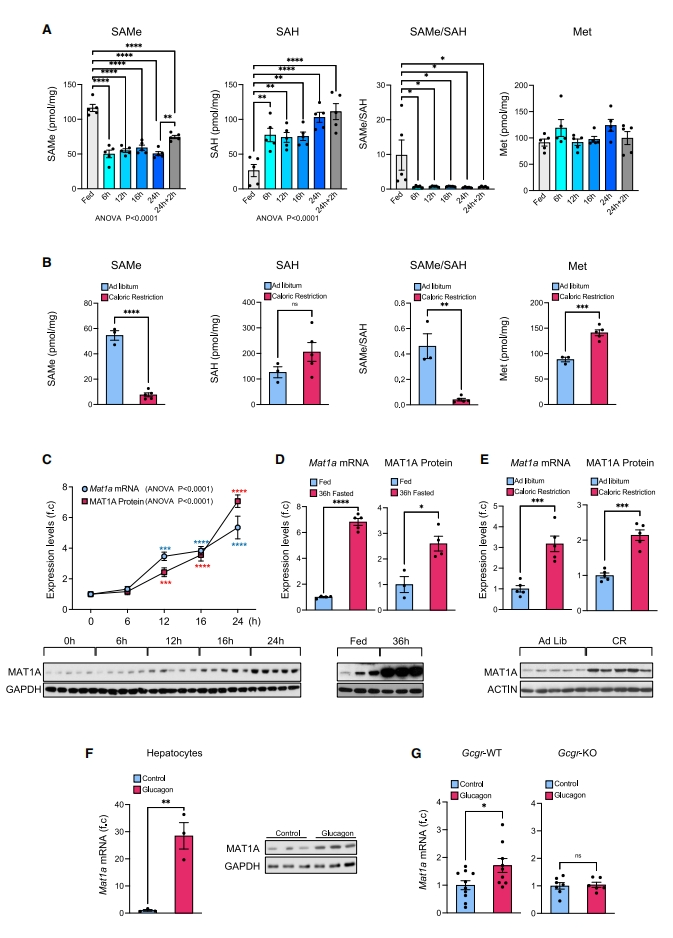
图一 食物限制导致肝脏SAMe的使用增加[7]
接下来,研究者利用MAT1A-KO小鼠模型探讨肝脏MAT1A在脂质代谢中的作用。MAT1A-KO小鼠的特点是肝脏存在明显的高蛋氨酸血症和慢性SAMe缺乏,8月龄时发展成大泡性脂肪变性,过半小鼠在18月龄发展为肝细胞癌[10]。实验发现,MAT1A-KO幼仔的死亡率增加,只有54.8%的幼仔可以存活到断奶(图二A)。此外,MAT1A-KO小鼠的体重从21日龄到90日龄持续下降(图二B)。50-60日龄的WT和MAT1A-KO小鼠的夜间和日间食物摄入相似,表明食物摄入减少不是MAT1A-KO小鼠体重降低的原因(图二C)。研究者还发现MAT1A-KO小鼠的能量消耗(EE)显著增强(图二D),运动活性略有下降(图二E)。MRI分析45-55日龄雄性小鼠,发现MAT1A-KO小鼠的脂肪含量较低(图二F),腹股沟白色脂肪组织(ingWAT)/体重的比值显著减少(图二G),脂肪细胞变小(图二H)。MAT1A-KO小鼠Ucp1 mRNA和蛋白质水平表达上调(图二I和J),以及异丙肾上腺素刺激后的脂肪分解反应增强(图二K),表明ingWAT褐变迹象增加。MAT1A-KO小鼠的棕色脂肪组织(BAT)显示出多房脂滴变小(图二L),Ucp1 mRNA和蛋白质表达增加(图二M和N)。在室温或在急性冷暴露(4℃)下,MAT1A-KO小鼠的肩胛间BAT表面温度显著升高,表明产热增强(图二O)。MAT1A-KO小鼠肝脏的不完全和完全脂肪酸氧化增加(图二P),但不伴随肝损伤(图二Q)。以上结果表明,MAT1A缺失引起肝脏SAMe的慢性缺乏,即使在进食条件下,也会导致分解代谢过度的状态,最终引起小鼠出生后死亡率增加,提示SAMe水平的持续下降可以作为食物限制的代谢传感器来诱导代谢反应。
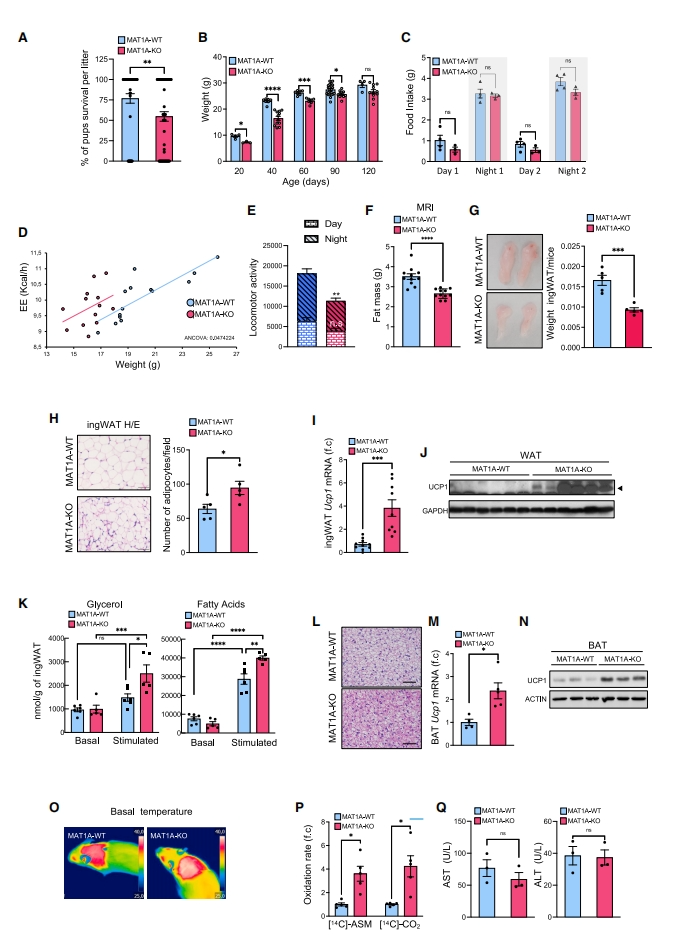
图二 肝脏MAT1A缺失增加体重减轻、能量消耗和出生后死亡率[7]
研究者进一步探索MAT1A-KO小鼠对禁食的反应。对出现脂肪变性前的50-60日龄小鼠进行禁食36小时处理,发现无论雌雄,MAT1A-KO小鼠在禁食期间的体重减轻都比WT小鼠更明显(图三A)。禁食导致MAT1A-KO小鼠的脂肪含量降低(图三B-D),能量消耗增强(图三E),BAT多房脂滴变小(图三F),UCP1蛋白水平增加(图三G),肝脏不完全脂肪酸氧化增加(图三H),分离出的肝细胞在基础和胰高血糖素处理条件下产生更多的ATP(图三I)。随后,研究者从禁食24小时的MAT1A-KO小鼠中分离出肝细胞,显示出基础肝细胞耗氧率(OCR)、ATP相关呼吸、最大呼吸和非线粒体耗氧量的增强,而补充SAMe可以消除这些反应(图三J)。SAMe处理还阻断了MAT1A-KO肝细胞中脂肪酸氧化活性的增强(图三K)。MAT1A-KO小鼠在禁食24小时后出现了肝损伤和内质网应激(图三L和M)。MAT1A-KO小鼠的40%热量限制喂养2个月或隔日禁食(ADF)也诱导出现内质网应激(图三N和O)。以上数据表明,禁食诱导MAT1A缺失小鼠过度激活脂肪组织的动员和使用,以及肝脏β氧化和ATP的产生,最终导致内质网应激和肝脏损伤。
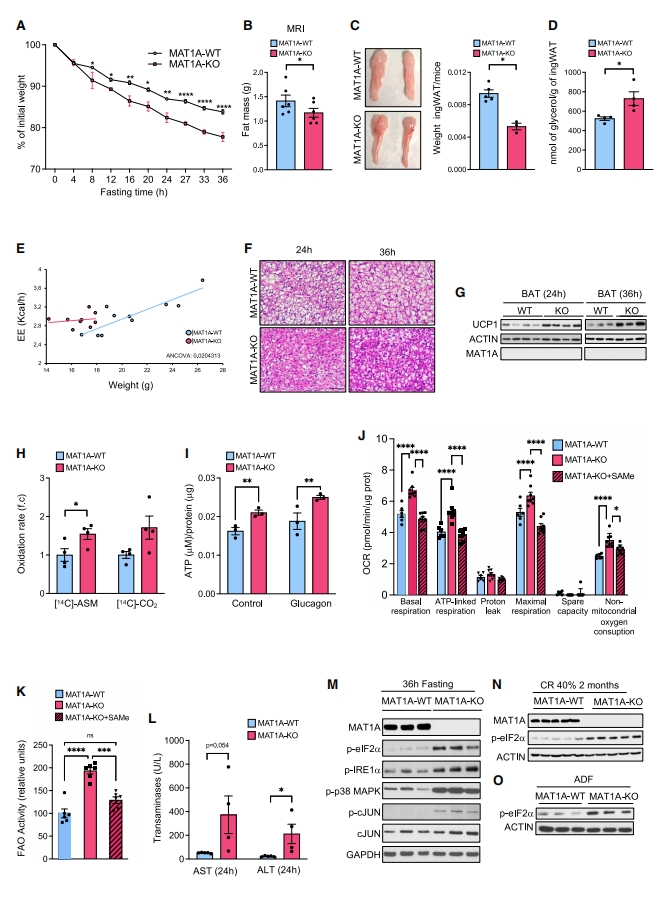
图三 MAT1A缺乏会恶化禁食期间的体重减轻并造成肝脏损伤[7]
FGF21主要在肝脏中表达,在饥饿和蛋白质限制引起的代谢反应中发挥核心作用,主要体现在诱导肝脏脂肪氧化、生酮和糖异生、脂解、WAT褐变以及改善葡萄糖耐受等方面[11]。实验发现,MAT1A-KO小鼠不管在喂食还是禁食的条件下,血清FGF21和肝脏Fgf21 mRNA水平都显著升高(图四A和B);同时,位于Fgf21转录起始位点(TSS)下游的CpG位点(#10–#15)的DNA甲基化水平显著降低(图四C和D)。进一步在MAT1A-KO小鼠肝脏内敲降Fgf21,可以有效降低血清FGF21水平的升高(图四E),缓解禁食期间的体脂和ingWAT重量减轻、脂肪细胞变小和能量消耗增强的表型(图四F-I),而肝脏β氧化、内质网应激和肝脏损伤表型没有改善(图四J-L)。以上结果表明,肝脏MAT1A缺失诱导了FGF21启动子低甲基化,增加了FGF21的肝脏合成和分泌,进而增强能量消耗。
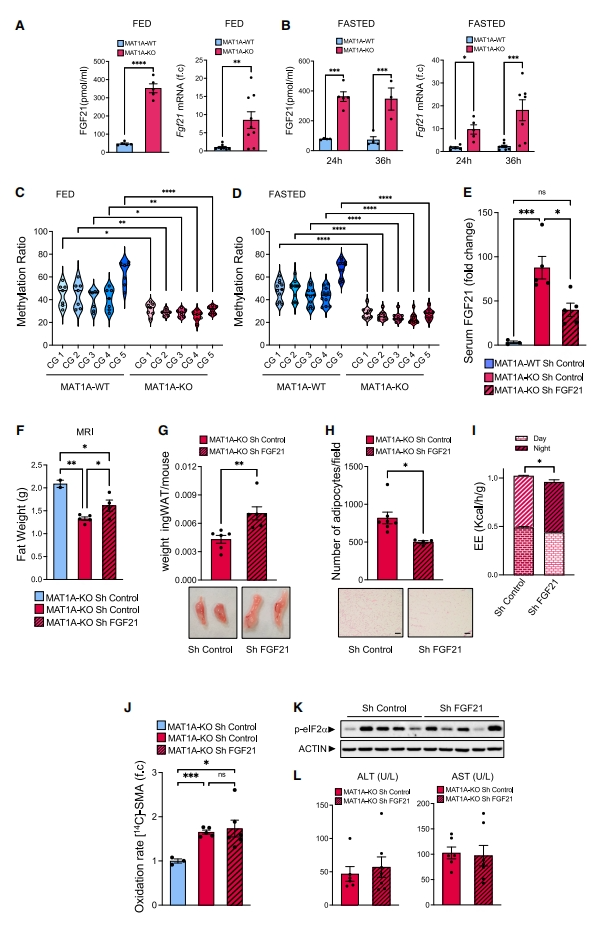
图四 MAT1A缺失降低FGF21启动子甲基化,增加FGF21循环水平和能量消耗[7]
随后,研究者使用核磁共振(NMR)光谱进行代谢流组学实验,追踪13C标记的Met与其他细胞代谢物的相互作用。给禁食24小时的小鼠在检测前30分钟静脉注射13C标记的Met。发现在MAT1A-KO小鼠的肝裂解物中,13C标记的SAMe、高半胱氨酸、高丝氨酸、乳酸和1,6-二磷酸果糖代谢物减少,而13C标记的Met、3-磷酸甘油、葡萄糖-6磷酸和葡萄糖增加(图五A和B),表明禁食期间MAT1A缺失促使Met的代谢通量从SAMe合成向葡萄糖合成转移。通过基于31P NMR的磷酸化蛋白质组学[12]测量PC和PE的总含量,发现24小时禁食的MAT1A-KO小鼠肝脏中PC和PC/PE比率明显下降(图五C)。PC合成途径之一涉及到肝脏磷脂酰乙醇胺N-甲基转移酶(PEMT)对PE的三甲基化,该反应需要三个SAMe分子,PEMT的减少导致PC/PE比率显著降低,导致线粒体呼吸和ATP产生增加[13]。研究者推测在禁食MAT1A-KO小鼠中观察到的β氧化、ATP合成、内质网应激和肝脏损伤的增强可能是由于PEMT活性降低所致。于是,在禁食36小时之前对肝脏PEMT进行急性敲降,发现敲降小鼠的肝脏Pemt基因表达显著降低(图五D),肝脏PC含量和PC/PE比值下降(图五E)。但是Pemt敲降没有导致明显的体重(图五F)和ingWAT质量(图五G)减轻,也没有导致Fgf21 mRNA或蛋白质表达增强(图五H)。相反,禁食期间的Pemt敲降诱导了肝脏β氧化(图五I),并伴有内质网应激和肝脏损伤增加(图五J和K)。以上数据表明,在禁食期间,Met循环流向SAMe和PC合成,其中SAMe水平对于PEMT调节具有重要作用;禁食后减少的SAMe将导致PC/PE比值下降,出现内质网应激和肝脏损伤。
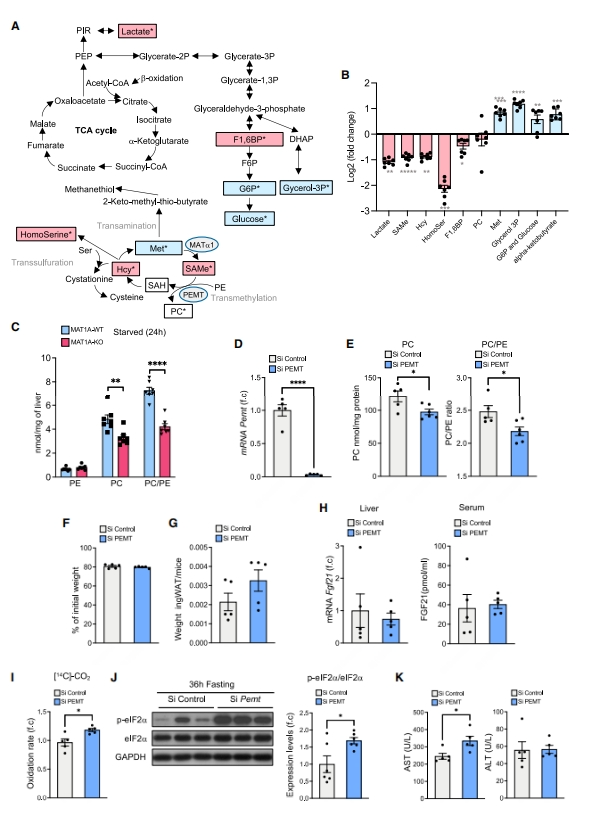
图五 禁食期间蛋氨酸循环流向SAMe和PC合成[7]
内质网向线粒体释放Ca2+,推动了线粒体β氧化。在禁食期间,内质网的特定区域可逆地与线粒体相连,促进这种Ca2+流动。这些接触点被称为线粒体相关膜(MAMs)。研究者使用透射电子显微镜(TEM)检测内质网和线粒体之间的接触程度,发现与WT小鼠相比,在喂食和禁食条件下,MAT1A-KO肝细胞的线粒体和内质网之间的接触比例显著提高(图六A和B),表明肝脏SAMe水平的降低有利于内质网-线粒体接触点的形成,促进Ca2+向线粒体转移和线粒体呼吸。PEMT主要位于MAMs,在禁食期间发挥作用,维持PC合成和磷脂酰丝氨酸(PS)进入线粒体(图六C)[14]。因此,禁食后MAT1A-KO小鼠的SAMe降低,也会降低MAMs中PEMT活性,导致PC/PE降低(图六C)[13]。于是研究者推测在禁食过程中,MAT1A蛋白的合成区域可能与PEMT所在的内质网MAMs区域相近,从而区室化产生其活性所需的SAMe(图六C)。事实发现24小时禁食、隔日禁食、急性或慢性热量限制都会导致细胞器中MAT1A蛋白的增加(图六D和E)。通过差速离心进行亚细胞分级,从新鲜肝组织中获得MAMs、内质网以及粗的和纯的富含线粒体(MP)的级分,发现在禁食6小时后,在MAMs中检测不到MAT1A蛋白,但在禁食16-24小时后高度富集(图六F)。在禁食的MAT1A-KO小鼠的MAMs中未检测到MAT1A(图六G)。最后,研究者测量小鼠的总肝脏提取物和粗线粒体提取物(包括MAMs和线粒体)中的SAMe,发现禁食不会改变WT和MAT1A-KO小鼠的总肝匀浆SAMe水平(图六H),但是禁食24小时MAT1A-KO小鼠的粗线粒体提取物中SAMe水平显著较低(图六I)。以上数据表明,食物缺乏或食物限制会导致SAMe在MAMs中的区室化合成,这种重要机制可以平衡食物缺乏导致的SAMe水平整体降低,防止过度的β氧化和内质网应激,从而防止肝损伤。
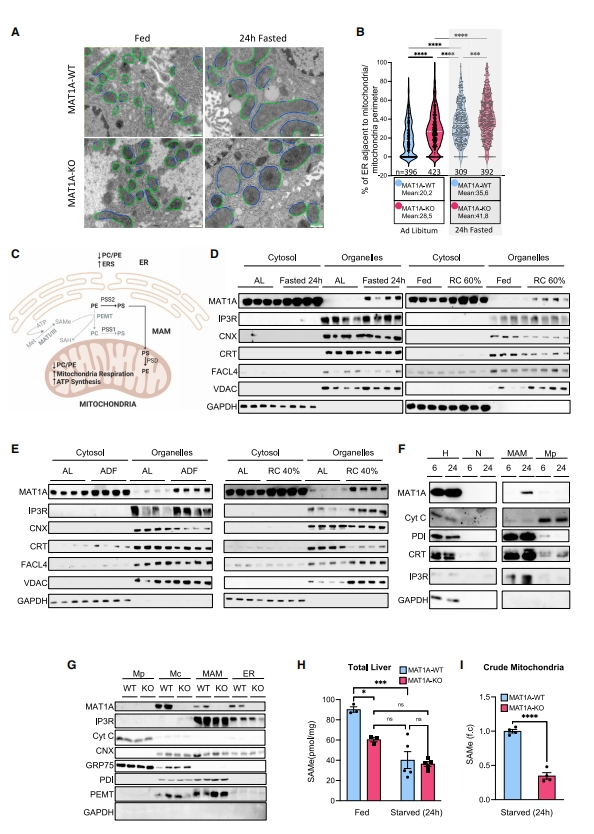
图六 禁食期间MAT1A定位于MAMs[7]
综上所述,该研究揭示了肝脏SAMe在禁食适应性代谢反应中发挥的关键作用,禁食期间肝脏SAMe水平的生理性降低通过微调肝脏线粒体相关膜(MAMs)的相互作用、β氧化和ATP合成,起到营养代谢传感器的作用。肝脏SAMe水平的长期降低会诱导FGF21启动子低甲基化,增强FGF21合成和分泌,从而促进白色和棕色脂肪组织的脂质分解代谢并增加能量消耗。尽管SAMe水平总体下调,但在禁食期间,MAMs中的MAT1A蛋白表达增加,造成SAMe区室化合成,这对于维持PEMT活性以及PC/PE比例发挥了关键作用,限制了线粒体β氧化和ATP合成的速率,最终缓解内质网应激和肝脏损伤。

GTP研发中心拥有自主知识产权的“人造精子细胞”介导的半克隆技术,可在体外实现“人造精子细胞”的多位点改造并进行功能元件测试,最后将“人造精子细胞”通过卵母细胞注射可以一步法获得基因改造小鼠。基于“人造精子细胞”单倍体打靶高效率的优势,可快速构建定制化小鼠,如基因组安全位点大片段敲入、条件性基因敲除/敲入和人源化基因改造等。多个复杂调控元件可以通过多次体外细胞打靶构建在同一株“人造精子细胞”中,经细胞水平质控后,一步法获得基因改造小鼠。

如有需要欢迎联系我们,GTP研发中心将竭诚为您设计最佳方案,制定专属于您的基因改造小鼠。
网址:http://www.sibcb.ac.cn/gtp/
邮箱:gtp_order@sibcb.ac.cn
地址:上海市岳阳路320号
责任编辑:甜点
新媒体运营:suway
参考文献
1. Longo VD, Mattson MP: Fasting: molecular mechanisms and clinical applications. Cell metabolism 2014, 19(2):181-192.
2. Sakata SF, Okumura S, Matsuda K, Horikawa Y, Maeda M, Kawasaki K, Chou JY, Tamaki N: Effect of fasting on methionine adenosyltransferase expression and the methionine cycle in the mouse liver. Journal of nutritional science and vitaminology 2005, 51(2):118-123.
3. Murray B, Barbier-Torres L, Fan W, Mato JM, Lu SC: Methionine adenosyltransferases in liver cancer. World journal of gastroenterology 2019, 25(31):4300-4319.
4. Malloy VL, Krajcik RA, Bailey SJ, Hristopoulos G, Plummer JD, Orentreich N: Methionine restriction decreases visceral fat mass and preserves insulin action in aging male Fischer 344 rats independent of energy restriction. Aging cell 2006, 5(4):305-314.
5. Elshorbagy AK, Nijpels G, Valdivia-Garcia M, Stehouwer CD, Ocke M, Refsum H, Dekker JM: S-adenosylmethionine is associated with fat mass and truncal adiposity in older adults. The Journal of nutrition 2013, 143(12):1982-1988.
6. Zubiete-Franco I, García-Rodríguez JL, Martínez-Uña M, Martínez-Lopez N, Woodhoo A, Juan VG, Beraza N, Lage-Medina S, Andrade F, Fernandez ML et al: Methionine and S-adenosylmethionine levels are critical regulators of PP2A activity modulating lipophagy during steatosis. J Hepatol 2016, 64(2):409-418.
7. Capelo-Diz A, Lachiondo-Ortega S, Fernandez-Ramos D, Canas-Martin J, Goikoetxea-Usandizaga N, Serrano-Macia M, Gonzalez-Rellan MJ, Mosca L, Blazquez-Vicens J, Tinahones-Ruano A et al: Hepatic levels of S-adenosylmethionine regulate the adaptive response to fasting. Cell metabolism 2023, 35(8):1373-1389 e1378.
8. Gil B, Casado M, Pajares MA, Boscá L, Mato JM, Martín-Sanz P, Alvarez L: Differential expression pattern of S-adenosylmethionine synthetase isoenzymes during rat liver development. Hepatology 1996, 24(4):876-881.
9. Finan B, Clemmensen C, Zhu Z, Stemmer K, Gauthier K, Muller L, De Angelis M, Moreth K, Neff F, Perez-Tilve D et al: Chemical Hybridization of Glucagon and Thyroid Hormone Optimizes Therapeutic Impact for Metabolic Disease. Cell 2016, 167(3):843-857 e814.
10. Martinez-Chantar ML, Corrales FJ, Martinez-Cruz LA, Garcia-Trevijano ER, Huang ZZ, Chen L, Kanel G, Avila MA, Mato JM, Lu SC: Spontaneous oxidative stress and liver tumors in mice lacking methionine adenosyltransferase 1A. FASEB journal : official publication of the Federation of American Societies for Experimental Biology 2002, 16(10):1292-1294.
11. Martinez-Garza U, Torres-Oteros D, Yarritu-Gallego A, Marrero PF, Haro D, Relat J: Fibroblast Growth Factor 21 and the Adaptive Response to Nutritional Challenges. International journal of molecular sciences 2019, 20(19).
12. Bernardo-Seisdedos G, Bilbao J, Fernández-Ramos D, Lopitz-Otsoa F, Gutierrez de Juan V, Bizkarguenaga M, Mateos B, Fondevila MF, Abril-Fornaguera J, Diercks T et al: Metabolic Landscape of the Mouse Liver by Quantitative (31) P Nuclear Magnetic Resonance Analysis of the Phosphorome. Hepatology 2021, 74(1):148-163.
13. van der Veen JN, Kennelly JP, Wan S, Vance JE, Vance DE, Jacobs RL: The critical role of phosphatidylcholine and phosphatidylethanolamine metabolism in health and disease. Biochimica et biophysica acta Biomembranes 2017, 1859(9 Pt B):1558-1572.
14. Cui Z, Vance DE: Expression of phosphatidylethanolamine N-methyltransferase-2 is markedly enhanced in long term choline-deficient rats. The Journal of biological chemistry 1996, 271(5):2839-2843.



