技术分享:描绘肝癌侵袭前沿区细胞图谱,解析肿瘤细胞-肝细胞通讯介导的局部免疫抑制和肿瘤进展
实体肿瘤是一种复杂的、高度异质性的生态系统,其中癌细胞能够与各种细胞类型相互作用,包括免疫细胞和基质细胞,以及细胞外基质(ECM)。肿瘤边缘区是肿瘤细胞侵入癌旁组织并与其他细胞直接接触的区域,也是肿瘤细胞浸润和侵袭最活跃的区域[1]。因此,全面了解肿瘤组织及其边缘区域的细胞组成、空间异质性及其与肿瘤微环境(TME)的相互作用,不仅可以揭示肿瘤是如何发展和转移的,还可以加速研发新型癌症治疗方法[2]。
单细胞RNA测序(scRNA-seq)是一种强大的工具,可用于分析单细胞的基因表达水平,剖析细胞内和细胞间的分子动力学,已被广泛用于描述多种类型的实体瘤及其相关TME[3]。然而,单独的scRNA-seq不能提供细胞的空间信息[4],并且由于缺乏多个区域的采样,人们对肿瘤内的单细胞空间异质性仍然知之甚少[5]。近期发展起来的空间增强分辨率组学测序(Stereo-seq)技术能够同时实现“纳米级分辨率”和“厘米级全景视场”,通过结合DNA纳米球阵列芯片和RNA原位杂交,每个细胞可以捕获几百个数据点[6]。该技术能够在单细胞水平上深入表征整个肿瘤生态系统的功能和位置信息,并精确揭示其细胞组成与分布,TME中的细胞-细胞通讯,特别是在肿瘤边缘区域。肝癌,包括肝细胞癌(HCC)和肝内胆管细胞癌(ICC),是最常见且最具侵袭性的肿瘤类型,全球的发病率和死亡率高。因此,剖析和了解肝癌生态系统,特别是在肿瘤边缘区域,将有助于探索肿瘤浸润和侵袭机制,开发新的肝癌治疗策略。
2023年6月,Cell Research期刊上报道了一项新研究,利用Stereo-seq和scRNA-seq技术,描绘了人原发性肝癌不同区域组织的单细胞和空间转录图谱。该研究将肝脏肿瘤边界两侧各250 µm内的区域定义为肿瘤侵袭前沿区(invasive zone),其具有高度的细胞和转录异质性,表现为局部免疫抑制,肿瘤细胞代谢重编程以及血清淀粉样蛋白A1和A2(SAA1和SAA2,统称为SAAs)高表达的严重肝细胞受损[7]。
首先,研究者系统性表征人类肝癌的复杂转录结构。将23例病理诊断为原发性肝癌(HCC,n=6,ICC,n=17)患者作为发现队列(discovery cohort),并对其肿瘤组织(T)、肿瘤边缘区域(M)、癌旁组织(P)和正常或转移的淋巴结(LN)进行分析。其中来自21例患者的53个样本用于Stereo-seq分析,5例患者的16个样本用于scRNA-seq分析(图一a)。将另外5组癌症患者作为验证队列(validation cohort),用于实验验证发现队列的分析结果(图一a)。研究者从发现队列中获得33,111个细胞的scRNA-seq数据,并将其与最近发表的ICC scRNA-seq数据[8]整合,建立各细胞类型的无偏参考表达指纹图谱,从而定义Stereo-seq中不同细胞群体的空间断层图。通过SPOTlight[9]测定细胞类型,得到9种主要细胞类型的空间注释(图一b和c)。标志基因在相应细胞类型中的高表达支持了这一注释结果(图一d)。
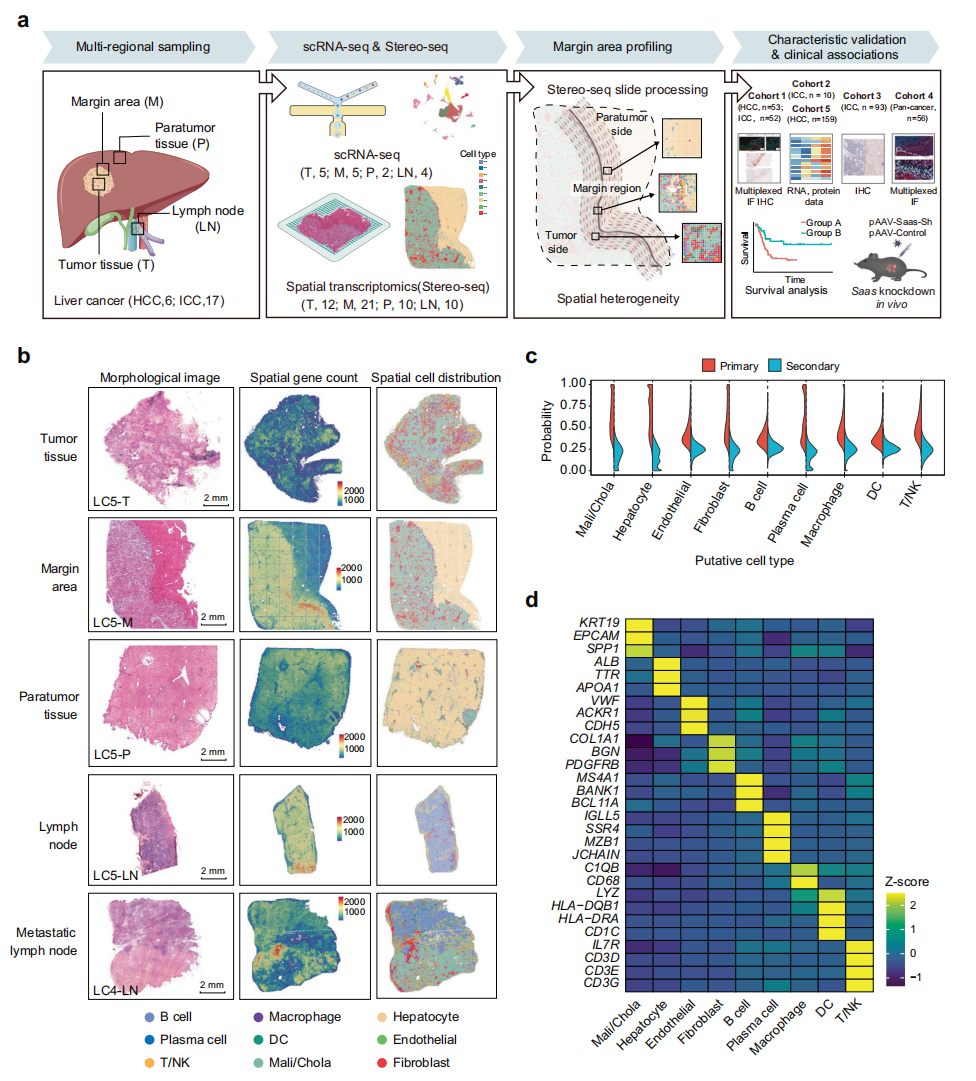
图一 人类原发性肝癌中多个区域位点的空间分辨转录组学分析[7]
接下来,研究者对肿瘤组织(T)、肿瘤边缘区域(M)、癌旁组织(P)和正常或转移的淋巴结(LN)四个区域的细胞组成和空间分布进行表征。结果显示,除肿瘤恶性细胞和肝细胞外,T/NK细胞、B细胞和成纤维细胞的分布比例较高(图二a)。根据H&E染色图像识别肿瘤边界,将边缘区域细分为肿瘤邻近部分(M-T)和癌旁部分(M-P)。进一步观察发现,肿瘤中内皮细胞和浆细胞多于其他区域,成纤维细胞和巨噬细胞更倾向于在M-T中富集,B细胞在LN中最为丰富(图二a和b)。空间上,免疫细胞如巨噬细胞主要聚集在肿瘤边界周围的边缘区域,表明边界周围有不同的免疫微环境(图二a和c)。分析不同区域的微环境,发现与肿瘤组织相比,M-T中高度富集上皮-间充质转化(EMT)、TME(缺氧、血管生成)、细胞凋亡、糖酵解、氧化磷酸化、免疫反应相关通路;与癌旁组织相比,M-P中参与PI3K/AKT/mTOR信号传导、IL6/JAK/STAT3信号传导、脂质代谢(脂质生成和胆固醇稳态)的通路富集程度更高,而脂肪酸代谢通路富集更低;LN中干扰素-γ反应、细胞因子-受体相互作用相关的通路富集程度最高(图二d)。以上数据表明,这四个不同区域的细胞具有高度异质性,肿瘤边缘区域是一个复杂区域,表现出低氧、显著炎症反应和高度免疫逃逸的特征。
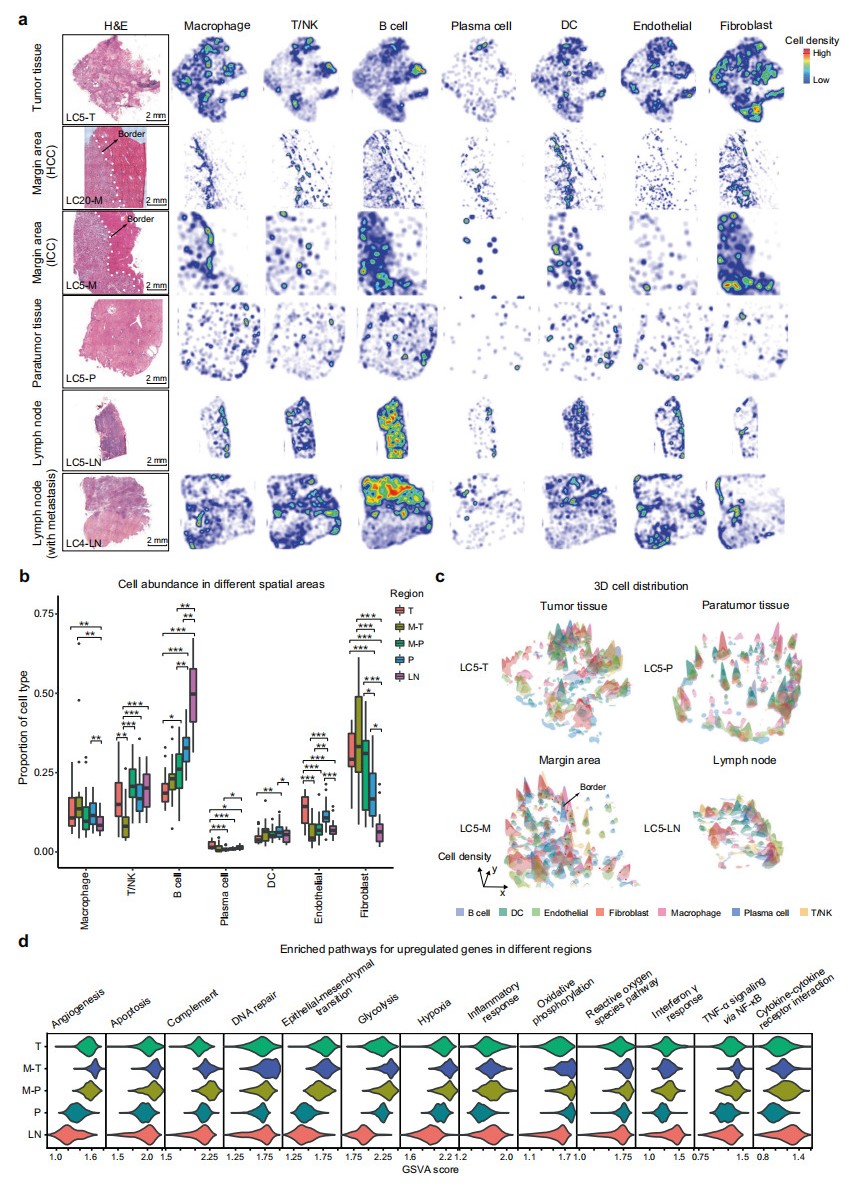
图二 人类原发性肝癌的组织空间异质性[7]
随后,研究者对肿瘤边缘区域的局部微环境进行全面分析,采用新的空间扫描和数字化模型(SDM)进一步研究肿瘤边界两侧的特征(图三a)。基于Stereo-seq数据,研究者将边缘区域精确分割为多层,每层代表距离边界250 µm宽的区域,分析边界的癌旁侧和肿瘤侧共六层以及更远区域的细胞组分和特征(图三a)。与癌旁侧相比,肿瘤侧的免疫细胞和成纤维细胞显著富集;免疫细胞尤其是在最靠近肿瘤侧边界的第一层内显著富集,占所有细胞组分的30%以上(图三b)。在检测到的免疫细胞中,与癌旁侧相比,巨噬细胞、DC、T/NK细胞和B细胞在最靠近肿瘤侧边界的区域都更为丰富(图三b和c),细胞比例依次从肿瘤侧的第三层(−750 µm至−500 µm)、第二层(−500 µm至−250 µm)和第一层(−250 µm至0 µm)逐渐增加(图三b)。验证队列1的原发性癌症患者样本边缘区域的多重免疫荧光(IF)结果显示,CD68+巨噬细胞在肿瘤侧的第一层中显著富集(图三c和d),验证了上述发现。免疫检查点基因包括CTLA4、CD96和TIGIT等的表达水平,也在肿瘤侧的第一层中高表达(图三e),表明该区域免疫细胞的免疫抑制状态升高。与癌旁侧相比,第一层肿瘤细胞也表现出增强的免疫逃逸信号,较低的缺氧反应,较高的糖酵解水平和增殖能力(图三f和g)。与其他肿瘤侧细胞相比,第一层肿瘤细胞在脂肪酸代谢相关的途径上调,包括脂肪酰基辅酶A合成和脂肪酸β氧化(图三g)。与其他癌旁侧细胞相比,癌旁第一层细胞的凋亡、血管生成、增殖能力、缺氧和糖酵解相关的途径显著富集(图三f和g),表明该区域富集了严重受损的肝细胞。基于上述数据,研究者以肿瘤边界为中心的500 µm宽的区域定义为一个独特的肿瘤侵袭前沿区,其特征是明显的免疫抑制环境,脂肪酸代谢的能量供应增强,肿瘤细胞的EMT能力增强,以及肝细胞严重受损。
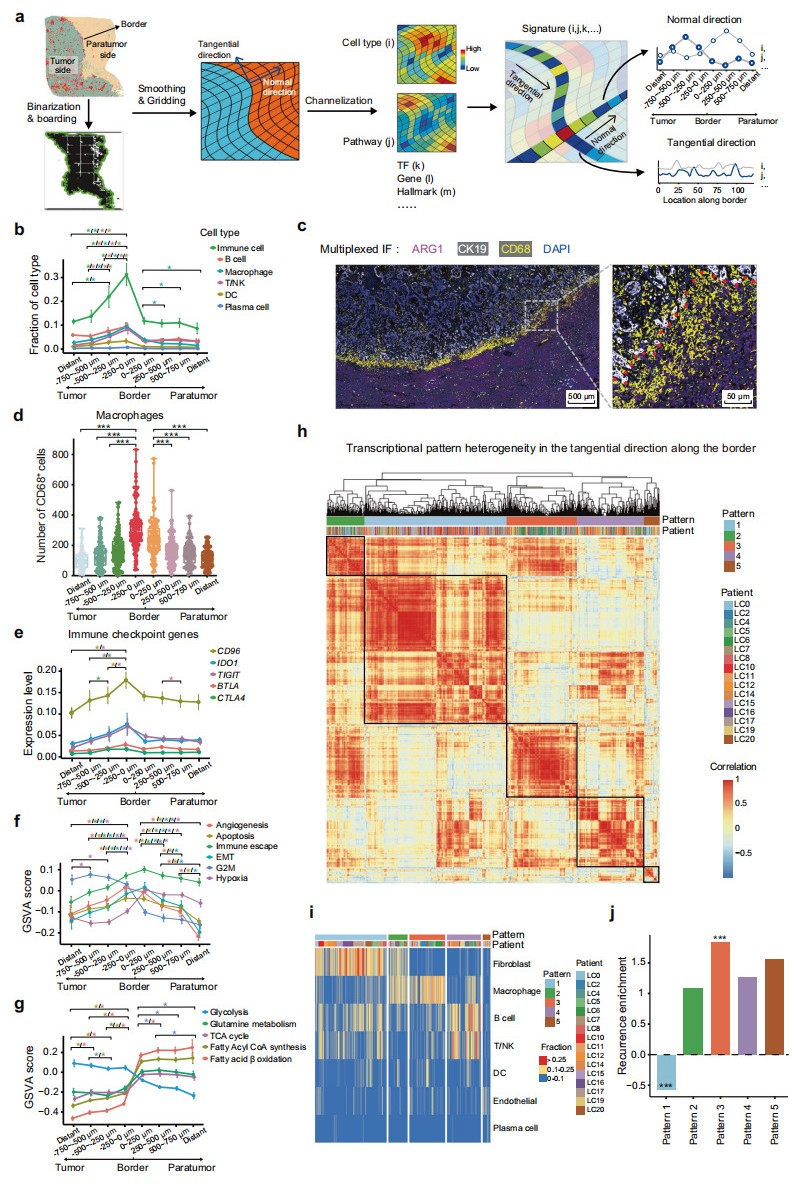
图三 肿瘤边缘区域的微环境表征[7]
基于Stereo-seq数据,研究者进一步将侵袭前沿区分为100个相等的亚区,提取所有亚区中除恶性细胞、胆管细胞和肝细胞外的细胞组分,探究亚区之间的相似性(图三h)。根据亚区之间的细胞组成,分为五种模式(图三h和i)。其中巨噬细胞占主导的模式3在肿瘤复发患者中显著富集,表明侵袭前沿区的巨噬细胞可能对肿瘤进展至关重要;而成纤维细胞占主导的模式1主要富集在无复发的病人中(图三i和j)。
先前有研究报道,肝细胞的炎症反应可能导致肝转移[10]。于是,研究者进一步表征癌旁肝细胞的炎症反应及其在肿瘤侵袭中的作用。将肝细胞从癌旁侧的三层中分离出来,进一步鉴定出两种肝细胞亚型,其中Hep1细胞在最靠近肿瘤旁边界的250 µm宽的区域内显著积累,表现出较高的SAA1和SAA2表达水平(图四a和b)。验证队列2的RNA-seq数据也显示,边界区域周围的SAA1和SAA2的表达水平高于相应的肿瘤组织和癌旁组织(图四c)。Stereo-seq和scRNA-seq数据交集显示Hep1细胞共有340个特异性高表达基因,包括SAA1和SAA2(图四d)。这些上调的基因主要富集在补体和凝血级联反应、细胞凋亡和胆固醇代谢相关通路(图四e),表明Hep1细胞存在先天免疫相关反应和肝细胞损伤。
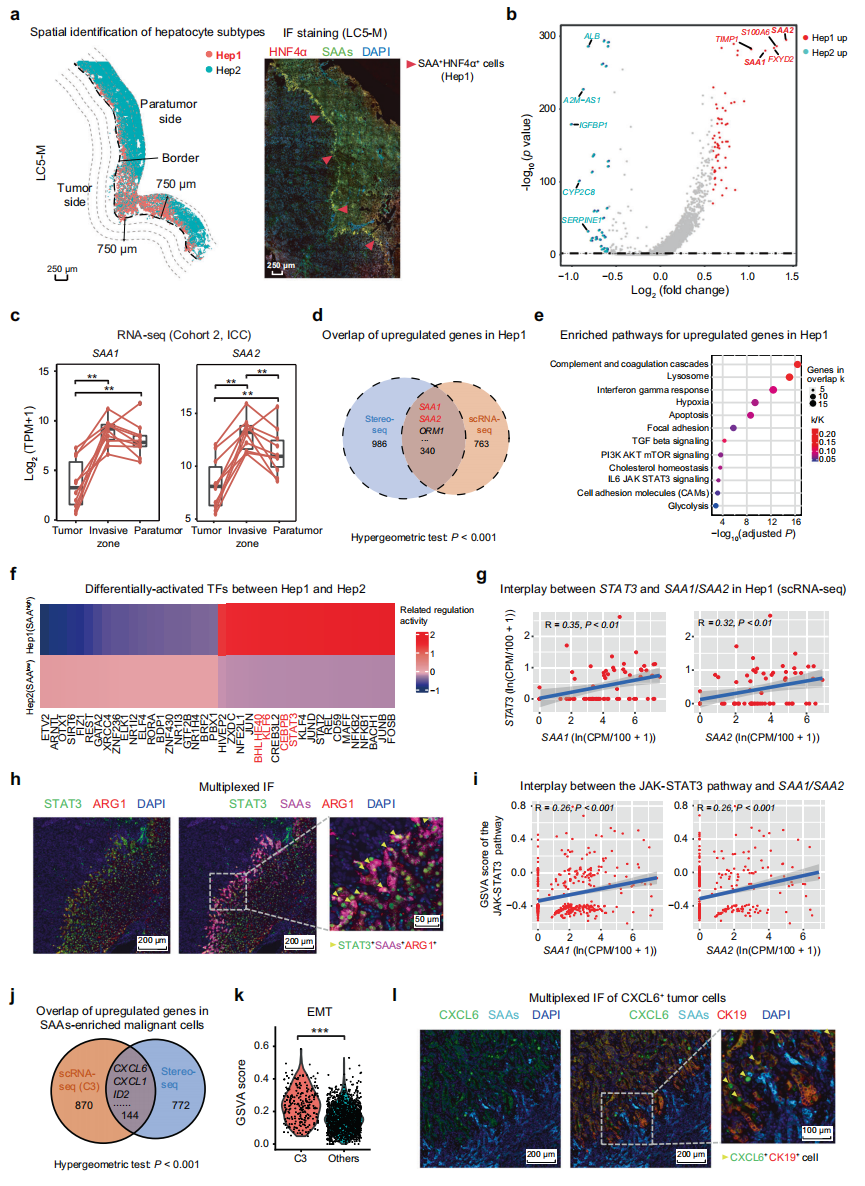
图四 侵袭前沿区JAK-STAT3激活诱导肝细胞高表达SAAs[7]
接下来,研究者探索SAA1和SAA2在Hep1细胞中高表达的机制。经单细胞调节网络推断和聚类(SCENIC)分析,发现多种转录因子在Hep1中的活性高于Hep2(图四f),且仅在Hep1中观察到STAT3和SAA1或SAA2之间呈显著正相关(图四g)。验证队列4的多重IF进一步证实侵袭前沿区SAAs+肝细胞中STAT3的表达更高(图四h)。在Hep1中,SAA1或SAA2表达水平与JAK-STAT3通路的基因集变异分析(GSVA)评分之间也存在正相关(图四i)。进一步,研究者分析靠近Hep1富集区域和其余区域的恶性细胞之间的差异基因表达,鉴定出一个具有与肿瘤细胞相似基因图谱的肿瘤细胞亚群3,主要来自Hep1附近的边缘区域。在scRNA-seq与Stereo-seq交集数据中,发现亚群3有144个基因特异性上调,如CXCL6、ITGAV、ID2等,主要富集在EMT、炎症反应和脂肪酸代谢相关通路中(图四j和k)。多重IF显示CXCL6+肿瘤细胞位于侵袭前沿区SAAs+肝细胞附近,高表达STAT3(图四h和l)。以上数据表明,肝肿瘤细胞可以通过分泌CXCL6,激活侵袭前沿区JAK-STAT3通路来诱导Hep1肝细胞中SAAs的过表达。
接下来,研究者探索SAAs+的Hep1肝细胞在侵袭前沿区的功能。基于边缘区域的scRNA-seq数据,并使用CellPhoneDB观察配体-受体对,进而分析细胞间的相互作用。结果显示,在肝细胞和巨噬细胞之间检测到最强的双向通讯,有超过25对显著的配体-受体对(图五a),特别是SAA1-SAAs受体(图五b)。SAAs受体基因,包括FPR1、FPR2、TLR4、TLR2、SCARB1和CD36,在巨噬细胞中富集(图五c)。这些巨噬细胞主要来源于边缘区域(图五d),在侵袭前沿区的Hep1亚型细胞附近积累,特别是肿瘤侧的第一层(图五e和f)。验证队列4的多重IF显示FPR1+巨噬细胞位于侵袭前沿区中靠近SAAs+肝细胞的位置(图五g和h);侵袭前沿区的FPR1+巨噬细胞和SAAs+肝细胞显著增多,且两种细胞数量之间具有很强的相关性(图五i和j)。以上数据表明,SAAs+肝细胞有助于细胞通讯并招募巨噬细胞。
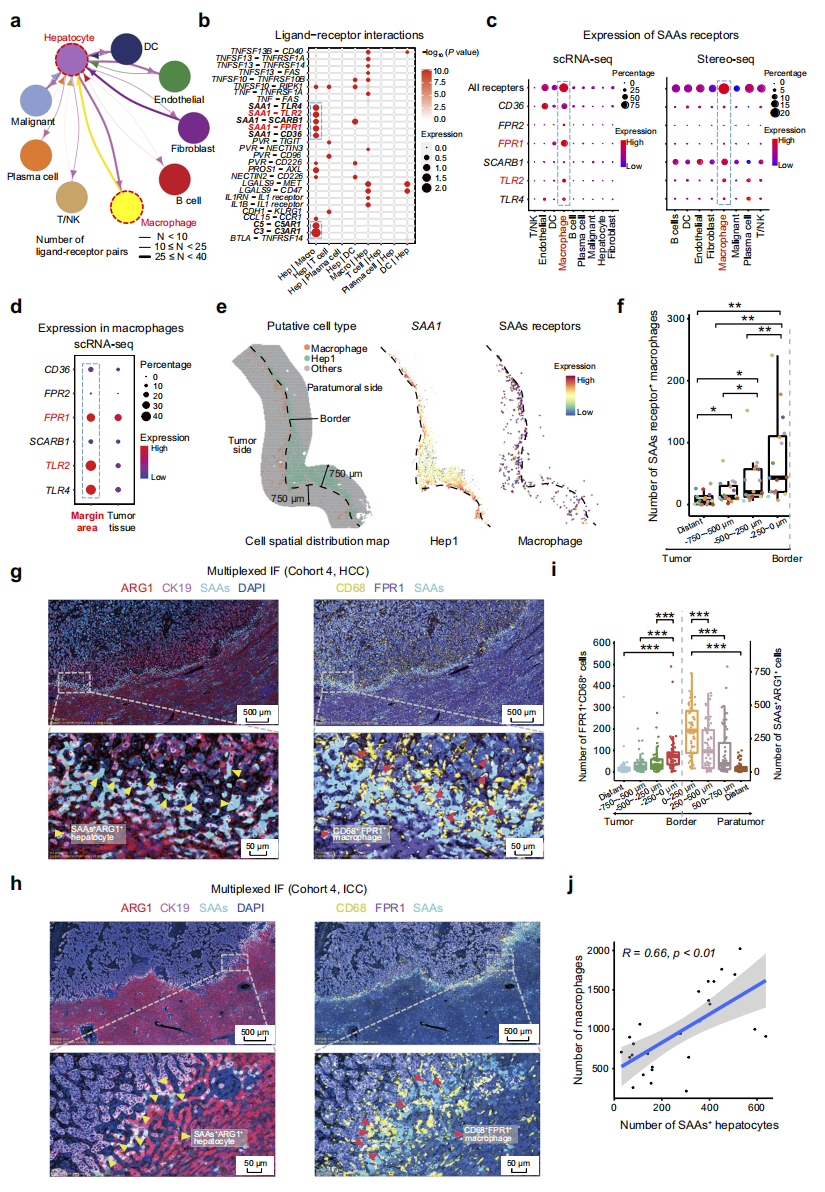
图五 Hep1肝细胞通过高表达SAAs介导细胞通讯并招募巨噬细胞[7]
研究者进一步分析配体-受体相互作用,发现恶性细胞可以通过侵袭前沿区中的CSF1-CSF1R或CSF1-SIRPA相互作用,直接将巨噬细胞极化为M2样表型(图六a)。在侵袭前沿区的巨噬细胞中,CD163、MRC1和SAAs受体TLR2以及多种细胞因子和趋化因子的表达上调,表现为M2样表型;而在肿瘤组织中的巨噬细胞则表现出ECM重塑,MMP9和MMP14的表达增加(图六b)。进而,研究者探究侵袭前沿区受损肝细胞和被招募巨噬细胞的临床意义。发现验证队列3患者较差的总生存期(OS)与边缘区域SAA1和SAA2表达水平的增加显著相关(图六c);侵袭前沿区SAAs+肝细胞的高比例表现出原发性癌症和继发性肝癌患者的较差OS(图六d)。在原位移植肝肿瘤小鼠模型实验中发现,Saa1和Saa2的特异性敲降显著抑制体内原发性和继发性肝肿瘤的生长(图六e和f)。此外,肝细胞中Saas的敲降显著降低了SAAs的表达,并减少了其随后在侵袭前沿区对巨噬细胞的招募及其M2极化(图六g-i)。以上数据表明,巨噬细胞被侵袭前沿区受损肝细胞分泌的SAAs特异性招募,并极化为M2表型。
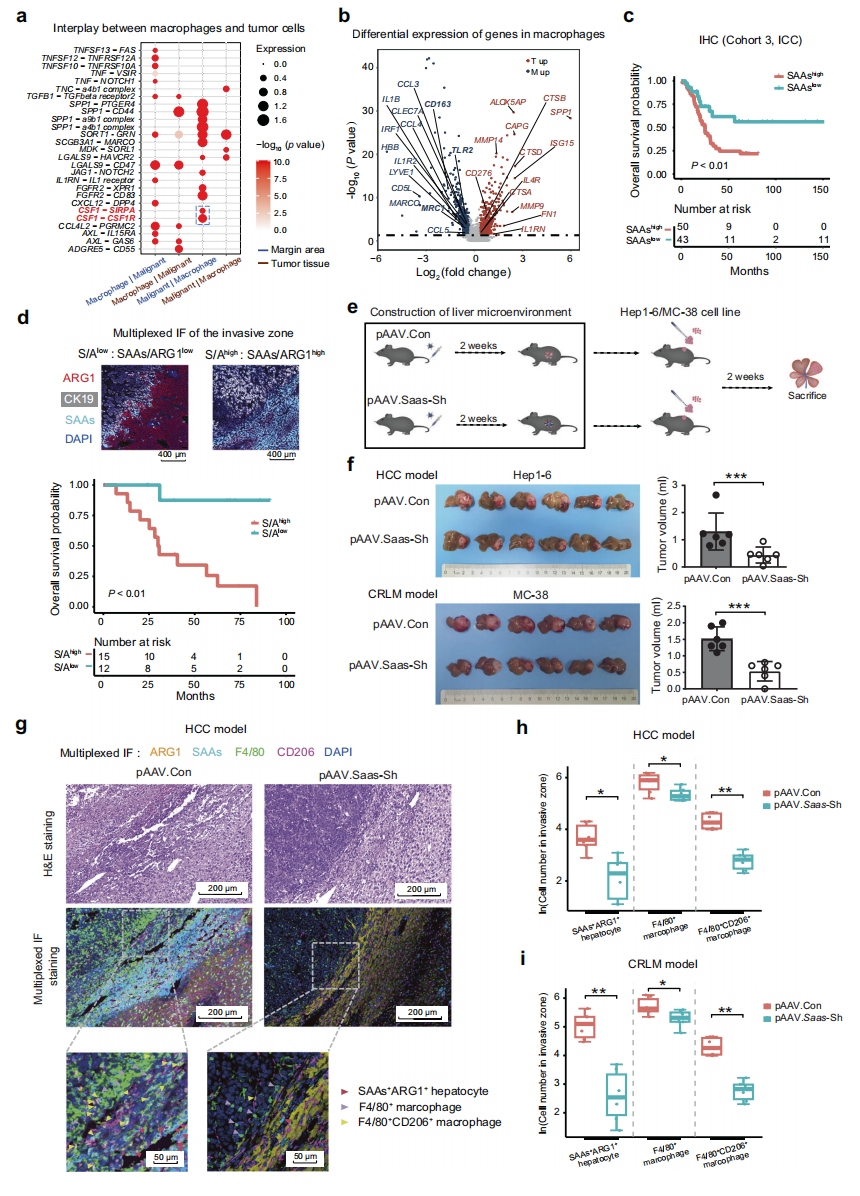
图六 侵袭前沿区受损肝细胞高表达SAAs促进了肝肿瘤的进展[7]
综上所述,研究者定义并表征了肝癌中以肿瘤边界为中心的500 µm宽的肿瘤侵袭前沿区。该区域是一个复杂和动态的生态系统,具有显著的免疫抑制、代谢重编程和严重受损的肝细胞。具体而言,肝脏肿瘤细胞分泌CXCL6激活Hep1肝细胞的JAK-STAT3通路,诱导其高表达SAAs;进而通过SAAs-FPR1促使SAAs+的Hep1肝细胞招募FPR1+巨噬细胞,并通过SAAs-TLR2诱导巨噬细胞M2极化,导致局部免疫抑制和肿瘤进展(图七)。该研究增强了人们对肝脏肿瘤侵袭和转移机制的理解,为肝癌和其他实体肿瘤的治疗提供了更精准的信息依据和新的干预角度。
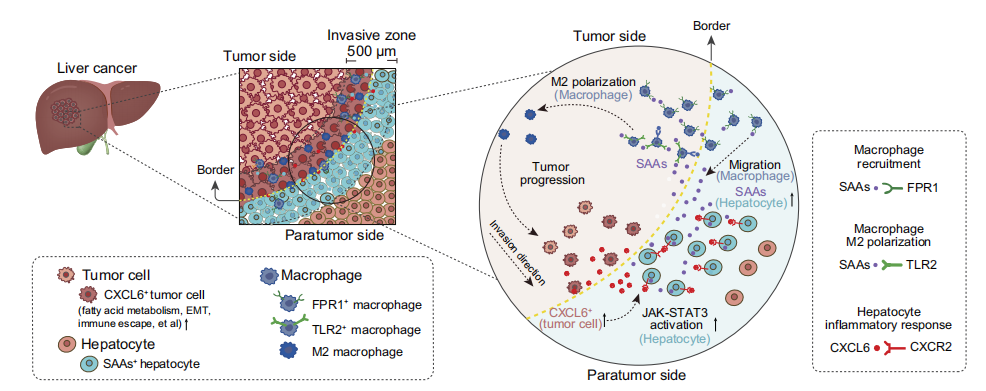
图七 肿瘤侵袭前沿区的生态系统示意图[7]

GTP研发中心拥有自主知识产权的“人造精子细胞”介导的半克隆技术,可在体外实现“人造精子细胞”的多位点改造并进行功能元件测试,最后将“人造精子细胞”通过卵母细胞注射可以一步法获得基因改造小鼠。基于“人造精子细胞”单倍体打靶高效率的优势,可快速构建定制化小鼠,如基因组安全位点大片段敲入、条件性基因敲除/敲入和人源化基因改造等。多个复杂调控元件可以通过多次体外细胞打靶构建在同一株“人造精子细胞”中,经细胞水平质控后,一步法获得基因改造小鼠。

如有需要欢迎联系我们,GTP研发中心将竭诚为您设计最佳方案,制定专属于您的基因改造小鼠。
网址:http://www.sibcb.ac.cn/gtp/
地址:上海市岳阳路320号
责任编辑:甜点
新媒体运营:suway
参考文献
1. Zheng L, Qin S, Si W, Wang A, Xing B, Gao R, Ren X, Wang L, Wu X, Zhang J et al: Pan-cancer single-cell landscape of tumor-infiltrating T cells. Science 2021, 374(6574):abe6474.
2. Donne R, Lujambio A: The liver cancer immune microenvironment: Therapeutic implications for hepatocellular carcinoma. Hepatology 2023, 77(5):1773-1796.
3. Saviano A, Henderson NC, Baumert TF: Single-cell genomics and spatial transcriptomics: Discovery of novel cell states and cellular interactions in liver physiology and disease biology. Journal of hepatology 2020, 73(5):1219-1230.
4. Longo SK, Guo MG, Ji AL, Khavari PA: Integrating single-cell and spatial transcriptomics to elucidate intercellular tissue dynamics. Nat Rev Genet 2021, 22(10):627-644.
5. Sun Y, Wu L, Zhong Y, Zhou K, Hou Y, Wang Z, Zhang Z, Xie J, Wang C, Chen D et al: Single-cell landscape of the ecosystem in early-relapse hepatocellular carcinoma. Cell 2021, 184(2):404-421 e416.
6. Liu C, Li R, Li Y, Lin X, Zhao K, Liu Q, Wang S, Yang X, Shi X, Ma Y et al: Spatiotemporal mapping of gene expression landscapes and developmental trajectories during zebrafish embryogenesis. Dev Cell 2022, 57(10):1284-1298 e1285.
7. Wu L, Yan J, Bai Y, Chen F, Zou X, Xu J, Huang A, Hou L, Zhong Y, Jing Z et al: An invasive zone in human liver cancer identified by Stereo-seq promotes hepatocyte-tumor cell crosstalk, local immunosuppression and tumor progression. Cell Res 2023, 33(8):585-603.
8. Zhang M, Yang H, Wan LF, Wang ZH, Wang HY, Ge C, Liu YH, Hao YJ, Zhang DD, Shi GN et al: Single-cell transcriptomic architecture and intercellular crosstalk of human intrahepatic cholangiocarcinoma. J Hepatol 2020, 73(5):1118-1130.
9. Elosua-Bayes M, Nieto P, Mereu E, Gut I, Heyn H: SPOTlight: seeded NMF regression to deconvolute spatial transcriptomics spots with single-cell transcriptomes. Nucleic acids research 2021, 49(9):e50.
10. Lee JW, Stone ML, Porrett PM, Thomas SK, Komar CA, Li JH, Delman D, Graham K, Gladney WL, Hua X et al: Hepatocytes direct the formation of a pro-metastatic niche in the liver. Nature 2019, 567(7747):249-252.



