技术分享:化学激活的ciCas9开关适用于多种Cas9效应器的基因编辑时间调控
CRISPR-Cas9是一种高效的基因组编辑工具,Cas9核酸内切酶在向导RNA(sgRNA)的引导下可以靶向基因组的任何位置[1],形成DNA双链断裂(DSB),损伤后修复可造成插入或缺失等,从而实现对基因组DNA的定向改造[2]。现已有多种工程化的Cas9效应器被开发用于特定的研究与应用。例如将dCas9与转录激活因子融合以调节基因表达,与染色质修饰因子融合靶向表观基因组编辑;将nCas9与DNA脱氨酶融合产生的胞嘧啶碱基编辑器(CBE)可实现C-to-T碱基转换,以及腺嘌呤碱基编辑器(ABE)可实现A-to-G碱基转换;先导编辑器(PE)可以使用RNA模板进行精确的DNA编辑。诱导型Cas9变体可以针对DSB的产生和随后的DNA编辑提供精准的时间调控[3],进一步丰富应用场景,如采用时间调控的nCas9和dCas9效应器来控制DNA转录、染色质修饰[4]和碱基编辑[5]。然而,这些时间调控的Cas9效应器系统往往比较复杂,需要通过筛选分裂酶等进行优化,不能直接用于所有的效应器,较为费时费力。基于所有Cas9效应器的功能发挥都需要结合DNA,因此针对其DNA结合属性而设计的时间调控元件,极有可能推广至所有的Cas9效应器。
2023年3月,Nature Chemical Biology期刊上报道了一种通用型、化学快速激活调控的单组分Cas9变体——ciCas9开关。研究者首先开发出化学调控的CRISPR转录激活系统,并证明ciCas9开关是通过调控DNA靶位点的结合而起作用的。进而研究者将其推广至多种Cas9效应器,开发出不同的化学调控Cas9效应器,包括有两种胞嘧啶碱基编辑器,两种腺嘌呤碱基编辑器,一种双碱基编辑器和一种先导编辑器,并证明这些效应器可以在数小时内实现高效率的精准碱基编辑[6]。
研究者之前已开发出一种单组分化学调控的Cas9变体ciCas9,包含有一个紧密的自抑制开关,可以被小分子快速激活实现基因编辑的时间调控[7]。其原理是用Bcl-xL蛋白取代Cas9的REC2结构域,并将BH3肽融合到C末端。Bcl-xL和BH3可相互作用形成紧密的分子内复合物,从而抑制Cas9的活性(图一a)。自抑制的ciCas9具有低活性,在小分子A-1155463(简称为A115)的作用下,Bcl-xL和BH3之间的相互作用被破坏,促使ciCas9在几分钟内以剂量依赖的方式产生DSB,激活编辑活性[7]。基于此,研究者首先测试ciCas9是否通过调节DNA靶位点结合来实现其化学调控开关作用的。采用CRISPR转录激活的方式进行验证,将转录激活因子Vp64-p65-Rta(VPR)与失活的ciCas9(dciCas9)相融合,测试其激活CXCR4表达的能力(图一b)。结果显示,在A115存在的情况下,dciCas9-VPR激活了CXCR4的表达(图一c),支持了dciCas9的自抑制机制是基于DNA结合属性。且dciCas9-VPR的转录激活具有A115的剂量依赖性(图一d)。以上数据表明,ciCas9开关的化学调控是通过调节DNA结合来实现的。
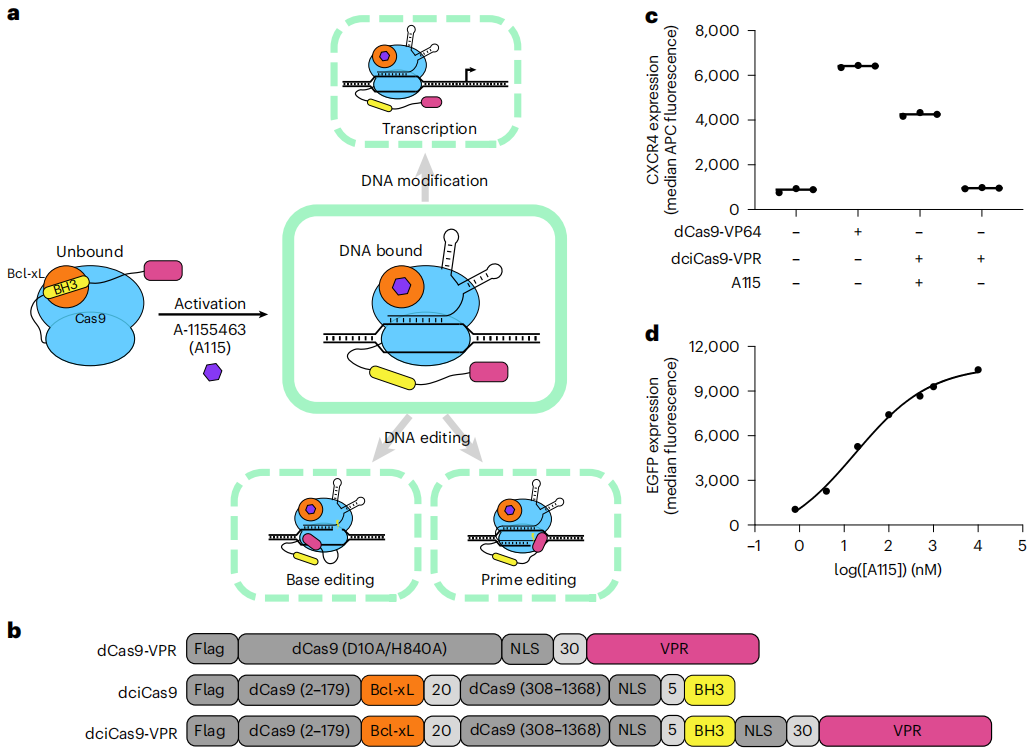
图一 ciCas9开关可用于开发化学调控的Cas9效应器[6]
为进一步推广ciCas9开关的应用,研究者将BE4max或AncBE4max脱氨酶与具有切口酶活性的nciCas9相融合开发出化学调控的胞嘧啶碱基编辑器,保留了原始结构域排列,并分别测试编辑效率(图二a)。首先测试传统BE4max和AncBE4max的编辑效率,结果显示两者在EMX1和HEK3两个靶位点均显示出高效的编辑(图二b和c)。随后,测试化学调控的胞嘧啶碱基编辑器,结果显示在A115存在时编辑器开关被完全激活,但不激活的对照组也具有高背景活性(图二d和e)。为了降低背景,研究者使用更高亲和力的BH3肽变体F22,使得nciCas9具有更强的自抑制作用。测试结果显示,两种新的化学调控胞嘧啶碱基编辑器都表现出较低的背景编辑,且A115诱导激活的编辑效果没有明显降低(图二f和g)。此外,化学调控的ciBE4max在另外两个靶位点ABE9和HEK2的测试中也具有高基因编辑效率和低背景(图二h和i)。于是,研究者将这两种F22变体的化学调控胞嘧啶碱基编辑器称为ciBE4max和ciAncBE4max,两者在靶位点的碱基编辑方式与传统的BE4max和AncBE4max相似(图二j和k)。以上数据表明,nciCas9可以直接替代nCas9,通过简单地融合脱氨酶即可开发出高效且低背景的化学调控胞嘧啶碱基编辑器。
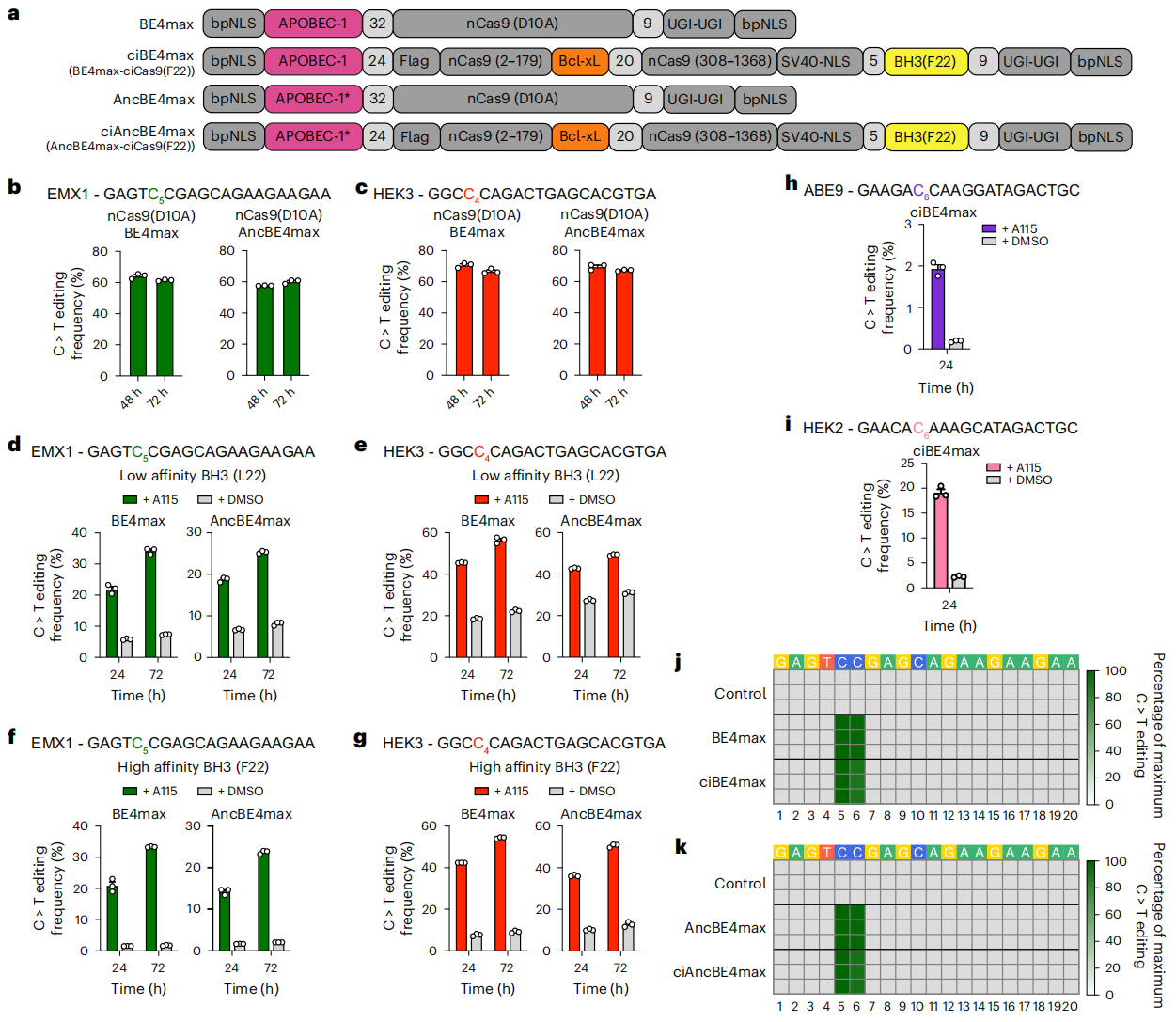
图二 ciCas9开关可用于开发化学调控的胞嘧啶碱基编辑器[6]
同样地,研究者将nciCas9与ABEmax或ABE8e脱氨酶相融合,设计出化学调控的腺嘌呤碱基编辑器,并测试编辑效率(图三a)。结果显示,传统的ABEmax和ABE8e实现了高效的基因编辑(图三b和c),两种化学调控的腺嘌呤碱基编辑器在A115激活下也能实现高效的基因编辑(图三d-g),高亲和力的F22变体减少了其在ABE16、ABE9和HEK2靶位点的背景(图三d,f和g),但在HEK3靶位点上仍有高背景(图三e),提示其高背景不是由nciCas9造成的。研究者将携带F22变体的化学调控腺嘌呤碱基编辑器命名为ciABEmax和ciABE8e,与传统的ABEmax和ABE8e有着相似的碱基编辑方式(图三h和i)。以上数据表明,ciCas9开关可用于开发化学调控的腺嘌呤碱基编辑器。
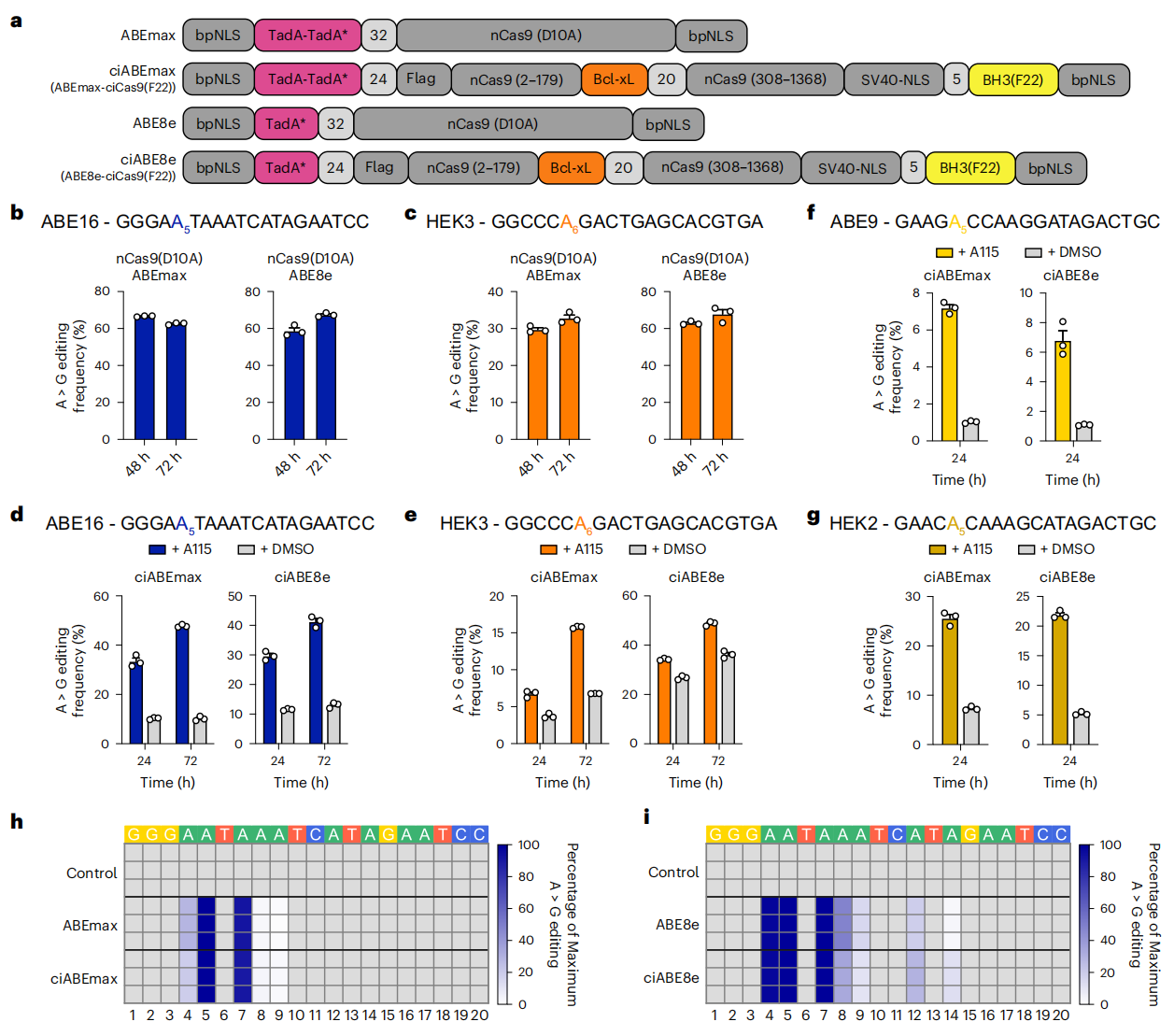
图三 ciCas9开关可用于开发化学调控的腺嘌呤碱基编辑器[6]
最后,研究者将ciCas9开关应用于双碱基编辑器以及先导编辑器。SPACE是一种A-to-G和C-to-T双碱基编辑器,由两个脱氨酶结构域与nCas9融合而成[8]。研究者基于ciCas9开关开发了SPACE的化学调控变体ciSPACE(图四a),在A115诱导后ciSPACE实现了预期的双碱基编辑,且具有低背景(图四b-e)。此外,研究者将nciCas9应用于PE2先导编辑器。PE2是由融合了M-MLV逆转录酶的nCas9(H840A)变体与pegRNA/sgRNA结合使用,实现碱基替换和小片段的插入或缺失[9]。化学调控的ciPE2同样保留原始的结构域排列(图四a)。使用升级版ciPE3b进行测试,结果显示在A115激活下同样具有高效的基因编辑效率和低背景(图四f)。以上数据表明,ciCas9开关可以直接应用于不同变体的Cas9效应器。
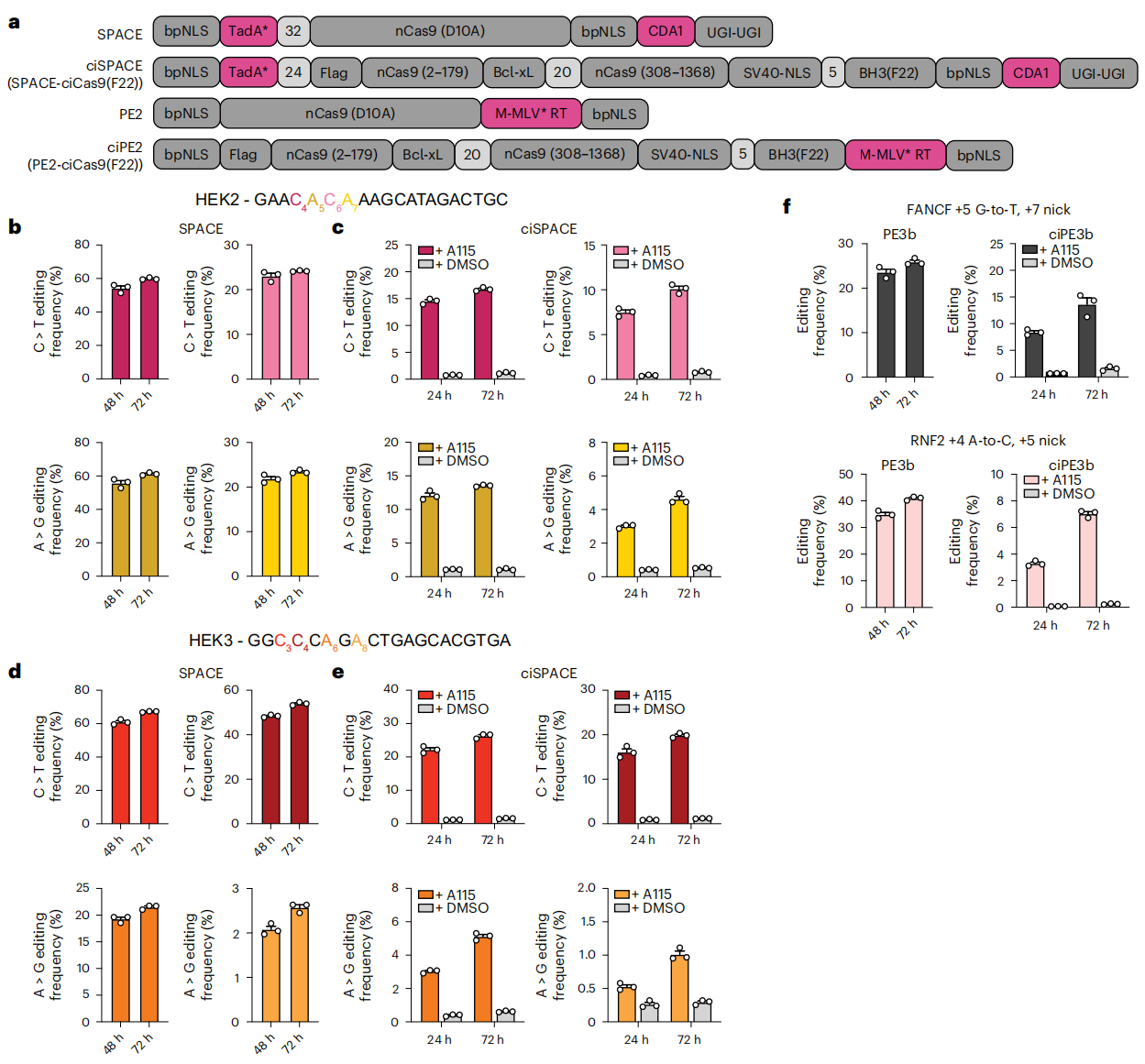
图四 ciCas9开关可用于开发化学调控的双碱基编辑器和先导编辑器[6]
综上所述,研究者针对DNA结合属性设计开发的化学调控ciCas9开关,具有广泛的适用性和推广性。利用ciCas9,研究者开发出7种Cas9效应器用于化学调控的基因编辑和转录激活。此外,研究者利用这些化学调控的碱基编辑器分析编辑动力学,发现基因编辑发生在数小时内,并且核苷酸的快速早期编辑可预测最终的编辑结果,同样靶位点的首选核苷酸编辑也增加了脱靶旁观者编辑的频率。ciCas9开关为开发化学调控的Cas9效应器提供了一种简单、通用的方法,为碱基编辑动力学研究提供了精确的时间控制方法。

GTP研发中心拥有自主知识产权的“人造精子细胞”介导的半克隆技术,可在体外实现“人造精子细胞”的多位点改造并进行功能元件测试,最后将“人造精子细胞”通过卵母细胞注射可以一步法获得基因改造小鼠。基于“人造精子细胞”单倍体打靶高效率的优势,可快速构建定制化小鼠,如基因组安全位点大片段敲入、条件性基因敲除/敲入和人源化基因改造等。多个复杂调控元件可以通过多次体外细胞打靶构建在同一株“人造精子细胞”中,经细胞水平质控后,一步法获得基因改造小鼠。

如有需要欢迎联系我们,GTP研发中心将竭诚为您设计最佳方案,制定专属于您的基因改造小鼠。
网址:http://www.sibcb.ac.cn/gtp/
地址:上海市岳阳路320号
责任编辑:甜点
新媒体运营:suway
参考文献
1. Cong L, Ran FA, Cox D, Lin S, Barretto R, Habib N, Hsu PD, Wu X, Jiang W, Marraffini LA et al: Multiplex genome engineering using CRISPR/Cas systems. Science 2013, 339(6121):819-823.
2. Anzalone AV, Koblan LW, Liu DR: Genome editing with CRISPR-Cas nucleases, base editors, transposases and prime editors. Nature biotechnology 2020, 38(7):824-844.
3. Gangopadhyay SA, Cox KJ, Manna D, Lim D, Maji B, Zhou Q, Choudhary A: Precision Control of CRISPR-Cas9 Using Small Molecules and Light. Biochemistry-Us 2019, 58(4):234-244.
4. Foight GW, Wang Z, Wei CT, Jr Greisen P, Warner KM, Cunningham-Bryant D, Park K, Brunette TJ, Sheffler W, Baker D et al: Multi-input chemical control of protein dimerization for programming graded cellular responses. Nature biotechnology 2019, 37(10):1209-1216.
5. Berrios KN, Evitt NH, DeWeerd RA, Ren D, Luo M, Barka A, Wang T, Bartman CR, Lan Y, Green AM et al: Controllable genome editing with split-engineered base editors. Nature chemical biology 2021, 17(12):1262-1270.
6. Wei CT, Popp NA, Peleg O, Powell RL, Borenstein E, Maly DJ, Fowler DM: A chemically controlled Cas9 switch enables temporal modulation of diverse effectors. Nature chemical biology 2023.
7. Rose JC, Stephany JJ, Valente WJ, Trevillian BM, Dang HV, Bielas JH, Maly DJ, Fowler DM: Rapidly inducible Cas9 and DSB-ddPCR to probe editing kinetics. Nat Methods 2017, 14(9):891-896.
8. Grunewald J, Zhou R, Lareau CA, Garcia SP, Iyer S, Miller BR, Langner LM, Hsu JY, Aryee MJ, Joung JK: A dual-deaminase CRISPR base editor enables concurrent adenine and cytosine editing. Nature biotechnology 2020, 38(7):861-864.
9. Anzalone AV, Randolph PB, Davis JR, Sousa AA, Koblan LW, Levy JM, Chen PJ, Wilson C, Newby GA, Raguram A et al: Search-and-replace genome editing without double-strand breaks or donor DNA. Nature 2019, 576(7785):149-157.



