技术分享:简单添加sgRNA胞嘧啶延伸可调控Cas9活性,提高基因编辑的有效性和安全性
CRISPR-Cas9系统实现了高效的基因组编辑[1]。然而,除了脱靶效应外,CRISPR-Cas9系统在哺乳动物细胞中的应用还存在着其他不良反应,例如p53的激活、严重DNA损伤造成的细胞毒性[2]、大片段靶向基因组缺失和染色体重排[3]等。利用CRISPR-Cas9基因编辑人类诱导性多能干细胞(hiPSCs)的过程中,由于DNA双链断裂(DSB)介导的p53激活可导致严重的细胞毒性和细胞周期阻滞,很难获得敲除或者同源定向修复(HDR)的克隆[4]。因此,精准调控Cas9的活性对于基因组编辑的有效性和安全性至关重要。抑制Cas9活性是提高其安全性的一种直接且有效的方法,例如抗Cas9蛋白、小分子抑制剂和寡核苷酸已被证明可以限制其活性[5, 6],采用光切割gRNA的Cas9核糖核蛋白(RNPs)也有助于调控Cas9的失活[7]。尽管这些方法已被证明可有效降低CRISPR-Cas9系统的基因组切割和编辑效率,但是它们能否提高安全性和适用性仍有待进一步探索与优化,以满足实际应用场景中的实用性。
2023年4月,Nature Biomedical Engineering期刊上报道了一种简便调控CRISPR-Cas9基因组编辑活性的新策略[C]sgRNA,即通过在传统sgRNA的5′ 末端添加胞嘧啶延伸来抑制Cas9编辑活性。该方法可与Cas12a以及CRISPR基因激活和干扰的系统相兼容。研究证明了短的胞嘧啶延伸可降低人类多能干细胞中p53的激活和细胞毒性,并在维持双等位基因编辑的同时增强了同源定向修复效率;而长的胞嘧啶延伸可降低靶标活性,但提高了单等位基因编辑的特异性和准确性。[C]sgRNA策略简便优化了CRISPR-Cas9技术,提高了基因编辑的有效性和安全性[8]。
首先,研究者构建了一个等位基因特异性插入/缺失监测系统AIMS,在同一对等位基因的相同编码区下游分别敲入P2A-tdTomato和P2A-Venus监测盒,从而根据荧光定位的变化来区分基因编辑的情况(图一a)。使用原始的P2A序列(P2A1)及其变体P2A2来设计测试了6个sgRNA(图一b)。研究者在小鼠胚胎干细胞(mESC)的膜蛋白Cdh1和转录因子Tbx3等位基因上构建AIMS,采用P2A1-sgRNA1进行编辑,出现了9种荧光模式的组合(图一a和c)。荧光模式与测序结果相一致,表明AIMS构建成功。为简化流程,研究者将表达sgRNA、Cas9和嘌呤霉素抗性构建在同一个质粒上,转染到AIMS细胞后进行嘌呤霉素药筛,发现约30%的细胞克隆出现嵌合模式,进而传代挑选单克隆进行后续分析(图一d)。6个sgRNA均在高于99.4%的mESC单克隆中诱导了双等位基因编辑(图一e)。当靶向内源性基因时,也能获得类似的结果(图一f)。在双等位基因编辑的单克隆中,并无明显的等位基因偏好性(图一g)。进一步测序分析tdTomato+/Venusindel和tdTomatoindel/Venus+杂合单克隆,发现AIMS的表型误差小于0.3%(图一h)。并与标准的T7E1分析方法(图一i)进行比较,发现AIMS比T7E1的测定结果更为准确(图一j)。
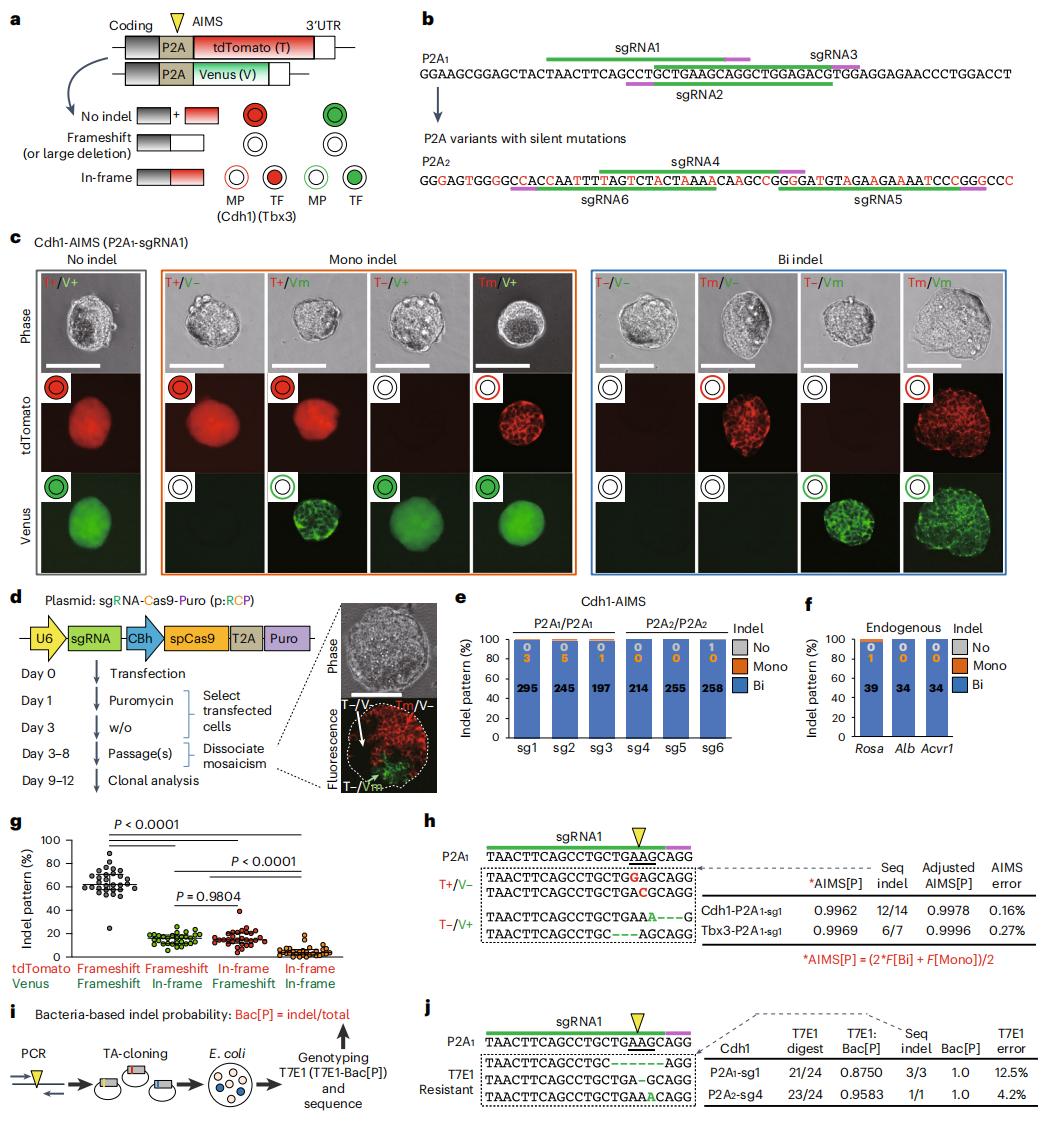
图一 开发可视化等位基因特异性基因组编辑监测系统AIMS [8]
接下来,研究者利用AIMS测试减少过度活性以提高单等位基因编辑的方法。发现减少转染的质粒量并不能提高单等位基因编辑(图二a)。据报道,在sgRNA间隔位点的5′ 端添加一些鸟嘌呤可阻断CRISPR-Cas9系统活性[9]。于是,研究者在sgRNA的5′ 端分别添加15个碱基的鸟嘌呤[15G]、胞嘧啶[15C]、腺嘌呤[15A]和胸腺嘧啶[15T](图二b),发现[15C]sgRNA显著增加了单等位基因编辑频率,[15T]sgRNA几乎不发生编辑(图二c)。进一步研究不同[C]延伸长度的影响,发现[C]sgRNA以延伸长度依赖的方式表现出双等位基因编辑减少和单等位基因编辑增加(图二d),且等位基因编辑无偏好性(图二e),对于不同sgRNA的影响有所不同(图二f),这是由于[C]延伸与有效的sgRNA-Cas9复合物相对浓度之间存在明确且相似的反比关系(图二g)。ANCOVA协方差分析发现[C]延伸的相对效应在sgRNA序列之间差异不大,说明它对不同sgRNA序列的影响是一致的(图二h)。以上数据表明,[C]sgRNA以延伸长度依赖的方式降低了有效sgRNA-Cas9复合物的相对浓度,进而调控了Cas9活性,这与sgRNA的序列无关。
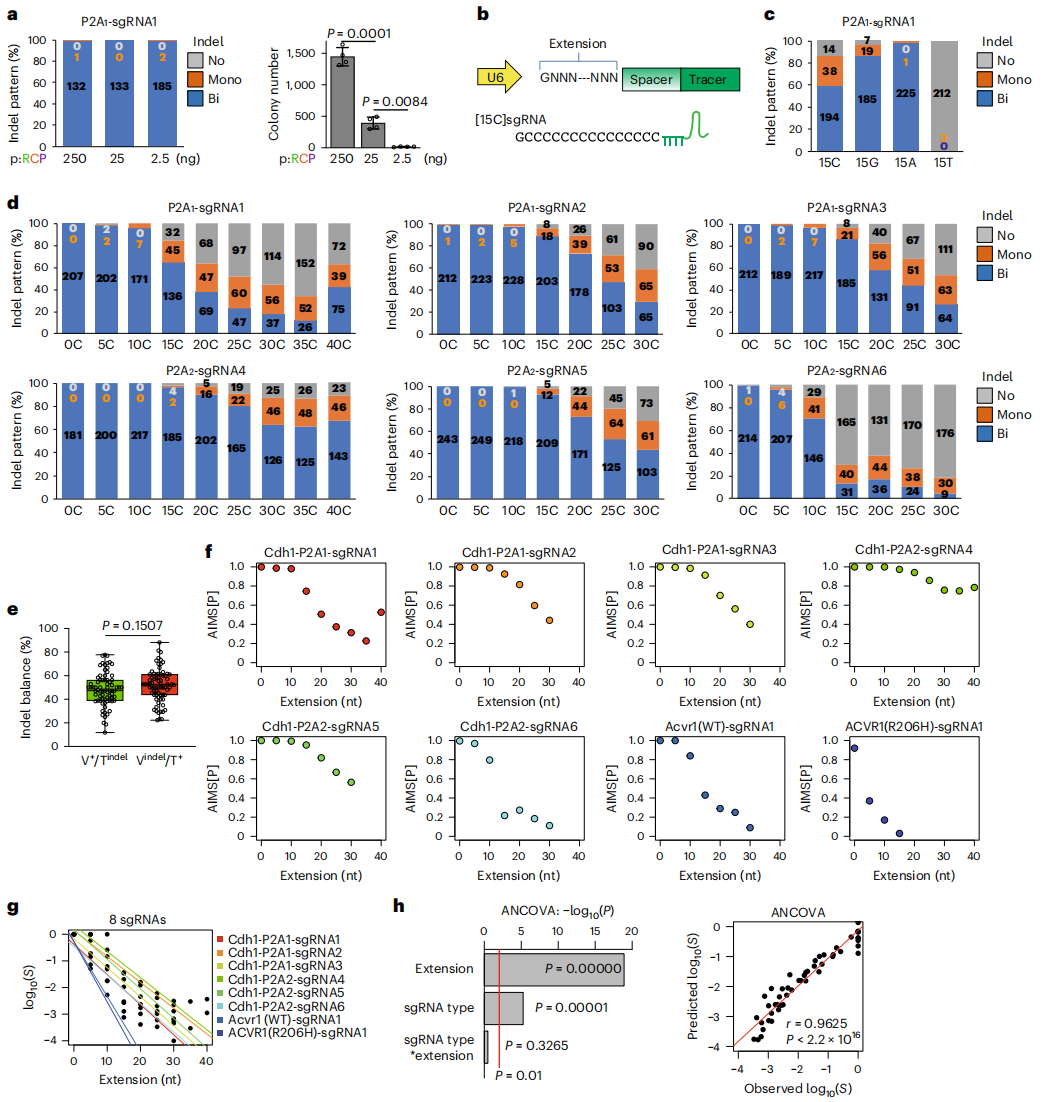
图二 通过在sgRNA 5′ 端添加[C]延伸来调控基因编辑频率[8]
为了验证[C]sgRNA的适用性,研究者在不同细胞系中进行测试。利用mESC获得Cdh1-AIMS的E12.5天小鼠胚胎,建立了成肝细胞系(HB-AIMS)进行基因编辑测试(图三a)。结果显示,与mESC-AIMS相比,随着[C]的延伸,HB-AIMS的Cas9活性迅速下降(图三a)。在人类脂肪干细胞(hADSC)的敲除实验中,[C]延伸同样诱导了单等位基因编辑(图三b)。另外,研究者将[C]sgRNA与其他抑制Cas9的方法进行比较。在共转染实验中,增加抗CRISPR蛋白AcrIIA4的质粒量可以抑制Cas9活性,但随之出现了细胞毒性(图三c);[C]sgRNA没有出现细胞毒性增加的现象,反而随着[C]延伸显著增加了细胞数量(图三d)。研究者还测试了[C]延伸与其他CRISPR工具的兼容性,发现[C]sgRNA可用于微调CRISPRa和CRISPRi系统的活性(图三e和f);当[C]延伸放在5′ 端时,同样可以随着[C]延伸的增加,抑制AsCpf1系统的活性(图三g)。以上数据表明,[C]sgRNA可便捷地应用于各种细胞系和各种CRISPR相关技术。
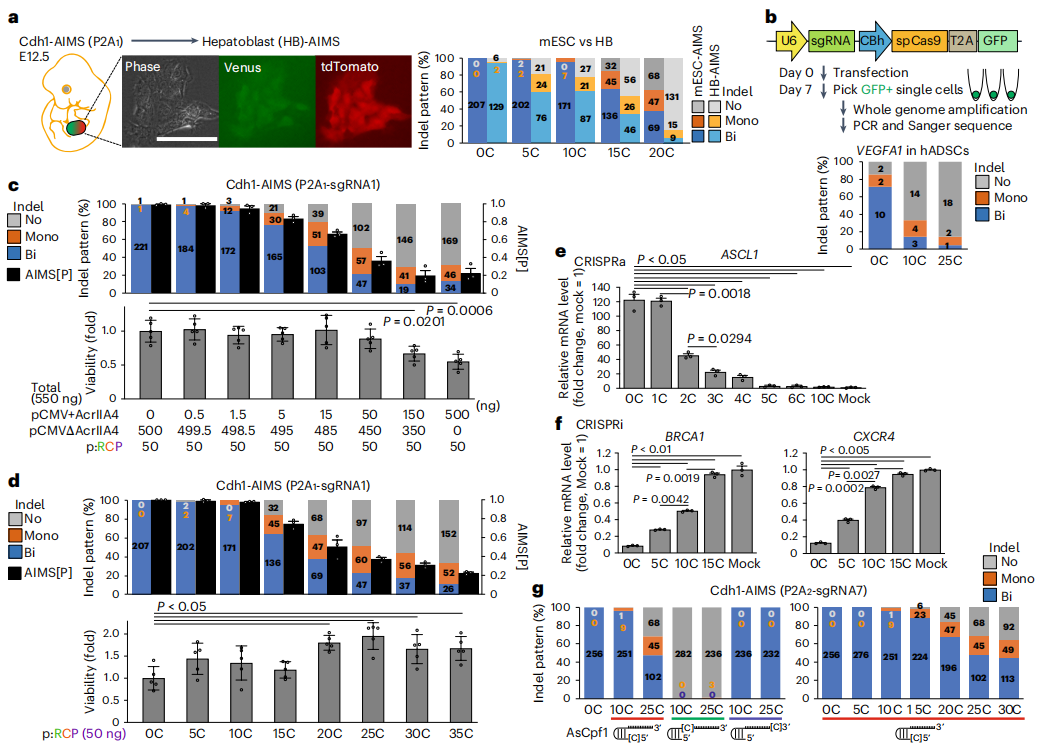
图三 [C]sgRNA策略的广泛应用性[8]
最后,研究者评估骨化性纤维发育不良(FOP)的基因治疗有效性,利用[C]sgRNA策略在FOP患者来源的hiPSC中进行R206H等位基因特异性校正(图四a)。T7E1测定证实了[0C]sgRNA的高效编辑活性以及[5C]-[20C]sgRNAs的编辑活性降低(图四b)。Bac[P]分析显示,WT等位基因在[5C]sgRNA的作用下比R206H等位基因的编辑频率下降得更多,校正后等位基因对二次编辑的敏感性也进一步降低(图四c)。[0C]sgRNA出现了严重的细胞毒性,p53在86%的存活细胞中被高度激活;相比之下,[5C]-[20C]sgRNAs显著缓解了细胞毒性和p53激活(图四d和e)。研究者测定了总体HDR和精确WT/校正的HDR频率,发现[5C]sgRNA总体HDR频率与[0C]sgRNA相当,并随着[C]延长而降低;[5C]和[10C]sgRNA可获得精确的WT/校正阳性克隆,但是[0C]无法获得(图四f)。在WT/校正的克隆中,由激活素-A介导的Smad1/5/8激活被阻断(图四g),表明该基因修复成功。研究者还对基因校正实验进行了计算建模,[0C]sgRNA和[5C]-[15C]sgRNA的单个等位基因HDR率分别为13.21%和26.93%(图四h),预测的总体HDR频率和WT/R206H HDR频率与实验结果密切相关(图四i)。计算模型估计了12种可能的HDR模式频率,当基因编辑频率高时,WT corrected indel/R206H_indel(组分12)和WT indel/R206H_Corrected_indel(组分6)占主导地位(图四j)。以上数据表明,要实现系统性地获得精确修复的杂合HDR克隆,需要多种机制来共同抑制Cas9活性,包括增强单等位基因编辑、抑制p53依赖的细胞毒性、提高HDR效率和抑制脱靶效应的二次HDR等位基因切割(图四k)。
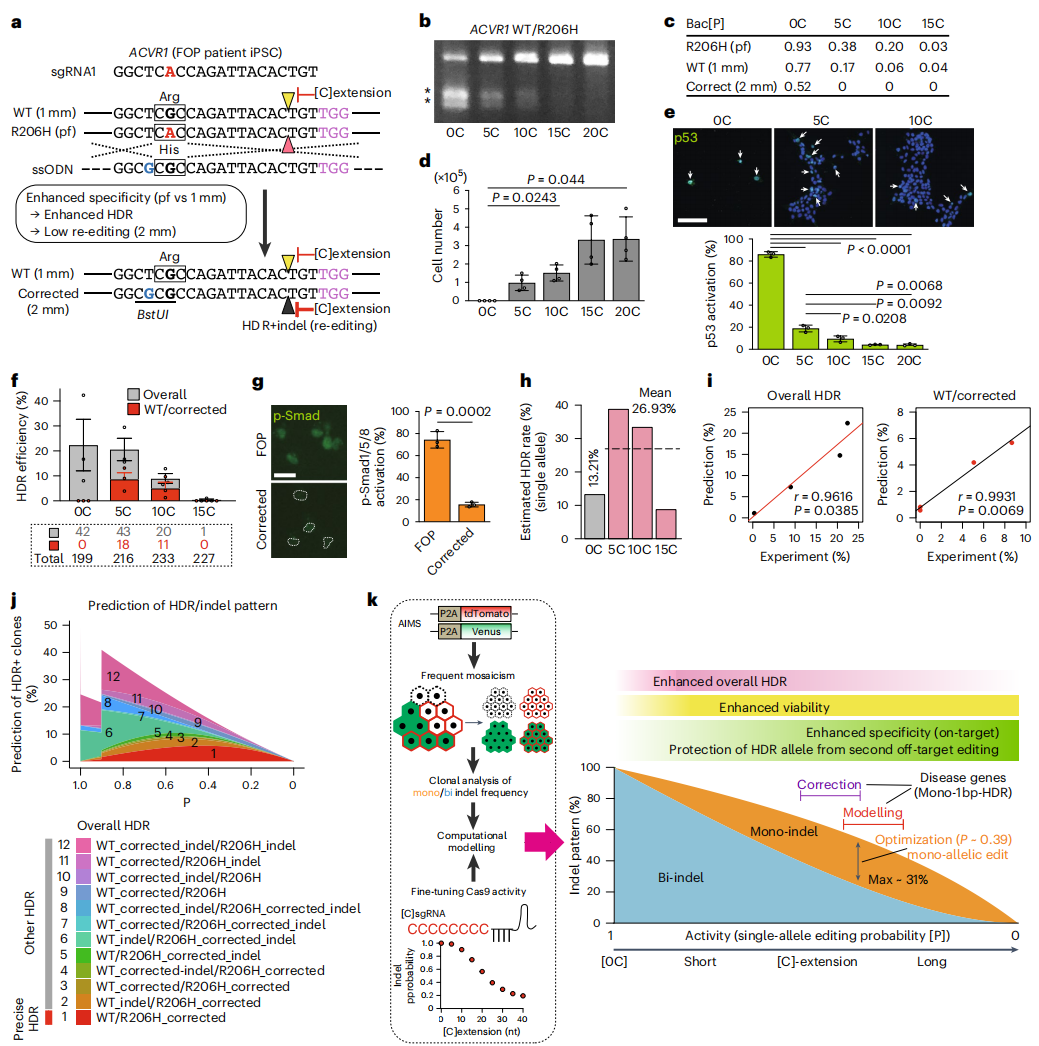
图四 FOP hiPSC的安全性、系统性、精确性基因校正与计算模拟[8]
综上所述,研究者在广泛使用的、基于质粒的基因组编辑方法的基础上,设计了一种易于调控Cas9活性的新策略——[C]sgRNA。在sgRNA上简单添加了胞嘧啶延伸,即可提高基因组编辑的有效性和安全性。[C]延伸降低了有效sgRNA-Cas9复合物的相对浓度,具有广泛的适用性。这种简单的基因组编辑优化方法,将为基因治疗和细胞治疗等临床应用打开新的治疗思路。

GTP研发中心拥有自主知识产权的“人造精子细胞”介导的半克隆技术,可在体外实现“人造精子细胞”的多位点改造并进行功能元件测试,最后将“人造精子细胞”通过卵母细胞注射可以一步法获得基因改造小鼠。基于“人造精子细胞”单倍体打靶高效率的优势,可快速构建定制化小鼠,如基因组安全位点大片段敲入、条件性基因敲除/敲入和人源化基因改造等。多个复杂调控元件可以通过多次体外细胞打靶构建在同一株“人造精子细胞”中,经细胞水平质控后,一步法获得基因改造小鼠。

如有需要欢迎联系我们,GTP研发中心将竭诚为您设计最佳方案,制定专属于您的基因改造小鼠。
网址:http://www.sibcb.ac.cn/gtp/
地址:上海市岳阳路320号
责任编辑:多一百
新媒体运营:suway
参考文献
1. Cong L, Ran FA, Cox D, Lin S, Barretto R, Habib N, Hsu PD, Wu X, Jiang W, Marraffini LA et al: Multiplex genome engineering using CRISPR/Cas systems. Science 2013, 339(6121):819-823.
2. Haapaniemi E, Botla S, Persson J, Schmierer B, Taipale J: CRISPR-Cas9 genome editing induces a p53-mediated DNA damage response. Nat Med 2018, 24(7):927-930.
3. Kosicki M, Tomberg K, Bradley A: Repair of double-strand breaks induced by CRISPR-Cas9 leads to large deletions and complex rearrangements. Nat Biotechnol 2018, 36(8):765-771.
4. Ihry RJ, Worringer KA, Salick MR, Frias E, Ho D, Theriault K, Kommineni S, Chen J, Sondey M, Ye C et al: p53 inhibits CRISPR-Cas9 engineering in human pluripotent stem cells. Nat Med 2018, 24(7):939-946.
5. Harrington LB, Doxzen KW, Ma E, Liu JJ, Knott GJ, Edraki A, Garcia B, Amrani N, Chen JS, Cofsky JC et al: A Broad-Spectrum Inhibitor of CRISPR-Cas9. Cell 2017, 170(6):1224-1233 e1215.
6. Hynes AP, Rousseau GM, Agudelo D, Goulet A, Amigues B, Loehr J, Romero DA, Fremaux C, Horvath P, Doyon Y et al: Widespread anti-CRISPR proteins in virulent bacteriophages inhibit a range of Cas9 proteins. Nat Commun 2018, 9(1):2919.
7. Zou RS, Liu Y, Wu B, Ha T: Cas9 deactivation with photocleavable guide RNAs. Mol Cell 2021, 81(7):1553-1565 e1558.
8. Kawamata M, Suzuki HI, Kimura R, Suzuki A: Optimization of Cas9 activity through the addition of cytosine extensions to single-guide RNAs. Nat Biomed Eng 2023,7:672-691.
9. Mullally G, van Aelst K, Naqvi MM, Diffin FM, Karvelis T, Gasiunas G, Siksnys V, Szczelkun MD: 5' modifications to CRISPR-Cas9 gRNA can change the dynamics and size of R-loops and inhibit DNA cleavage. Nucleic Acids Res 2020, 48(12):6811-6823.



