技术分享:单核苷酸突变识别精度的小巧型基因编辑工具CRISPR-Cas12j-8
RNA引导的CRISPR-Cas系统是一种原核生物的适应性免疫系统,已被广泛用于哺乳动物的基因组编辑[1]。CRISPR-Cas系统可以通过非同源末端连接(NHEJ)修复途径来治疗常染色体显性疾病[2]。可是,Cas蛋白酶的体积过大是限制其临床应用的主要因素之一。Cas12j是2020年新发现的一类体型小巧的CRISPR-CasΦ系统,仅有700-800个氨基酸[3]。该系统仅包含crRNA和Cas12j两个元件,不需要tracrRNA,已被证明在人源细胞系和植物细胞中表现出可观的基因编辑能力[3],为开发应用型的基因编辑工具提供了新的可能。
常染色体显性疾病主要由单核苷酸错义突变引起的,如果错义突变形成了一个新的原间隔序列邻近基序(PAM),那么CRISPR-Cas核酸酶可以通过PAM特异性的方法破坏突变等位基因[2]。另外,还可以将错义突变设计在引导序列内,通过引导特异性的方式进行破坏[4]。这就要求CRISPR-Cas系统具有极高的特异性。然而,目前许多单核苷酸突变仍不能通过引导特异性方法区分于野生型等位基因[5]。因此,开发出一种能够区分单核苷酸突变的高度特异性的应用型基因编辑工具至关重要。
2023年2月,Science Advances期刊报道了一种新的特异性极高的基因编辑工具Cas12j-8,编辑精度可达到区分单核苷酸突变。Cas12j-8蛋白小,特异性识别TTN PAM,编辑范围较广,并对PAM近端第1-13位的单个核苷酸错配高度敏感,脱靶效率低。因此结合单核苷酸多态性(SNP)位点可被用于等位基因特异性的基因组编辑,在单核苷酸突变致病的基因治疗中展现出广泛的应用潜力[6]。
首先,研究者分析了不同Cas12j同源物的CRISPR重复序列同源性[3],发现在这些重复序列的中间含有一个保守的“ATTGC”基序,且3′ 端含有一个“GAC”基序。选择鉴定6个Cas12j同源物(Cas12j-4、5、7、8、9和10)的基因组编辑活性。采用的筛选方法是先前建立的绿色荧光蛋白(GFP)激活表达系统[7],在翻译起始密码子(ATG)和GFP编码序列之间插入一个携带5 bp随机序列的目标DNA,以防止GFP的表达(图一A)。当Cas12j系统实现基因组编辑时,将在目标DNA上发生插入和缺失(indels),导致移码突变,进而促使部分细胞表达GFP(图一A)。每个Cas12j同源物及其相应的24 nt crRNA被克隆并进行密码子优化,其中Cas12j-10的crRNA未被预测到,采用的是其他同源物的crRNA骨架。以FnCas12a为阳性对照,将单个Cas12j同源物及其相应的crRNA转染到报告细胞中(图一B)。3天后,发现FnCas12a对照组和Cas12j-8实验组出现GFP阳性细胞;当Cas12j-10实验组携带Cas12j-9的crRNA骨架时,也能产生GFP阳性细胞;其中Cas12j-8实验组的阳性细胞最多(图一C)。WebLogo图和PAM序列分析显示,Cas12j-8与FnCas12a相似,优先识别TTN PAM(图一D和E)。以上结果表明,Cas12j-8系统具有基因组编辑活性。
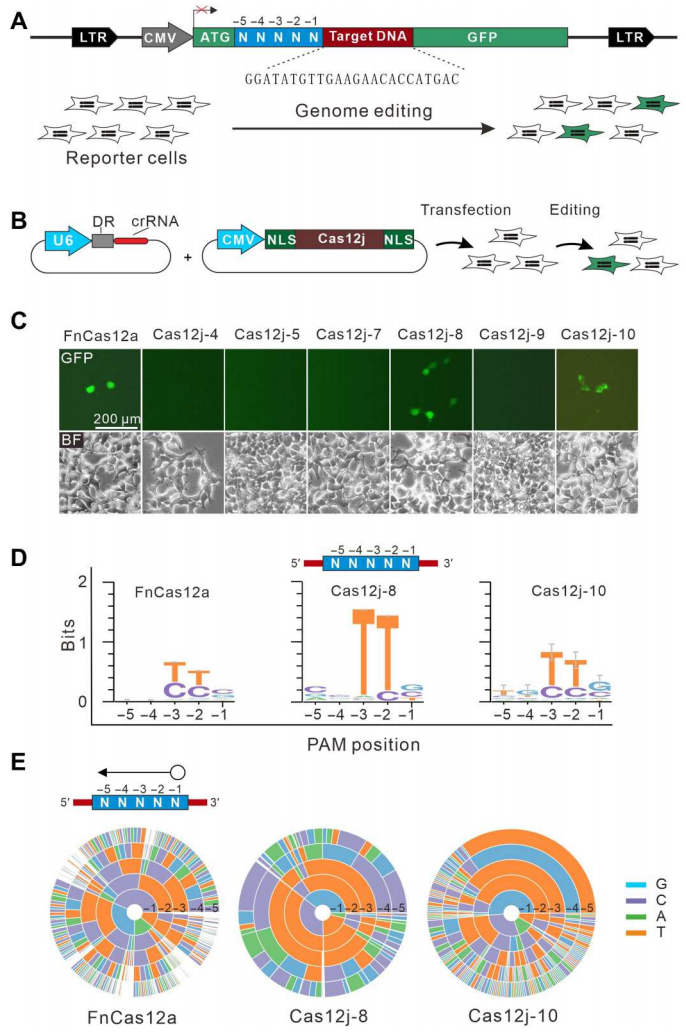
图一 Cas12j的哺乳动物基因组编辑活性测试[6]
接着,研究者测试Cas12j-8系统的基因编辑特异性。当crRNA与靶序列在PAM近端位置1-14位以及第16位发生单一核苷酸错配时,Cas12j-8仅产生极低水平的GFP阳性细胞(图二A)。靶向深度测序显示与GFP激活表达系统结果相一致的极低水平indels(图二A)。体外消化试验显示,纯化的Cas12j-8对PAM近端第1-5、7-8、10-11和13位的单个错配高度敏感,而对其他位置的单个错配敏感性相对较低(图二B和C)。由于体外实验的裂解活性远高于体内活性,因此通过体外裂解实验检测到更高的脱靶效应。研究者进一步采用GUIDE-seq分析全基因组编辑脱靶情况,以FnCas12a系统为对照,针对AAVS1位点设计了3个crRNA,将Cas12/crRNA质粒转染至HEK293T细胞,5天后进行深度测序分析。结果显示FnCas12a和Cas12j-8在所有目的靶标上都发生了切割,并且FnCas12a在位点1出现了两个脱靶效应,而Cas12j-8脱靶为零(图2D)。以上数据表明,Cas12j-8系统的基因组编辑具有高度特异性。
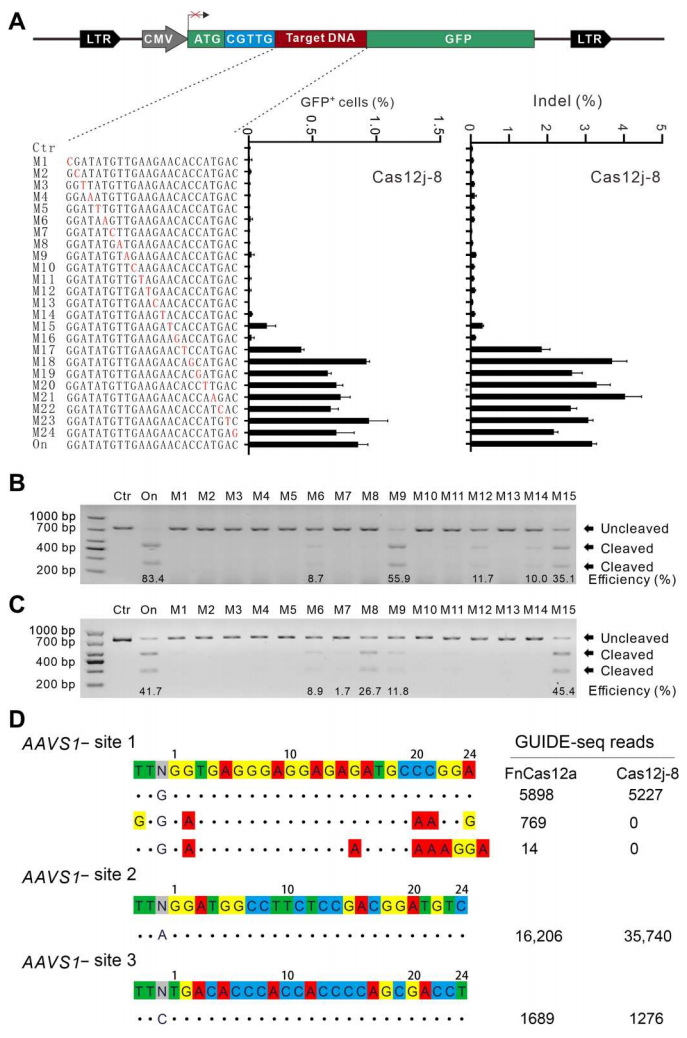
图二 Cas12j-8系统的基因编辑特异性分析[6]
随后,研究者对Cas12j-8系统的基因编辑活性进行评定,采用相同的体系平行比较同样识别TTN PAM的Cas12j-8、FnCas12a和Cas12j-2系统(图三A)。这三种蛋白在相似的基因表达水平下(图三B),都可以在目的靶位点上产生indels,其中Cas12j-8的编辑活性显著高于FnCas12a和Cas12j-2(图三C和D)。Cas12j-8蛋白较小,可以通过单个AAV递送进行基因组编辑,编辑效率也不错(图三C和D)。以上数据表明,Cas12j-8系统在哺乳动物细胞中具有较高的基因组编辑活性。
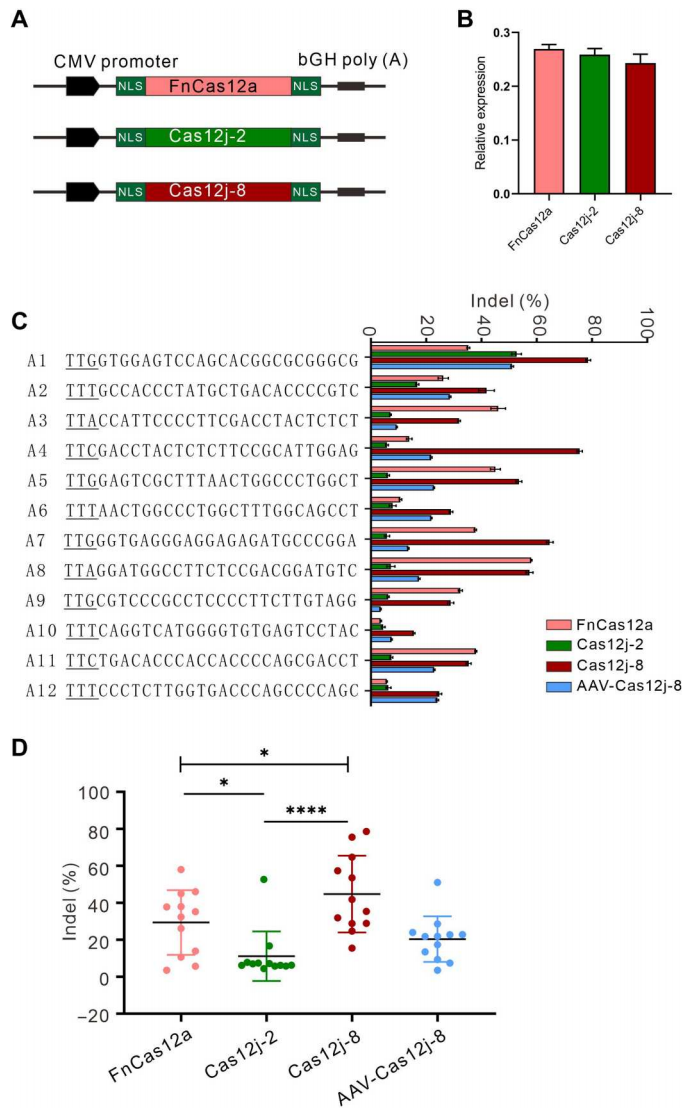
图三 Cas12j-8系统的基因编辑活性分析[6]
为了将Cas12j-8应用于碱基编辑,研究者基于LbCas12a的晶体结构构建了Cas12j-8的SWISS-MODEL,推测Cas12j-8的E567残基与LbCas12a的DNA裂解酶活重要位点E925相对应(图四A和B)。于是,将E567突变为丙氨酸以获得失活的Cas12j-8(dCas12j-8)。经测试,dCas12j-8确实无法编辑内源性位点(图四C)以及体外裂解目标DNA(图四D)。进一步将dCas12j-8与工程腺嘌呤脱氨酶(TadA8e)融合后,得到一个仅有938个氨基酸的碱基编辑器Cas12j-8ABE8e(图四E)。在20个内源性位点上测试Cas12j-8ABE8e的碱基编辑活性,发现仅有4个位点出现A到G的碱基转换,转换率为5.1%-11.3%(图四E)。以上数据表明,Cas12j-8ABE8e可用于碱基编辑,不过碱基编辑效率较低,后续仍需进一步优化。
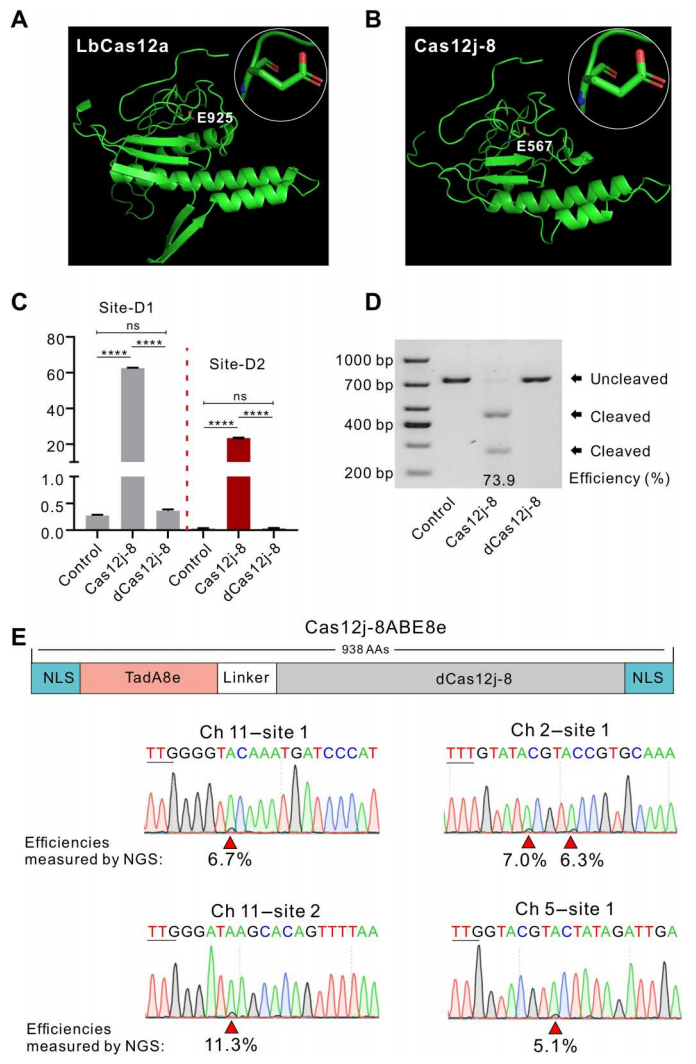
图四 Cas12j-8系统可实现碱基编辑[6]
近期一项研究表明,若一个天然变体顺式位于致病突变的等位基因上,那么这个天然变体可以用来特异性破坏突变等位基因[8]。作为概念证明,研究者使用Cas12j-8系统来特异性地破坏包含单核苷酸多态性(SNP)位点的等位基因。选择同时包含2个SNP位点的靶基因进行测试,以便区分编辑后的等位基因。共设计4个靶位点进行Cas12j-8编辑测试,发现靶等位基因可以被有效地编辑破坏,而非靶等位基因的脱靶效应较低(图五A-D)。预测分析显示Cas12j-8系统可用于等位基因特异性地基因编辑ClinVar数据库[9]中25,931个临床相关变体和dbSNP数据库[10]中485,130,147个SNP位点,可编辑数量大约是Un1Cas12f1的两倍(图五E),具有广泛的临床应用前景。
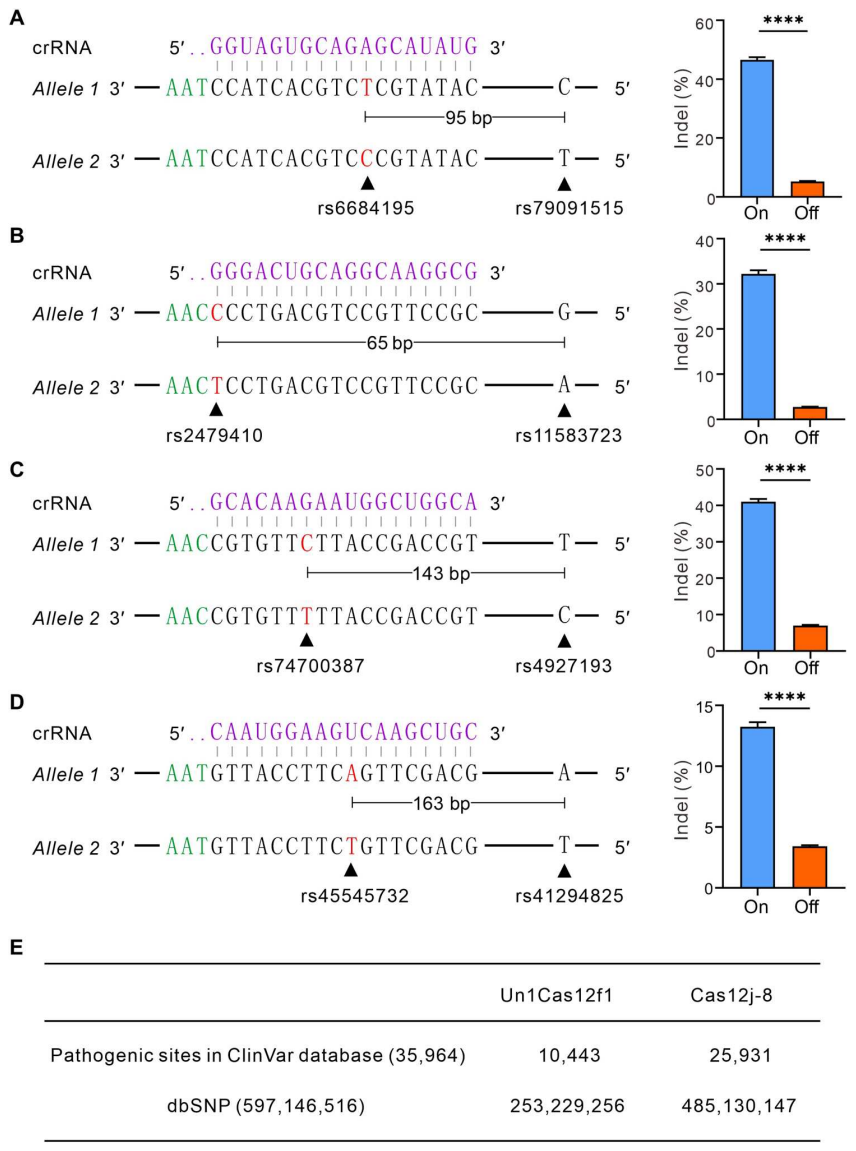
图五 Cas12j-8系统可实现等位基因特异性的基因组编辑[6]
综上所述,Cas12j-8系统具有蛋白小、基因编辑活性高、可区分单个核苷酸突变的多重优势,可作为一种理想的基因组编辑新工具。Cas12j-8的基因编辑活性与AsCas12a和Un1Cas12f1相当,能够实现SNP位点依赖的等位基因特异性破坏。由于Cas12j-8系统识别简单的TTN PAM,编辑范围较大,使其在ClinVar和dbSNP数据库中的可编辑位点数量大约是Un1Cas12f1的两倍,为相关疾病的临床治疗提供了广泛的潜在应用前景。

GTP研发中心拥有自主知识产权的“人造精子细胞”介导的半克隆技术,可在体外实现“人造精子细胞”的多位点改造并进行功能元件测试,最后将“人造精子细胞”通过卵母细胞注射可以一步法获得基因改造小鼠。基于“人造精子细胞”单倍体打靶高效率的优势,可快速构建定制化小鼠,如基因组安全位点大片段敲入、条件性基因敲除/敲入和人源化基因改造等。多个复杂调控元件可以通过多次体外细胞打靶构建在同一株“人造精子细胞”中,经细胞水平质控后,一步法获得基因改造小鼠。

如有需要欢迎联系我们,GTP研发中心将竭诚为您设计最佳方案,制定专属于您的基因改造小鼠。
网址:http://www.sibcb.ac.cn/gtp/
地址:上海市岳阳路320号
责任编辑:多一百
新媒体运营:suway
参考文献
1. Cong L, Ran FA, Cox D, Lin S, Barretto R, Habib N, Hsu PD, Wu X, Jiang W, Marraffini LA et al: Multiplex genome engineering using CRISPR/Cas systems. Science 2013, 339(6121):819-823.
2. Diakatou M, Dubois G, Erkilic N, Sanjurjo-Soriano C, Meunier I, Kalatzis V: Allele-Specific Knockout by CRISPR/Cas to Treat Autosomal Dominant Retinitis Pigmentosa Caused by the G56R Mutation in NR2E3. Int J Mol Sci 2021, 22(5).
3. Pausch P, Al-Shayeb B, Bisom-Rapp E, Tsuchida CA, Li Z, Cress BF, Knott GJ, Jacobsen SE, Banfield JF, Doudna JA: CRISPR-Cas Phi from huge phages is a hypercompact genome editor. Science 2020, 369(6501):333-+.
4. Gao X, Tao Y, Lamas V, Huang M, Yeh WH, Pan B, Hu YJ, Hu JH, Thompson DB, Shu Y et al: Treatment of autosomal dominant hearing loss by in vivo delivery of genome editing agents. Nature 2018, 553(7687):217-221.
5. Christie KA, Courtney DG, DeDionisio LA, Shern CC, De Majumdar S, Mairs LC, Nesbit MA, Moore CBT: Towards personalised allele-specific CRISPR gene editing to treat autosomal dominant disorders. Sci Rep 2017, 7(1):16174.
6. Wang Y, Qi T, Liu J, Yang Y, Wang Z, Wang Y, Wang T, Li M, Li M, Lu D et al: A highly specific CRISPR-Cas12j nuclease enables allele-specific genome editing. Sci Adv 2023, 9(6):eabo6405.
7. Hu Z, Wang S, Zhang C, Gao N, Li M, Wang D, Wang D, Liu D, Liu H, Ong SG et al: A compact Cas9 ortholog from Staphylococcus Auricularis (SauriCas9) expands the DNA targeting scope. Plos Biol 2020, 18(3):e3000686.
8. Christie KA, Robertson LJ, Conway C, Blighe K, DeDionisio LA, Chao-Shern C, Kowalczyk AM, Marshall J, Turnbull D, Nesbit MA et al: Mutation-Independent Allele-Specific Editing by CRISPR-Cas9, a Novel Approach to Treat Autosomal Dominant Disease. Mol Ther 2020, 28(8):1846-1857.
9. Landrum MJ, Lee JM, Benson M, Brown G, Chao C, Chitipiralla S, Gu B, Hart J, Hoffman D, Hoover J et al: ClinVar: public archive of interpretations of clinically relevant variants. Nucleic acids research 2016, 44(D1):D862-868.
10. Sherry ST, Ward MH, Kholodov M, Baker J, Phan L, Smigielski EM, Sirotkin K: dbSNP: the NCBI database of genetic variation. Nucleic acids research 2001, 29(1):308-311.



